
- •Раздел 1. Общие вопросы генетики поведения.
- •1.2. История развития генетики поведения как науки.
- •1.3. Понятие признака в генетике поведения
- •1.4. Методы оценки признаков поведения (поведенческое фенотипирование).
- •1.5. Некоторые принципы генетического анализа поведения.
- •Глава 2. Пути реализации генетической информации на уровне поведения
- •2.1. Генетика морфологических особенностей нервной системы и их связь с изменчивостью признаков поведения.
- •2.2. Связь поведения с некоторыми биохимическими показателями.
- •2.3. Гормональная регуляция изменчивости признаков поведения и эндокринологическая генетика.
- •Глава 3 . Генетика поведения бактерий.
- •3.1. Генетические основы социального поведения бактерий.
- •3.2. Генетика хемотаксиса у бактерий.
- •3.3. Самоидентификация и взаимное узнавание бактерий.
- •Глава 4. Генетика поведения одноклеточных животных
- •4.1. Особенности поведения одноклеточных животных.
- •4.2. Генетика поведения инфузорий
- •4.3. Генетика поведения Dictyostelium discoideum
- •Глава 5. Генетика поведения беспозвоночных животных.
- •5. 1. Генетика поведения круглых червей.
- •5.2. Генетика поведения моллюсков.
- •5. 3. Генетика поведения насекомых
- •5.3.1. Насекомые как объект генетики поведения.
- •5.3.2. Влияние отдельных генов на поведение насекомых
- •5.3.3. Некоторые аспекты генетики поведения общественных насекомых.
- •5.3.4. Генетические основы нейрогуморальной регуляции поведения насекомых.
- •5.3.5. Эволюционные аспекты поведения насекомых.
- •5.3.6. Генетика полового поведения близких видов саранчовых (Acridoidea)
- •Глава 6. Генетика поведения дрозофилы.
- •6.1. История изучения поведенческих мутаций дрозофилы.
- •6.2. Зрительные мутации дрозофилы.
- •6.3. Мутации двигательной системы у дрозофилы.
- •6.4. Температурочувствительные мутации у дрозофилы
- •6.5. Мутации, нарушающие циркадные ритмы у дрозофилы
- •6.6. Мутации, изменяющие половое поведение дрозофилы.
- •6.7. Использование мозаиков для выявления структур, затронутых поведенческими мутациями.
- •6.8. Метод локализации фокуса действия мутации на карте презумптивных органов дрозофилы.
- •6.9. Селекционно-генетический метод в анализе поведения дрозофилы.
- •Глава 7. Генетика поведения птиц.
- •7.1. Птицы как объект генетического анализа поведения.
- •7.2. Средовая модификация некоторых форм врожденного поведения у птиц.
- •7.3. Импринтинг и его роль в постнатальном онтогенезе выводковых птиц.
- •7.4. Гибридологический анализ поведения птиц.
- •7.5. Отдельные гены и признаки поведения птиц.
- •7.6. Эволюционная модификация поведения птиц.
- •Глава 8. Генетика поведения млекопитающих.
- •8.1. Генетика поведения собак.
- •8.2. Генетика поведения грызунов.
- •8.3. Генетика поведения кошек.
- •Типы наследования некоторых признаков и аномалий у кошек
- •8.4. Генетика поведения лошадей и крупного рогатого скота.
- •8.5. Генетика поведения лис.
3.3. Самоидентификация и взаимное узнавание бактерий.
В ходе эволюции у разных организмов развились разнообразные, в том числе весьма сложные и эффективные, механизмы различения «свой»-«чужой». Способность отличать своих от чужих — одно из фундаментальных свойств живого. На этой способности основаны важнейшие биологические процессы и явления, в том числе половое размножение (выбор партнера), видообразование (формирование репродуктивной изоляции), защита от патогенных микробов и вирусов, отторжение чужеродных тканей, колониальность, многоклеточность, социальность, включая разные формы кооперации и даже альтруизма (который, как правило, направлен на «своих», прежде всего на родственников).
В ходе эволюции у разных организмов развились разнообразные, в том числе весьма сложные и эффективные, механизмы различения своего и чужого. У высших организмов (эукариот) эти механизмы изучены значительно лучше, чем у прокариот (бактерий и архей).
Поскольку генетические и биохимические основы различения «своих» и «чужих» у бактерий изучены пока недостаточно, то каждое новое открытие в этой области привлекает к себе пристальное внимание.
Американскими микробиологами был раскрыт механизм взаимной идентификации у бактерии протея (Proteus mirabilis) (Рис.3.5). Этот микроб живет в пищеварительном тракте человека и обычно безвреден, но при неблагоприятном стечении обстоятельств может вызывать инфекционные заболевания.
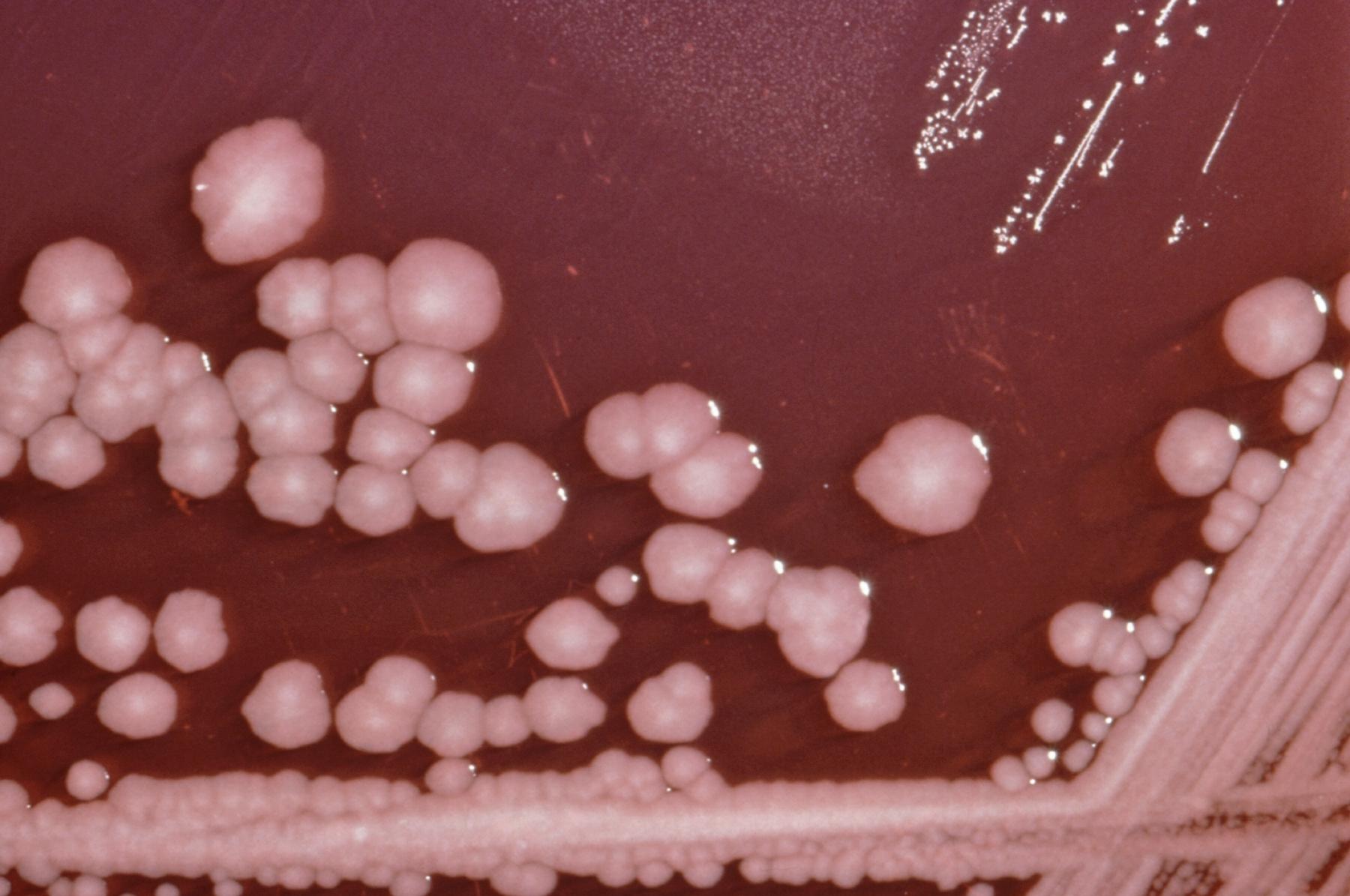
Рис.3.5. Бактерии Proteus mirabilis
Для протея характерен довольно сложный аналог социального поведения, что выражается, в частности, в согласованных передвижениях больших групп бактериальных клеток.
В геноме этой бактерии выявлен участок, отвечающий за самоидентификацию и взаимное узнавание микробов. Мутации в этом участке могут приводить к мгновенному разделению популяции микробов на группы, относящиеся друг к другу как к «чужакам». Представители таких групп «отказываются» образовывать смешанные скопления.
Когда две колонии или «стаи» (swarms) Proteus mirabilis встречаются на поверхности питательной среды, происходит одно из двух: либо колонии сливаются, либо они сохраняют самостоятельность. В последнем случае между ними образуется хорошо видимая граница. Выбор одного из двух вариантов зависит от степени родства. Если обе стаи принадлежат к одному и тому же штамму (клону, генетической линии), они, скорее всего, сольются; если к разным — между ними, скорее всего, возникнет граница. Данная особенность используется в медицинской практике для идентификации штаммов микроба.
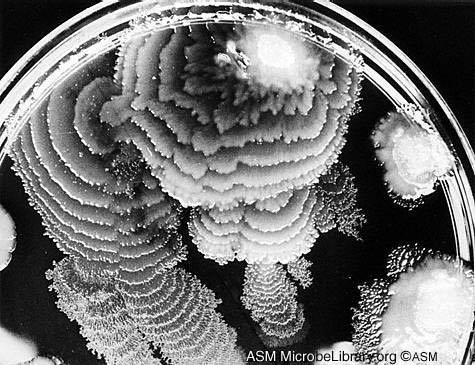
Рис.3.6. Колонии Proteus mirabilis
Было установлено, что многие штаммы Proteus mirabilis выделяют токсичные белки — протицины, предназначенные для борьбы с конкурирующими штаммами. Каждый штамм выделяет свой протицин, к которому он устойчив, и к тому же обладает устойчивостью к некоторым (но не всем) чужим протицинам. Однако образование границ между неродственными «стаями» определяется не протицинами, а чем-то другим. Дело в том, что некоторые штаммы не выделяют протицинов, но при этом всё равно образуют границы, в том числе и с другими штаммами, лишенными протицинов.
Чтобы выяснить причины этого явления, исследователи «заставили» бактерий быстро мутировать, активизировав перемещение мобильных генетических элементов (транспозонов). Из одного «предкового» штамма (BB2000) было получено 3600 мутантных штаммов-потомков. Среди них обнаружился один, который «отказался» смешиваться с другими мутантами и с предковым штаммом BB2000. Его назвали Ids (от слов «identification of self» — «самоидентификация»). Очевидно, в штамме Ids произошла мутация, вследствие которой этот штамм изменил свою самоидентификацию и начать воспринимать родительский штамм BB2000 как «чужой».
Оказалось, что у штамма Ids транспозон встроился в участок генома, содержащий шесть генов, функции которых до сих пор не были известны. Ученые обозначили этот участок idsABCDEF. По-видимому, транспозон нарушил нормальную работу этих генов, что и привело к «отчуждению» бактерий-мутантов от предков и родственников.
Чтобы проверить это предположение, у бактерий из предкового штамма BB2000 удалили все шесть генов. Получились мутанты, которые считали штамм Ids «своим», а предков — «чужими». Стало ясно, что данные шесть генов действительно играют ключевую роль в самоидентификации данных бактерий и в различении своих и чужих. После этого началась кропотливая генно-инженерная работа с целью выяснения функций каждого из шести генов в отдельности.
Выяснилось, что удаление гена idsA само по себе не ведет к изменению самоидентификации бактерий. Этот ген, по-видимому, играет какую-то вспомогательную роль. Однако удаление или поломка хотя бы одного из генов idsB, idsC, idsD, idsE, idsF приводит к тому, что бактерии начинают считать предковый штамм «чужим». При этом бактерии, у которых удален ген B, C, D или E, считают «своими» бактерий, у которых удалены все шесть генов. Однако бактерии с удаленным геном F считают «чужими» всех, кроме самих себя.
Исследователи обнаружили, что похожие генетические системы идентификации есть и у других штаммов Proteus mirabilis, а также у других видов бактерий. Была проведена дополнительная серия экспериментов, в которой участок idsABCDEF пересаживался (целиком или по частям) другому, неродственному штамму Proteus mirabilis (штамм HI4320) вдобавок к его собственной генетической идентификационной системе. Бактерии из штамма HI4320, которым участок idsABCDEF от штамма BB2000 пересадили целиком, начинают считать свой родной штамм HI4320 «чужим». Однако если в пересаженном участке поврежден ген D или E, отчуждения не происходит: бактерии продолжают считать штамм HI4320 «своим».
Эти и другие, более сложные, эксперименты позволили ученым установить, что самую важную роль в различении «своих» и «чужих» играют гены D и E. Именно они определяют «индивидуальность» бактерии. В этих генах обнаружились вариабельные области, то есть участки, сильно различающиеся у разных штаммов. Гены D и E образуют уникальную генетическую «идентификационную карточку», по которой штаммы бактерий различают друг друга.
Гены B, C и F, по-видимому, нужны для того, чтобы эта «идентификационная карточка» могла быть должным образом представлена и опознана. У разных штаммов эти гены различаются слабо, и если взять любой из этих генов у одного штамма и пересадить другому, самоидентификация бактерий не изменится.
Судя по ряду косвенных признаков, система идентификации у Proteus mirabilis не ограничена участком idsABCDEF. Скорее всего, существуют и другие, пока еще не выявленные, гены, участвующие в различении «своих» и «чужих». Вся система в целом может оказаться весьма сложной.
Существуют и другие ситуации, в которых бактерии могут решать дилемму «свой»-«чужой». Например, ситуация симбиоза.
Симбиоз с различными бактериями чрезвычайно широко распространен как у животных, так и у растений. Очень часто многоклеточный организм не получает симбионтов автоматически от своих родителей, а должен сам каким-то образом найти их в окружающей среде и «получить» их в начале своей жизни. Учитывая огромное разнообразие микроорганизмов во всех природных местообитаниях, понятно, что в такой ситуации первостепенное значение приобретает точность «взаимного узнавания» симбионта и хозяина.
Но механизмы этого узнавания, безусловно, существуют.
Есть паразитические бактерии, у которых очень близкие формы паразитируют на совершенно разных хозяевах. Например, одна из разновидностей бактерии Salmonella enterica заражает только людей, а другая имеет широкий круг хозяев — от приматов до грызунов. При этом геномы обеих разновидностей чрезвычайно похожи друг на друга. Пока не удалось обнаружить в их геномах то ключевое различие, от которого зависит выбор хозяина.
В случае симбиоза оба участника заинтересованы в точном узнавании партнера. Ученым удалось расшифровать механизм взаимного узнавания в симбиотической ассоциации растений с азотфиксирующими бактериями. В этом случае ключевую роль играют выделяемые бактериями сигнальные вещества, которые определенным образом влияют на работу генов в клетках корня растения-хозяина.
Частично расшифрован и механизм выбора партнера у другой пары симбионтов — тихоокеанского кальмара Euprymna scolopes и светящейся бактерии Vibrio fischeri.

Рис.3.7. Кальмар Euprymna scolopes светится благодаря симбиотическим бактериям Vibrio fischeri, живущим в особом «светящемся органе».
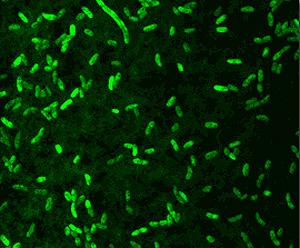
Рис.3.8. Светящиеся бактерии Vibrio fischeri
Известно, что одни разновидности светящейся бактерии Vibrio fischeri живут в симбиозе с рыбами, другие — с кальмарами. Ученые выяснили, что способность заселять светящиеся органы кальмаров определяется одним-единственным бактериальным геном. Пересадка этого гена от «кальмарной» разновидности микроба его «рыбной» разновидности приводит к тому, что последняя приобретает способность жить в симбиозе с кальмарами.
Геномы «кальмарной» и «рыбной» разновидности Vibrio fischeri были полностью изучены и оказались весьма сходными (90% сходства по набору генов и 98,8% — по аминокислотным последовательностям белков).
Исследователи обнаружили, что один из тех генов, которые активно работают у Vibrio fischeri в период заселения светящихся органов молодых кальмаров, в геноме «рыбного» штамма отсутствует. Дальнейшие исследования показали, что этот ген (его условное обозначение — rscS) действительно играет ключевую роль в содружестве бактерий с кальмарами.
Ген rscS кодирует белок-рецептор, который располагается на клеточной мембране, реагирует на какие-то внешние сигналы и передает их внутрь клетки, активизируя другой регуляторный белок — транскрипционный фактор SypG. В свою очередь, SypG стимулирует активность группы генов, которые кодируют белки, необходимые для синтеза особых полисахаридов, которые выводятся из бактериальной клетки и располагаются на ее наружной поверхности. От этих полисахаридов зависит способность бактерий размножаться в слизи, выделяемой светящимся органом кальмара.
Ген rscS встречается в двух аллельных вариантах (rscSA и rscSB), причем только аллель rscSA позволяет бактерии вступить в симбиоз с кальмаром. Именно этот вариант гена присутствует у всех «кальмарных» штаммов и у того «рыбного» штамма, который сумел наладить отношения с кальмаром. У всех остальных «рыбных» штаммов либо присутствует второй вариант гена (rscSB), либо ген вовсе отсутствует (как у той «рыбной» линии, чей геном был полностью прочтен авторами).
Чтобы убедиться в том, что наличие аллеля rscSA у V. fischeri является необходимым и достаточным условием симбиоза с кальмарами, ученые провели ряд генно-инженерных экспериментов. Они выводили из строя этот ген у «кальмарных» штаммов — и бактерии теряли способность жить в содружестве с кальмарами. Они внедряли ген в «рыбные» штаммы — и они приобретали эту способность. Таким образом, предположение полностью подтвердилось.
На основе сравнения нуклеотидных последовательностей трех других генов исследователи построили эволюционное древо изученных штаммов микроба. Судя по этому древу, изначально V. fischeri не имел гена rscS и жил в симбиозе только с рыбами. Затем один из штаммов микроба приобрел этот ген. Все штаммы, имеющие ген rscS, образуют на древе монофилетическую (происходящую от одного предка) группу. Это означает, что ген был приобретен только один раз. Сначала он существовал только в одном варианте — rscSA. Некоторые из потомков того микроба, который впервые обзавелся новым геном, покинули своих прежних хозяев — рыб — и перешли к симбиозу с кальмарами. Впоследствии кое-кто из них вернулся к симбиозу с рыбами, причем это возвращение сопровождалось изменениями в гене rscS. Так появился второй вариант этого гена — rscSB, который, возможно, каким-то образом улучшает «взаимопонимание» между микробами и рыбами.
Это первый известный случай, когда главным «действующим лицом» во взаимном узнавании симбионтов оказался регуляторный ген.
Ученые считают, что необходимо дальнейшее изучение генетики процессов самоидентификации и взаимного узнавания бактерий для их дальнейшего практического использования.
