
- •Практическое занятие № 3. Механизм репликации днк
- •Типы репликации кольцевых днк
- •I. Репликация днк e. Coli
- •Сравнительная характеристика днк-полимераз e. Coli
- •Элонгация
- •Терминация
- •II. Репликация днк эукариот
- •Репликация днк: праймер
- •Точки начала репликации
- •Точки начала репликации Saccharomyces cerevisiae
- •Сборка предрепликационного комплекса
- •Механизм синтеза эукариотической днк
I. Репликация днк e. Coli
Казалось, что более 90% ДНК-полимеразной активности обнаруживаемой в экстрактах клеток E. coli могло быть приписано ДНК-полимеразе I. Однако, вскоре после выделения этого фермента в 1955 году стало очевидным, что ДНК-полимераза I не подходит на роль фермента способного осуществить репликацию крупной хромосомы E. coli. Во-первых, скорость, с которой ДНК-полимераза I добавляет нуклеотиды к 3/-концу растущей цепи ДНК (600 нуклеотидов/мин) является слишком малой, чтобы объяснить скорость движения репликативной вилки при удвоении ДНК в бактериальной клетке. Во-вторых, ДНК-полимераза I обладает относительно низкой процессивностью. В-третьих, результаты генетических экспериментов показали, что в процессе репликации участвует множество генов (другими словами множество дополнительных белков) так, что ДНК-полимераза I не может сама по себе обеспечить удвоение ДНК. В-четвертых, что наиболее важно, в 1969 году John Cairns получил необычный штамм E. coli с измененным геном ДНК-полимеразы I, который экспрессировал каталитически неактивный фермент. Несмотря на то, что этот штамм оказался чрезвычайно чувствительным к действию химических агентов повреждающих ДНК, тем не менее, он был жизнеспособным.
Поиски других ДНК-полимераз в клетках E. coli привели к открытию в начале 70-х годов ХХ столетия двух других бактериальных ферментов – ДНК-полимеразы II и ДНК-полимеразы III. Изучение свойств указанных ферментов позволило установить, что ДНК-полимераза II участвует в репарации ДНК определенного типа, в то время как ДНК-полимераза III оказалась основным ферментом репликации ДНК E. coli. Отличительные свойства всех трех ДНК-полимераз приведены в табл. А. ДНК-полимеразы IV и V, идентифицированные в 1999 году принимают участие в специфических процессах репарации ДНК.
Таблица А
Сравнительная характеристика днк-полимераз e. Coli
|
ДНК-полимераза |
||
I |
II |
III |
|
Структурный ген* |
polA |
polB |
polC (dnaE) |
Число различных видов субъединиц |
1 |
7 |
≥10 |
Молекулярная масса |
103.000 |
88.000† |
791.500 |
3/5/-экзонуклеазная корректирующая активность |
+ |
+ |
+ |
5/3/-экзонуклеазная активность |
+ |
– |
– |
Скорость полимеризации (нуклеотидов/сек) |
16-20 |
40 |
250-1000 |
Эффективность (процессивность) (количество добавленных нуклеотидов до отсоединения полимеразы) |
3-200 |
1.500 |
≥500.000 |
* для ферментов включающих более одной субъединицы, приведенные в таблице обозначения генов относятся только к полимеризующей активности. Обратите внимание на то, что обозначение dnaE является несколько устаревшим обозначением принятого в настоящее время символа polC.
† молекулярная масса указана только для полимеризующей субъединицы. ДНК-полимераза II имеет несколько общих с ДНК-полимеразой III субъединиц, включая: , , , /, и -субъединицы.
Таким образом, ДНК-полимераза I не является основополагающим ферментом репликации, вместо этого он выполняет массу «подчищающих» функций, принимая участие в процессах репликации, рекомбинации и репарации. Более того, специфические функции данного фермента расширяются благодаря его специфической 5/3/-экзонуклеазной активности. Данная активность в отличие от корректирующей 3/5/-экзонуклеазной активности связана с отдельным структурным доменом, который может быть отщеплен от интактного фермента посредством мягкого протеолиза. После отщепления 5/3/-экзонуклеазного домена образующийся так называемый большой фрагмент ДНК-полимеразы I или фрагмент Кленова (мол. масса 68.000 Да) сохраняет полимеразную и корректирующую 3/5/-экзонуклеазную активности. Специфическая 5/3/-экзонуклеазная активность интактной ДНК-полимеразы I ответственна за замещение небольших сегментов ДНК (РНК) спаренных с матричной цепью в процессе так называемой nick-трансляции. Характерно, что другие ДНК-полимеразы утратили 5/3/-экзонуклеазную активность.
Основной фермент репликации ДНК у E. coli – ДНК-полимераза III является гораздо более сложно устроенным, чем ДНК-полимераза I комплексом, включающим 10 разных видов (типов) субъединиц (табл. В) (рис. 1). Полимеризующая и корректирующая активности этого фермента связаны с его - и -субъединицами, соответственно. Субъединица взаимодействуя с - и -субъединицами образует кор-фермент, который способен катализировать реакцию полимеризации, но с ограниченной эффективностью. Два кор-фермента могут быть соединены с другим набором субъединиц: -комплексом, состоящим из семи субъединиц шести различных типов – 2/, который способствует значительному увеличению процессивности полимеразы. (Мерой процессивности ДНК-полимеразы является длина фрагмента вновь синтезированной макромолекулы, которую фермент способен образовать в одном цикле, не диссоциируя от матрицы). За процесс сборки холофермента и его димеризацию отвечают -субъединицы. Субъединицы и кодируются одним и тем же геном, однако -субъединица является укороченным вариантом субъединицы . Таким образом -субъединица включает домен идентичный и дополнительный сегмент ответственный за связывание с кор-ферментом. Полный ансамбль из 13 белковых субъединиц девяти (9) различных видов обозначают ДНК-полимеразой III*.
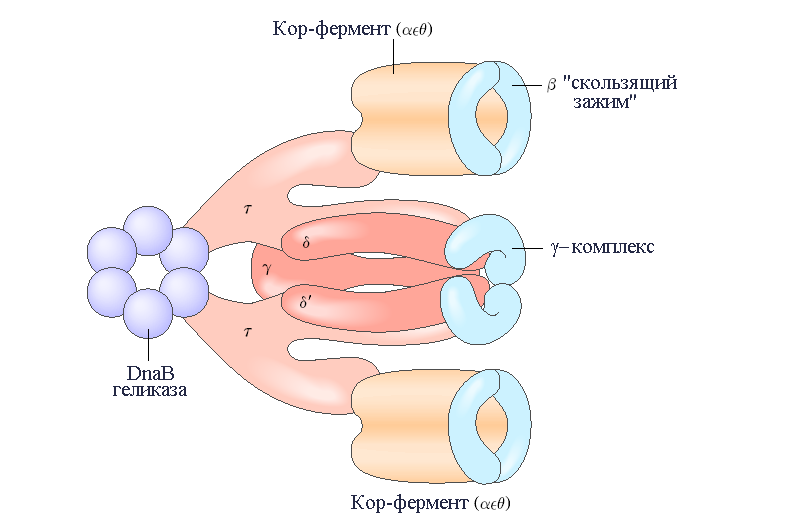
Рис. 1 |
Строение ДНК-полимеразы III E. coli. Показано, что холофермент ДНК-полимеразы III включает два домена, представленных кор-ферментами состоящими из субъединиц , и . Два кор-фермента связаны в димер с помощью -комплекса построенного из семи субъединиц шести различных типов – 2/ (субъединицы и не показаны). Субъединицы и кодируются одним и тем же геном, однако -субъединица является укороченным вариантом субъединицы . Таким образом -субъединица включает домен идентичный и дополнительный сегмент ответственный за связывание с кор-ферментом. С каждым кор-субансамблем взаимодействует так называемый -«скользящий зажим» состоящий из двух -субъединиц. В целом холофермент ДНК-полимеразы III связан с dnaB-геликазой с помощью субъединицы . PDB ID 2POL |
ДНК-полимераза III* способна полимеризовать ДНК, но с намного более низкой процессивностью, чем необходимо для удвоения всей хромосомы E. coli.
Таблица В
Характеристика субъединиц ДНК-полимеразы III E. coli
Субъединица |
Число субъединиц в холоферменте |
Молекулярная масса субъединицы |
Ген |
Функции субъединиц |
|
|
2 |
129.900 |
polC (dnaE) |
полимеразная активность |
кор-фермент |
|
2 |
27.500 |
dnaQ (mutD) |
3/→5/-экзонуклеазная корректирующая активность |
|
|
2 |
8.600 |
holE |
|
|
|
2 |
71.100 |
dnaX |
стабилизация связывания с матрицей, димеризация кор-фермента |
-комплекс, который обеспечивает взаимодействие -субъединиц вокруг отстающей цепи в каждом фрагменте Оказаки |
|
1 |
47.500 |
dnaX* |
формирование «скользящего зажима» |
|
|
1 |
38.700 |
holA |
открывает «скользящий зажим» |
|
/ |
1 |
36.900 |
holB |
формирование «скользящего зажима» |
|
|
1 |
16.600 |
holC |
взаимодействие с SSB-белками |
|
|
1 |
15.200 |
holD |
взаимодействие с и |
|
|
4 |
40.600 |
dnaN |
«скользящий зажим» ДНК для оптимальной процессивности |
|
* -субъединица кодируется частью гена субъединицы так, что 66% N-концевой последовательности совпадает с полной аминокислотной последовательностью . -субъединица образуется путем сдвига рамки считывания, что приводит к ранней терминации трансляции.
Увеличение процессивности фермента достигается путем присоединения 4-х -субъединиц на каждый холоэнзим ДНК-полимеразы III. -субъединицы взаимодействуют попарно с образованием кольцеобразной структуры, которая обхватывает ДНК и действует на подобие «хомута». Каждый димер взаимодействует с субансамблем кор-фермента ДНК-полимеразы III* и «скользит» вдоль ДНК в ходе репликации. «Скользящий зажим» препятствует отделению полимеразы от молекулы ДНК, существенно увеличивая тем самым процессивность вплоть до 500.000 добавленных нуклеотидов.
В репликации ДНК принимает участие множество ферментов и белковых факторов
В репликации ДНК E. coli принимает участие не только ДНК-полимераза III. Этот сложный процесс зависит также от действия 20 или более вспомогательных ферментов и белковых факторов, каждый из которых выполняет свои специфические функции. Сложный многофункциональный комплекс, обеспечивающий удвоение бактериальной ДНК получил название ДНК-репликазной системы или реплисомы. Вполне понятно, что необходимая сложность ферментативного устройства реплисомы зависит как от структуры самой ДНК, так и оказывается под давлением требований точного воспроизведения структуры ДНК при ее удвоении. В этом разделе рассматриваются основные группы ферментов репликации в свете тех проблем, которые они призваны разрешать.
Доступность материнских цепей исходной ДНК с целью того, чтобы они могли служить матрицами для образования дочерних цепей, достигается в результате их предварительного разделения. Этот процесс зависит от проявления каталитической активности геликаз – ферментов, которые, двигаясь вдоль молекулы ДНК, разделяют ее цепи, используя энергию гидролиза АТР. При этом разделение цепей неизбежно создает специфические топологические проблемы, связанные со спиральной структурой ДНК. Эти проблемы относительно легко разрешаются благодаря участию ДНК-топоизомераз (см. лекцию 3). Разделенные цепи материнской ДНК стабилизируются специфическими ДНК-связывающими белками или SSB-белками. Как было указано выше, для того чтобы ДНК-полимераза могла обеспечить репликацию ДНК, на матричных цепях должны быть синтезированы комплементарные им праймеры – как правило, короткие участки РНК, образование которых зависит от действия праймазы. В конечном итоге РНК-праймеры удаляются путем их замещения участками ДНК. У E. coli этот процесс зависит от участия РНК-азы Н и ДНК-полимеразы I. После удаления праймера и заполнения пробела фрагментом ДНК во вновь синтезированной дочерней цепи остается брешь, суть которой заключается в отсутствии канонической фосфодиэфирной связи. Подобные бреши сшиваются ДНК-лигазами.
Репликация хромосомы E. coli протекает в несколько стадий
Процесс синтеза молекулы ДНК подразделяют на несколько стадий: инициацию, элонгацию и терминацию в зависимости от характера протекающих реакций и ферментов, принимающих участие в их катализе. Следует отметить, что пониманию основополагающих принципов синтеза основных биологических информационных полимеров – ДНК, РНК и белков способствует такое же подразделение этих процессов на указанные выше стадии. Описываемые ниже события являются результатом исследований, выполненных in vitro c использованием выделенных и очищенных ферментов E. coli хотя принципы лежащие в основе этих реакций высококонсервативны и характерны для всех репликативных систем.
Инициация
Точка начала репликации ДНК E. coli, обозначаемая oriC, включает 245 пар оснований: локус ori включает элементы ДНК-последовательностей, которые являются высококонсервативными для всех точек начала репликации у бактерий. Общее расположение консервативных последовательностей приведено на рис. 2.
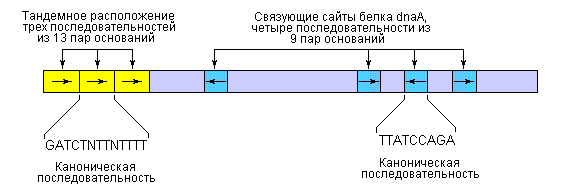
Рис. 2 |
Порядок расположения консервативных последовательностей в точке начала репликации хромосомы E. coli – oriC. Несмотря на то, что повторяющиеся последовательности не совсем идентичны, некоторые нуклеотиды проявляют высокую степень консервативности по отношению к определенным положениям, формируя общую последовательность. В тех положениях, где отсутствует полное совпадение, символ N означает присутствие любого из четырех нуклеотидов. Стрелками показаны ориентации нуклеотидных последовательностей. |
Ключевые последовательности в области точки начала репликации представлены двумя сериями повторов: тремя повторами из 13 пар оснований и четырьмя повторами из 9 пар оснований. Как следует из табл. С в фазе инициации репликации принимают участие, по меньшей мере, 9 различных ферментов и белковых факторов. Эти ферменты и белки раскрывают спираль ДНК в точке начала репликации и формируют предзатравочный комплекс. Ключевым компонентом инициации является белок dnaA.
Таблица С
Белки, необходимые для инициации репликации хромосомы E. coli в локусе oriC
Белок |
Молекулярная масса |
Число субъединиц |
Функция |
Белок dnaA |
52.000 |
1 |
Узнает специфические последовательности oriC. Открывает дуплекс ДНК в локусе ori. |
Белок dnaB (геликаза) |
300.000 |
6* |
Разделяет цепи ДНК |
Белок dnaC |
29.000 |
1 |
Участвует в связывании dnaB в точке начала репликации |
HU |
19.000 |
2 |
Гистоноподобный белок; ДНК-связывающий белок; стимулирует инициацию репликации |
Белок dnaG (праймаза) |
60.000 |
1 |
Синтезирует РНК-праймеры |
SSB-белок |
75.600 |
4* |
Связывается с одноцепочечным участком ДНК |
РНК-полимераза |
454.000 |
5 |
Усиливает активность dnaA |
ДНК-гираза (ДНК-топоизомераза II) |
400.000 |
4 |
Устраняет топологические напряжения, возникающие в ДНК в результате разделения ее цепей |
Dam-метилаза |
32.000 |
1 |
Метилирует последовательности (5/)GATC в точке начала репликации |
* в указанных случаях субъединицы идентичны
Белковый комплекс из 4-5 молекул инициаторного белка dnaA взаимодействует с каждым из четырех повторов из 9 пар оснований в точке начала репликации и обеспечивает плавление ДНК в области трех повторов из 13 пар оснований, которая обогащена А-Т парами. Этот процесс требует участия АТР и бактериального гистоноподобного белка HU. После этого белок dnaC обеспечивает взаимодействие белков dnaВ с денатурированной областью ДНК. Белки dnaВ в виде кольцеобразных гексамеров связываются с каждой цепью ДНК и действуют как геликазы расплетая двойную спираль и формируя две потенциальные репликативные вилки. Если при этом в условиях in vitro в систему репликации добавить SSB-белки и ДНК-топоизомеразу II (ДНК-гиразу) E. coli dnaВ-геликазы с большой скоростью начнут разделять тысячи пар оснований двигаясь в противоположных направлениях от точки начала репликации. Множество молекул SSB-белков кооперативно связываются с одноцепочечной ДНК стабилизируя одиночные цепи и препятствуя их ренатурации в то время как гираза устраняет топологическое напряжение индуцируемое dnaВ-геликазой. После добавления в систему репликации E. coli in vitro дополнительных ферментов dnaВ-зависимый процесс разделения цепей становится сопряженным с собственно удвоением ДНК.
Фаза инициации репликации ДНК, как известно, является единственной стадией всего процесса, которая регулируется, причем регулируется, так что удвоение ДНК происходит только один раз в каждом клеточном цикле. В целом механизм регуляции репликации все еще мало понятен, однако результаты ряда генетических и биохимических исследований проливают некоторый свет на основы такой регуляции.
В частности установлено, что на выбор времени начала репликации влияет метилирование ДНК и ее взаимодействие с бактериальной плазматической мембраной. Участок ДНК, в составе локуса oriC, метилируется специфической Dam-метилазой (DNA adenine methylation), которая вводит метильную группу по N6-положению аденина в составе короткого палиндрома (5/)GATC. Следует отметить, что локус oriC E. coli чрезвычайно обогащен последовательностями GATC – в составе 245 пар оснований их насчитывается 11, в то время как средняя встречаемость GATC в хромосоме кишечной палочки не превышает 1 на 256 пар оснований.
В норме GATC-палиндромы в локусе oriC полностью метилированы. Сразу после удвоения ДНК в точке начала репликации данные участки становятся полуметилированными: родительские цепи остаются метилированными по последовательностям GATC, но вновь синтезированныее дочерние цепи ДНК имеют неметилированные сайты в последовательности GATC. Полуметилированное состояние локуса oriC становится сигналом для его взаимодействия с плазматической мембраной бактериальной клетки и соответственно изолирования этой точки начала репликации от повторной ее активации (правда механизм такого взаимодействия пока не известен). Спустя определенное время последовательность oriC высвобождается из взаимодействия с плазматической мембраной и снова полностью метилируется с участием Dam-метилазы, получая, тем самым, возможность повторно связывать белки dnaA, а, следовательно, активироваться для следующего цикла репликации. В регуляции инициации репликации также определенную роль играет процесс медленного гидролиза АТР под действием белка dnaA, который включается в циклическое превращение его активной (с АТР) и неактивной форм (без АТР) происходящее в интервале времени от 20 до 40 мин.
