
- •3. Нуклеосома как единица структурной организации хроматина. Октамер гистонов в составе нуклеосомы. Линкер и линкерный гистон.
- •4. Полуконсервативный механизм репликации днк. Опыт Мезельсона и Сталя (1958).
- •5. Свойство анипаралленльности цепей в молекуле днк. Биологический смысл. Правило Чаргаффа.
- •6. Конформационные формы молекулы днк. Денатурация и ренатурация днк. Температура отжига (плавления) днк.
- •Вопрос 7
- •Вопрос 8
- •Вопрос 9
- •11. Транскрипция у прокариот. Основные этапы транскрипции: инициация, элонгация, терминация. Регуляция транскрипции (в вопросе 10).
- •12. Транскрипция у эукариот. Основные этапы транскрипции: инициация, элонгация, терминация. Регуляция транскрипции (в вопросе 10).
- •16. Рекомбинантная днк. Инструментарий для создания рекомбинантных молекул днк.
- •17. Системы рестрикции-модификации (r-m системы), их биологическая роль.
- •19. Клонирование днк in vitro.
- •21. Плазмиды. Плазмидные векторы для клонирования in vivo. Методы трансформации плазмидной днк. Скрининг (отбор) трансформированных бактериальных клеток.
- •22. Получение библиотеки геномной днк с помощью плазмидных векторов.
- •24. Плазмидный вектор для экспрессии гена. Экспрессионные векторы.
- •30. Метод гель-электрофореза. Назначение метода и принцип его действия. Разделение и анализ фрагментов днк. Маркеры молекулярного веса днк. Красители, применяемые при проведении гель-электрофореза.
- •31. Пцр и ее применение
- •Подготовки исследуемой пробы материала, которая в большинстве случаев сводится к выделению днк или рнк;
- •Собственно полимеразной цепной реакции;
- •Детекции продукта пцр (амплифицированной нуклеиновой кислоты).
24. Плазмидный вектор для экспрессии гена. Экспрессионные векторы.
Экспрессионный вектор, транскрипционный вектор (expression vector, transcription vector) [лат. expressio — выражение; лат. vector — везущий, несущий] — рекомбинантный вектор, содержащий все регуляторные элементы, обеспечивающие экспрессию (транскрипцию) чужеродных фрагментов ДНК, которые в него встраиваются. Э.в. может также использоваться для клонирования ДНК.
В настоящее время плазмидные векторы являются одним из возможных средств доставки трансгенов и экспрессии белков. Традиционный плазмидный вектор для экспрессии трансгенов в клетках млекопитающих (например, вектор pcDNA3.1 компании Invitrogen) содержит такие функциональные элементы, как промотор ранних генов цитомегаловируса человека (CMV), участок клонирования, сигнал полиаденилирования мРНК гена гормона роста быка, ген устойчивости к антибиотику G418, ген β-лактамазы, обеспецивающий устойчивость к ампициллину, участок инициации репликации Col E1 [Invitrogen, Каталог 2002 г., с.159].
С целью усиления экспрессии трансгенов было предложено вводить в его состав такие регуляторные элементы, как интронные последовательности различных генов, последовательности инициации трансляции, посттранскрипционные и транспортные элементы и т.д. [Romano G. "Current development of nonviral-mediated gene transfer." Drug News Pespect. (2007), v.20, pp.227-231]. Каждый из подобных элементов в отдельности позволяет увеличить экспрессию трансгена, причем величина эффекта, как правило, зависит от выбранной модельной системы экспрессии.
В качестве интронной последовательности, в частности, используется последовательность IVS2 из гена бета-глобина кролика, которая помещается в плазмидный вектор непосредственно после промотора. Данный элемент используется, например, в плазмидном векторе pVSV-G компании Clontech, предназначенном для экспрессии белка оболочки вируса везикулярного стоматита. По опубликованным данным подобный элемент позволяет усилить экспрессию трансгена в 1,6 раз
Наиболее биобезопасными векторами считают плазмиды – двухцепочечные кольцевые молекулы ДНК, обладающие малой иммуногенностью и не способные к интеграции в геном клетки-мишени, что определяет их онкологическую безопасность. Однако, несмотря на перечисленные достоинства, плазмидным векторам присущ ряд недостатков, таких как низкая эффективность трансфекции клеток и кратковременная экспрессия трансгенов. Альтернативой плазмидным векторам служат вирусные конструкции. Вектор состоит из следующих элементов: промотор ранних генов цитомегаловируса человека (CMV), интрон, последовательность инициации трансляции, участок клонирования, посттранскрипционный регуляторный элемент, сигнал полиаденилирования мРНК гена гормона роста быка, сигнал полиаденилирования мРНК ранних генов вируса SV40, ген β-лактамазы, обеспецивающий устойчивость к ампициллину, участок инициации репликации Col E1. Элементы вектора перечислены в порядке их расположения.
25
Полиморфизм ДНК — полиморфизм молекул ДНК вследствие мутаций, происходящих в некодирующих последовательностях, таких как сателлитная ДНК, умеренные повторы, интроны, спейсерные участки между генами. Эти мутации имеют нейтральный характер, они непроявляются фенотипически и не влияют на жизнеобеспечение особи, т. е. они мало подвержены давлению естественного отбора. Современная молекулярная генетика применяет полиморфизм «эгоистической» ДНК в качестве информативных и чувствительных маркеров локализации генов, наличия мутаций, получения генетического отпечатка отдельных индивидуумов. Наиболее популярным методом является метод ПДРФ (полиморфизм длины рестрикционных фрагментов, см.), в основу которого положен полиморфизм в специфических участках генома — сайтах узнавания для действия ферментов эндонуклеаз. Полиморфизм вданных участках обусловлен возникновением нейтральных точковых мутаций, затрагивающих уникальные последовательности некодирующих участков ДНК, в которых либо исчезают, либо проявляются сайты узнавания. Полиморфные локусы встречаются во всех хромосомах с частотой один полиморфный сайт на300-500 п. н., и данный тип изменчивости ДНК исторически стал использоваться намного раньше других маркеров для характеристики специфических участков генома.
ДНК-ма́ркеры (ДНК-маркёры), или молекулярно-генетические маркеры — полиморфный признак, выявляемый методами молекулярной биологии на уровне нуклеотидной последовательности ДНК для определенного гена или для любого другого участка хромосомы при сравнении генотипов различных особей, пород, сортов, линий.
За последние годы накопилось много данных об эффективности использования молекулярно-генетических маркеров на уровне как белков, так и ДНК, РНК, для решения многих задач генетики, селекции, сохранения биоразнообразия, изучения механизмов эволюции, картирования хромосом, а также для семеноводства и племенного дела.
Наиболее широко используемые молекулярно-генетические маркеры условно можно подразделить на следующие типы — маркеры участков структурных генов, кодирующих аминокислотные последовательности белков (электрофоретические варианты белков), маркеры некодирующих участков структурных генов и маркеры различных последовательностей ДНК, отношение которых к структурным генам, как правило, неизвестно — распределение коротких повторов по геному (RAPD — случайно амплифицируемая полиморфная ДНК; ISSR — инвертированные повторы; AFLP — полиморфизм в сайтах рестрикции) и микросателлитные локусы (тандемные повторы с длиной элементарной единицы в 2-6 нуклеотидов).
В настоящее время насчитывается несколько десятков типов молекулярных маркеров. Наиболее широко используемые ДНК-маркеры перечислены. Их разделяют на три группы, согласно основному методу анализа:
Основные классы молекулярных маркеров
AFLP (amplified fragment length polymorphism) – полиморфизм длины амплифицированных фрагментов.
CAPS (cleaved amplified polymorphic sequences) – расщепленные амплифицированные полиморфные последовательности.
DArT (diversity array technology) – ДНК-чип технология для изучения разнообразия.
IRAP (inter-retrotransposon amplified polуmorphism) – полиморфизм амплифицированных последовательностей между ретротранспозонами.
ISSR (inter simple sequence repeats) – межмикросателлитные последовательности.
RAPD (random amplified polymorphic DNA) – случайно амплифицированная полиморфная ДНК.
RFLP (restriction fragment length polymorphism) – полиморфизм длины рестрикционных фрагментов.
SCAR (sequence characterized amplified region) – амплифицированная область, охарактеризованная нуклеотидной последовательностью.
SNP (single-nucleotide polymorphism) – однонуклеотидный полиморфизм.
SSAP (sequence-specific amplification polymorphism) – полиморфизм сиквенс-специфичной амплификации.
SSCP (single strand conformation polуmorphism) – полиморфизм конформации одноцепочечной ДНК.
SSR (simple sequence repeats) – простые повторяющиеся последовательности (микросателлиты).
STS (sequence tagged site) – сайт/локус, маркированный нуклеотидной последовательностью.
Среди молекулярных маркеров различают маркеры с известной локализацией (в определенной хромосоме или участке хромосомы, или вблизи конкретного гена) и маркеры, о локализации которых ничего не известно (как правило, это мультилокусные маркеры). Как те, так и другие находят свое применение в генетических исследованиях и в селекции. Молекулярные маркеры с неизвестной локализацией нельзя использовать для маркирования определенного гена или хромосомы, зато их успешно применяют в филогенетических исследованиях, для паспортизации сортов растений и пород животных. Некоторые мультилокусные маркеры подходят для создания генетических карт (DArT и AFLP -маркеры), а также для геномной селекции (DA T). На выбор ДНК-маркеров подходящего типа для решения конкретной задачи влияют и такие характеристики, как уровень внутривидового полиморфизма и возможность автоматизации процесса анализа полиморфизма ДНК
Основные направления использования монолокусных маркеров:
–составление молекулярных карт хромосом и геномов; – картирование генов и QTL;
– маркирование генов, хромосом и геномов;
–сравнительная генетика и геномика;
–отбор с помощью ДНК-маркеров в селекции;
–геномная селекция (только SNP-маркеры);
–молекулярная паспортизация сортов/ пород;
–диагностика заболеваний;
–экологический мониторинг;
–исследование генетического разнообразия;
– филогенетические исследования
– популяционная генетика.
Основные направления использования мультилокусных маркеров:
– составление молекулярных карт хромосом и геномов (только AFLP- и DArT-маркеры);
– картирование генов и QTL (только AFLPи DArT-маркеры);
– геномная селекция (DArT-маркеры);
– молекулярная паспортизация сортов/ пород;
– экологический мониторинг;
– исследование генетического разнообразия;
– филогенетические исследования;
– популяционная генетика.
Рестрикция ДНК — это разрезание молекулы ДНК специфическими ферментами, которые называются эндонуклеазами рестрикции, или, на лабораторном жаргоне, — рестриктазами.
Рестриктазы были найдены у бактерий. Все рестриктазы разрезают фосфодиэфирную связь между соседними нуклеотидами в ДНК. Рестриктные фрагменты (фрагменты ДНК, образовавшиеся в ходе рестрикции) имеют фосфатную группу на 5’-конце и гидроксильную группу на 3’-конце. Каждая рестриктаза имеет свой специфический сайт узнавания.
Все рестриктазы делятся на несколько групп. Рестриктазы из разных групп различаются сайтами узнавания, местом разрезания относительно сайта узнавания и структурой молекул фермента.
Эндонуклеазы рестрикции
Большинство методик в генной инженерии включают выделение определенных фрагментов ДНК (DNA) и последующее их соединение с другими фрагментами для получения новых комбинаций генов. Для этих целей используются ферменты, которые специфически разрезают и вновь сшивают молекулы ДНК. Наиболее важной группой ферментов являются эндонуклеазы рестрикции (рестриктазы), катализирующие специфическое расщепление двунитевой ДНК. Известно большое число рестриктаз. Для их обозначения используются сокращенные названия микроорганизмов - продуцентов. В качестве примера рассмотрим EcoRI — эндонуклеазу, выделенную из Escherichia coli. Подобно многим другим рестриктазам, этот фермент расщепляет ДНК по палиндромной последовательности, т. е. короткому сегменту ДНК, в котором обе цепи при считывании в направлении 5'→3' имеют одинаковую последовательность. Для EcoRl это последовательность 5'-GAATTC-3'. Гомодимер EcoRI расщепляет фосфодиэфирные связи обеих цепей между G и А. Это приводит к образованию комплементарных «липких» концов (ААТТ), которые удерживаются вместе за счет спаривания оснований. Их, однако, можно легко отделить друг от друга путем небольшого нагревания. При охлаждении липкие концы гибридизуются вновь в правильной ориентации. Места расщепления можно соединить с помощью ДНК-лигазы.
Б. Клонирование ДНК
Обычно содержание в клетке какого-либо сегмента ДНК, например отдельного гена, очень незначительно. Поэтому для проведения экспериментов с фрагментами ДНК их необходимо многократно копировать (клонировать). В классической методике клонирования ДНК используется способность клеток бактерий поглощать и реплицировать короткие кольцевые молекулы ДНК, известные как плазмиды. Сначала клонируемый фрагмент ДНК вырезается из исходной ДНК с помощью рестриктазы (см. выше). Для демонстрации метода на схеме показано расщепление с помощью EcoRI. На практике обычно используются два разных фермента. В качестве переносчика («вектора») служит плазмида с единственным участком, узнаваемым EcoRI. Кольцевая плазмида линеаризуется с помощью EcoRI и затем смешивается с изучаемым фрагментом ДНК. Поскольку фрагмент и вектор имеют одинаковые липкие концы, некоторые из молекул будут гибридизоваться таким образом, что клонируемый фрагмент окажется интегрированным в структуру вектора. Затем концы линейной молекулы ковалентно сшиваются с помощью ДНК-лигазы с образовании новой («рекомбинантной") плазмиды. При обработке большого количества клеток некоторые из них поглощают рекомбинантную плазмиду (так называемая трансформация). Трансформированные клетки реплицируют плазмиду вместе с собственным геномом. Обычно используют плазмиды, придающие трансформированной клетке устойчивость (резистентность) к определенному антибиотику. При инкубации популяции клеток в присутствии антибиотика будут реплицироваться только те клоны, которые содержат плазмиду. Из полученного клона выделяют плазмиду и после расщепления рестриктазой EcoRI получают множество копий клонированного фрагмента ДНК
26
Типы ДНК-маркеров. ДНК-полиморфизм тестируют различными способами, включая прямое определение нуклеотидной последовательности интересующего исследователя участка (секвенирование). ДНК-маркерные технологии условно можно разделить на две основные группы: с применением полимеразной цепной реакции (ПЦР) и без такового. Использование полиморфизма длины рестриктных фрагментов (restriction fragment length polymorphism-RFLP) является основным методом молекулярного маркирования без применения ПЦР. Для анализа данного полиморфизма выделенная из растительных тканей ДНК «разрезается» специфичными бактериальными ферментами – рестриктазами, мишенью которых служат короткие специфические последовательности ДНК – сайты рестрикции [7]. Продукты рестрикции разделяют электрофорезом в агарозном геле. После электрофоретического разделения их переносят из агарозного геля на нейлоновую мембрану с помощью Саузерн-блоттинга, «проявляют» путем гибридизации с радиоактивными зондамифрагментами ДНК и затем анализируют по положению на радиоавтографах [8]. В качестве зондов могут служить случайные продукты рестрикции геномной ДНК, однако чаще всего используют клоны уникальных и редко повторяющихся последовательностей ДНК, применяемых для составления молекулярных карт геномов и физического картирования генов [9]. В отличие от морфологических маркеров RFLP-маркеры кодоминантны, что позволяет различать гомо- и гетерозиготы. С применением данной маркерной системы составлены генетические карты для многих видов растений. У риса, например, картировано 3500–4000 RFLP-маркеров [10].
ДНК-фингерпринтирование (DNA fingerprinting, DNA fingerprint technique — метод создания генетических «отпечатков пальцев», основанный на анализе полиморфизма ДНК. Первоначально геномная ДНК рестрицируется эндонуклеазами, затем образующиеся фрагменты разделяются при помощи электрофореза в геле и переносятся на фильтры (напр., нитроцеллюлозные фильтры). После этого фильтры гибридизуют со специфическими мечеными зондами (ДНК фага М13, различные синтетические олигонуклеотиды, мини- и микросателлитные ДНК и др.). Фрагменты ДНК, гомологичные зондам, образуют полиморфные полосы гибридизации, отдельные из которых специфичны для каждого индивидуума, поэтому метод может быть использован для генетической идентификации разных индивидуумов одного вида. ДНК-ф. применяется при картировании генов, определении отцовства и материнства, в криминалистике. Метод ДНК-ф. предложен А. Джеффрисом в 1985 г.
27
Гибридизация нуклеиновых кислот
Многие этапы анализа рекомбинантной ДНК основаны на комплементарности взаимодействия цепей нуклеиновых кислот - необходимом условии синтеза ДНК и РНК. Путем нагревания или обработки щелочью двухцепочечную ДНК разделяют на отдельные цепи (денатурированная ДНК). Денатурированную ДНК инкубируют в условиях, обеспечивающих гибридизацию нуклеиновых кислот, то есть повторное образование двухцепочечных молекул путем спаривания нуклеотидов комплементарных цепей. Эта реакция настолько специфична, что гибрид одноцепочечной молекулы ДНК с комплементарной цепью (РНК или ДНК) можно выявить, даже если кДНК составляет лишь одну десятитысячную часть от общего количества ДНК.
Метод позволяет различить полностью и частично гомологичные последовательности.
Специфичность гибридизации нуклеиновых кислот, часто в сочетании с фракционированием или амплификацией, позволяет выявить нужный ген среди десятков тысяч других или нуклеиновую кислоту возбудителя инфекции даже тогда, когда единственная ее копия приходится на несколько клеток человека.
Для выявления гибридизационных зондов используют радиоактивную метку или нерадиоактивные методы.
Часто применяется гибридизация нуклеиновых кислот с аллель-специфическими зондами. Такой зонд представляет собой синтетический одноцепочечный олигонуклеотид, обычно длиной 15-20 нуклеотидов. Синтезируют два зонда, различающиеся одним нуклеотидом. Один из них точно соответствует нормальной последовательности, а другой - мутантной; в последнем замещенный нуклеотид расположен в средней части зонда. Условия гибридизации подбирают таким образом, что олигонуклеотид связывается только с идеально комплементарной ему последовательностью. Аллель-специфические олигонуклеотидные зонды можно использовать в сочетании с блоттингом по Саузерну , но сейчас их чаще используют в сочетании с амплификацией ДНК.
Блоттингом (буквально - промакивание) называют перенос фрагментов макромолекул (ДНК, РНК или белка), разделенных с помощью электрофореза в геле, на твердую подложку - мембрану. В исследованиях генома человека часто используют метод блоттинга, разработанный Саузерном, при котором олигонуклеотидный зонд, находящийся в растворе, гибридизуется с ДНК, адсорбированной на мембране. Геномную ДНК (обычно выделенную из лейкоцитов или клеток плода) расщепляют на короткие фрагменты, разделяют их в агарозном геле, переносят на мембрану, после чего идентифицируют специфические участки с помощью гибридизации с олигонуклеотидными зондами. Этим методом выявляют уникальные фрагменты ДНК, размер которых составляет приблизительно одну миллионную часть генома.
Значимость метода для медицины обусловлена возможностью исследовать определенный фрагмент геномной ДНК у любого человека.
Метод используют для выявления крупных перестроек в ДНК и некоторых точечных мутаций (большинство точечных мутаций этим методом выявить нельзя).
Денатурация ДНК осуществляется с помощью щелочи. После окончания электрофореза гель помещают в растворор основания (щелочи), в котором двухчепочечные фрагменты ДНК теряют связи и становятся одноцепочечными.
-Перенос ДНК с геля на нитроцеллюлозный или нейлоновый фильтр производиться в буферном растворе. Непосредственно на поверхность геля кладут фильтр и стопку фильтровальной бумаги. За счет капиллярного эффекта создается ток буфера, перпендикулярный плоскости геля. Вымываемая из геля ДНК задерживается фильтром и практически полностью оказывается на его поверхности. После переноса одночепочечные нити фиксируют на фильтре. Расположение фрагментов на фильтре точно соответствует их расположению в геле. 3. Для того чтобы визуально выявить нужные фрагменты (фиксированная на фильтре ДНК не видна), проводят гибридизацию со специфическим по нуклеотидной последовательности меченным радионуклидам или флюоресцентной меткой олигонуклеотидным синтетическим зондом (такой зонд состоит из 16-30 пар осваний), или клонированным фрагментом ДНК. Нуклеотидная последовательность зонда должна быть полностью или частично комплементарна изучаемому участку геномной ДНК.
При инкубации фильтра с раствором, содержащим меченый зонд, происходит гибридизация комплементарных цепей ДНК зонда и фрагмента на фильтре. Неспецифические связанные молекулы зонда отмываются с помощью специальной процедуры.
Радиоактивно меченные участки выявляют путем экспонирования фильтра с рентгеновской пленкой (ауторадиография). После проявления на пленке видны полосы меченной зондом ДНК.
Нерадиоактивные метки визуализируют с помощью флюоресценции или опосредованно с помощью антител.
Нозерн-блоттинг — метод, сходный с методом Саузерн-блот-гибридизации, но используемый применительно к РНК. Суммарный препарат РНК первоначально разделяют электрофоретически в геле, а затем переносят на нитроцеллюлозный или нейлоновый фильтр. После фиксации РНК на фильтре с помощью спекания или УФ-облучения этот фильтр используют для гибридизации с одноцепочечными мечеными зондами. Метод позволяет выявлять индивидуальные РНК в сложной популяции этих молекул; предложен Дж. Олвайном с сотр. в 1977 г. Название метода основано на игре слов: как противоположное ранее предложенному Е. Саузерном (в переводе — Южный) методу анализа ДНК.
Методы секвенирования: по Максаму-Гилберту; по Сенгеру, современные методы секвенирования.
1.секвенирование по методу Максама-гилберта заключается в том, что выделенная ДНК, радиоактивно помеченная с одного конца (либо 3’ либо 5’), с помощью специальных химических веществ разбивается на одноцепочечные фрагменты.
Затем, полученный препарат делят на 4 части и в каждую добавляют вещества, которые модифицируют одно, либо 2 основания (либо А+Г, либо Ц+Г, либо только Г, только Ц. Отдельно модифицировать А или Т на сегодняшний момент невозможно).
В итоге получается так. Представьте, что у вас есть пробирка с ДНК, вы в нее добавляете химикат, который выщепляет=уничтожает все Гуанины (Г). В цепи их много, соответсвенно, она рвется много раз на разных расстояниях от меченого конца. Выходит, что есть у вас например отрезки длиной в 7, 10. 13 и 16 нуклеотидов, каждый из них радиоактивно помечен (это важно, чтобы потом их было видно при получении результатов на рентгеновской пленке) и на конце каждого у вас отщепился Гуанин. Затем вы эту смесь разгоняете на электрофорезе и все эти кусочки светятся и расходятся как по линеечке на разные расстояния. Вы, глядя на рентгеновскую пленку, на которую перенесли результаты фореза за счет свечения радиоактивных кусочков, можете сказать: ага, вот тут отрезок длиной в 7 нуклеотидов, значит, в анализируемом мною отрезке ДНК Гуанин стоял на 8 месте. Идете дальше, и видите: ага, тут гуаниновый отрезок светится на длине в 10 нуклеотидов, значит, на 11м месте тоже стоял он.
И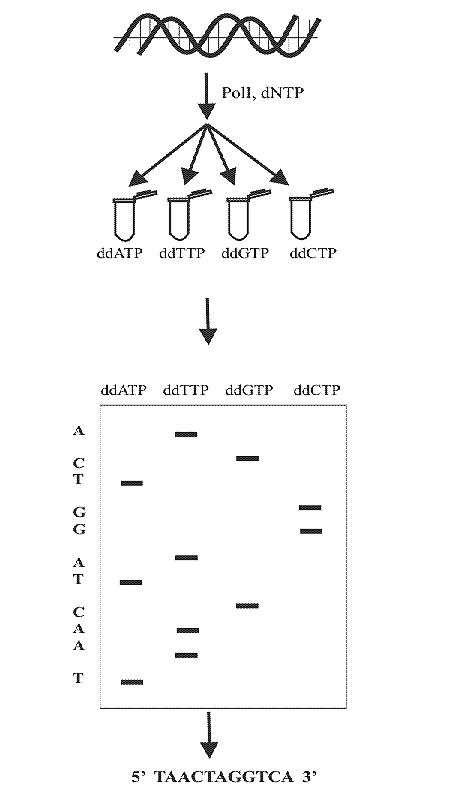 точно так же вы делаете со всеми 4-мя
пробирками: вы заливаете в каждую из 4
ячеек по одному раствору с мечеными
Г,Ц,А,Т. В итоге выходит такая лесенка,
у которой внизу самые короткие участки,
а вверху самые длинные. Идя снизу вверх
вы «читаете» последовательность
точно так же вы делаете со всеми 4-мя
пробирками: вы заливаете в каждую из 4
ячеек по одному раствору с мечеными
Г,Ц,А,Т. В итоге выходит такая лесенка,
у которой внизу самые короткие участки,
а вверху самые длинные. Идя снизу вверх
вы «читаете» последовательность
2. По Сенгеру метод похож, разница только в том, что мы не рвем уже имеющиеся цепи ДНК, а в 4х пробирках синтезируем комплиментарную матричной цепь. Соответсвенно, в каждой пробирке есть праймеры, 4 дезоксирибонуклеотида (А,Т.Г,Ц), а также немного радиоактивно помеченных ДИдезоксирибонуклеотида
Суть метода в том, что в каждую пробирку добавляется какой-то один ДИдезоксирибонуклеотид (по факту, почти тоже самое что и обычный А/Г/Т/Ц, но только с одного конца у него должна быть группа ОН, к которой прикрепляется следующий нуклеотид, но вместо ОН, там стоит просто Н. Его так же называют просто терминатором). Соответственно, на каждом таком нуклеотиде синтез останавливается. Получаются цепочки разных длин, каждая из которых заканчивается в одной пробирке либо А, либо Г и тд.
Дальше совершенно так же делается форез с формированием «лесенки». Это один из двух методов Сенгера,, более распространенный. Его называют методом терминации.
Второй метод – +/- секвенирование. В нем учавствует 8 пробирок, там тоже проводят ПЦР, но вместо того чтобы добавлять терминаторы, в 4 пробирки по отдельности в каждую добавляют по одному основанию и в 4 по отдельности не добавляют по одному какому-то основанию. Дальше проводят форез, получают лесенку.
3. Современное секвенирование
Все современные метода основаны на одном принципе: сиквенировании относительно небольших участков ДНК, испульзуя циклические ферментативные реакции с дальнейшим сбором информации в виде иллюстраций. Полученные данные используются для восстановления нуклеотидной последовательности. Методы отличаются точностью и длиной отрезков ДНК, которые можно «прочесть» этими способами, но общая схема работы одинаковая.
В них первый этап – получение кучи отдельных коротких последовательностей, которые затем можно будет сшит в одну последовательность.
Второй – создание ампликонов с помощью ПЦР, которые будут использованы как образцы (ампликон – это источник/либо продукт амплификации/репликации)
Третий – определение нуклеотидной последовательности всех фрагментов.
Примеры принципов различных современных методов: Пиросеквенирование, ионное полупроводниковое секвенирование, С.на основе лигирования.
29. Выделение ДНК. Особенности выделения ДНК из растительных клеток. Основные этапы выделения тотальной ДНК (лизис клеток, состав буфера для экстракции, депротеинизация и удаление примесей, преципитация (осаждение) ДНК, очистка, сушка и растворение). Хранение препаратов ДНК.
Общая схема выделения ДНК для всех организмов одинаковая. У растений особенность заключается в том, что есть клеточная стенка с кучей трудноразрушаемых веществ типа целлюлозы, а так же крупные вакуоли, содержащие кучу сахаров, аминокислот, дубильных веществ и тд.
Основные этапы:
Механическое разрушение клеток (например с помощью пестика в пробирке)
Химический лизис мемран клетки с помощью детергента (все происходит в буфере, чтобы поддерживать определенную кислотность и не повредить ДНК). В качестве детергента можно использовать например SDS или СТАВ.
Депротеинизация (избавление от белков в растворе и других примесей). Кроме белков из разных частей клетки есть еще глобулины, которые связаны с самой ДНК, а так же ферменты – ДНКазы, которые могут разрушить «оголенную» ДНК при проведении всех этих процедур.
Дальше нужно процентрифугировать раствор, добавив в него то, на что будет осаждаться ДНК (например кремнеевую мембрану).. Этот этап называется преципитацией = осаждением ДНК. . При этом используется спирт (этанол, либо изопропанол (96%)).
Потом, убрав все примеси, надо добавить буфер, который снимет ДНК с компонента, на который мы ее осадили (это называется элюция – выведение в р-р адсорбированного вещества).
Хранится препарат с ДНК при температуре -20 * С.
Тотальная ДНК – вся ДНК клетки (из ядра и митохондрий, либо хлоропластов)
