
- •Список исполнителей
- •Содержание
- •Перечень сокращений, условных обозначений, символов, единиц, терминов
- •Общие данные об объекте исследований
- •Аналитическая часть
- •Заключение
- •Задание № ____ на проведение патентных исследований
- •Календарный план
- •Форма регламента поиска
- •Форма отчета о поиске
- •Способ испытания ели
- •Растения огурца, устойчивые к заболеваниям
- •Способы семеноводства с использованием высокопродуктивного неразрушающего отбора проб семян
- •Способ и аналитический набор для демонстрации генетической идентичности
- •Новый тип генетического маркера на основе транспозона
- •Способ молекулярного маркирования пола хмеля обыкновенного (humulus lupulus l.)
- •Биологический днк маркер для определения сортов картофеля, набор и способ сортовой идентификации картофеля
- •Способ паспортизации и контроля за генетической изменчивостью в клеточных культурах различной длительности пассирования
Способ и аналитический набор для демонстрации генетической идентичности
Изобретение относится к области молекулярной биологии и может быть использовано для генетического анализа. Демонстрацию генетической идентичности, генетического разнообразия, геномных вариаций или полиморфизмов и кодоминантную оценку в пределах определенного популяционного пула осуществляют с помощью способа, включающего получение и оценку результатов гибридизации фрагментированного образца ДНКс олигонуклеотидами, способными специфически идентифицировать присутствие и отсутствие мобильного генетического элемента в геномной позиции. Указанные олигонуклеотиды используют также в составе аналитического набора для демонстрации генетической идентичности, генетического разнообразия, геномных вариаций или полиморфизмов и кодоминантной оценки в пределах определенного популяционного пула. Данный аналитический набор, а также указанный способ применяют для различения индивидуумов, а также для гарантированной и ускоренной селекциирастенийили животных. Применение изобретения обеспечивает надежное воспроизводимое получение распределениямаркеровс недорогой и технически простой системой детекции. 6 н. и 12 з.п. ф-лы, 28 ил., 2 табл.
ПРИЛОЖЕНИЕ К
Заявка на изобретение: 200129366
Новый тип генетического маркера на основе транспозона
Изобретение относится к области молекулярной биологии и биотехнологии и может быть использовано в процессах генотипирования. Предложен способ фингерпринтинга эукариотического организма. Последовательность нуклеиновой кислоты эукариотического организма амплифицируют с помощью праймера, гибридизующегося в жестких условиях либо с миниатюрным транспозируемым элементом, либо с его фрагментом или производным. Продукты амплификации разделяют по массе. Результаты указанного разделения используют для определения эукариотического организма. Применение изобретения позволяет осуществить фингерпринтинг с высокой эффективностью и разрешающей способностью. 7 з.п. ф-лы, 3 табл., 18 ил.
ПРИЛОЖЕНИЕ Л
Описание изобретения к патенту: 2272840
Способ молекулярного маркирования пола хмеля обыкновенного (humulus lupulus l.)
Изобретение относится к областимолекулярнойбиологии, в частности при селекциихмелядля определенияполарастений на стадии проростков. Определяют пол гибридных растенийхмеляна стадии сеянцев с помощьюмолекулярногомаркирования, где используют две комбинации олигонуклеотидных праймеров для проведения полимеразной цепной реакции, которые обеспечивают амплификацию специфичных для мужских растений фрагментов ДНК. Изобретение повышает эффективность селекции и позволяет сократить затраты труда, средств, площадей и времени. 3 ил., 1
ПРИЛОЖЕНИЕ М
Описание изобретения к патенту: 2244416

ПРИЛОЖЕНИЕ Н
Описание изобретения к патенту: 2413774
Биологический днк маркер для определения сортов картофеля, набор и способ сортовой идентификации картофеля
Для определения сортовой принадлежности растений картофеля выделяют ДНК из свежего растительного материала и исследуют ее с помощью ПЦР. ПЦР проводят, используя набор, содержащий биологический маркер, реакционную смесь, состоящую из 160 мкМ каждого динуклеотидтрифосфата dNTP, 1.6 мМ MgCl2, 0.3 мкМ каждого праймера из набора, 0.3 единицы фермента Taq-полимеразы, 1× буфера для Taq-полимеразы и эталонные ДНК известных сортов картофеля в количестве 20-50 нг. Получают полиморфные маркеры ДНК, визуализируют их с помощью электрофореза в геле, а затем для типирования выявленных в изученной группе аллелей проводят сравнение аллельных спектров анализируемой и контрольной групп образцов. Биологический маркер, содержащий полиморфную ДНК, представляет собой специфическую нуклеотидную последовательность, получаемую при использовании диагностического набора из трех пар праймеров и характеризует сортовую принадлежность. наиболее распространенных и хозяйственно-значимых сортов картофеля, а также образцов родственных видов Solarium, наиболее часто используемых в скрещиваниях при создании новых сортов картофеля. 3 н. и 2 з.п. ф-лы, 3 ил., 4 табл.
Изобретение относится к биотехнологии, в частности к молекулярно-генетическим исследованиям, к области, связанной с сортовой идентификацией картофеля, контроля сортовой однородности картофеля, интрогрессиигенетическогоматериала в потомстве от скрещиваний при создании новых сортов.
Известен способ молекулярного маркирования и идентификации пола хмеля обыкновенного с использованием двух комбинаций олигонуклеотидных праймеров для проведения полимеразной цепной реакции, которые обеспечивают амплификацию специфичных для мужских растений фрагментов ДНК (RU 2272840 (13) 2004.03.09). Недостатком этого способа является гено- и геномспецифичность праймеров, использование которых возможно только на хмеле и только для определения пола растений хмеля.
Известен способ молекулярного маркирования селекционных достижений клевера лугового на основе RAPD-маркеров в оценке ДНК-полиморфизма RAPD-методом с использованием набора 10-членных праймеров (RU 2244416 (13) 2002.02.06). Недостатком этого способа является малая воспроизводимость самого RAPD-метода и зачастую невозможность использования данных, полученных в разных лабораториях.
Известен способ идентификации гибридов сельскохозяйственных культур путем определения аллельного состояния исследуемых локусов по продуктам амплификации, полученным с использованием праймеров к подобранным в тест-системе маркерам, сравнение генотипа образца с известными генотипами, причем отдельно выделяют ДНК и идентифицируют генотип материнской ткани семени и отдельно выделяют ДНК и идентифицируют генотип гибридной ткани семени (UA 79346 (13) 2005.07.18). Недостатком этого способа является возможность его использования только для идентификации семян гибридов сельскохозяйственных культур и непригодность идентификации сортов картофеля.
Известны способы идентификации генотипов ячменя на основе использования системы микросателлитных ДНК-маркеров (UA 67905 (13) 2003.03.20), праймеров к LTR-последовательности ретротранспазонов ячменя (UA 15273 (13) 2006.01.03). Также известны способы регистрации и паспортизациигенотипов ячменя на основе анализа компонентов электрофоретического спектра, получаемого с помощью полимеразной цепной реакции IRAP (UA 15277 (13) 2006.01.03), реакции REMAP (UA 16082 (13) 2006.02.20), дифференциации генотипов сортов ячменя на основе полимеразной цепной реакции с праймерами, гомологичными фрагментам ретротранспазонов (LTR-праймеры) и рядом расположенного простого микросателлитного повтора (ISSR-праймеры) (UA 16084 (13). Вид документа U (14). Дата публикации 2006.02.20).
Недостатками данных способов является геном специфичность (ячмень) предлагаемых праймеров и, следовательно, непригодность их для сортовой идентификации картофеля.
Наиболее близким к предлагаемому способу определения сортовой принадлежности культурных растений найдены следующие способы.
Способы оценки типичности инбредных линий и уровня гибридности семян F1 подсолнечника по микросателлитным локусам НА 432, НА 514, НА 1442 и ORS 5 с использованием праймеров, соответствующих фланкирующим областям этих локусов (RU 2294965 (13) 2005.07.04).
Известен способ определения типичности и уровня гибридности генотипов подсолнечника путем определения процента типичных для определенной линии или гибрида растений путем выделения ДНК из трехдневных проростков 100 семян, проведения амплификации определенных микросателлитных локусов в полимеразной цепной реакции, распределения продуктов реакции в 10% полиакриламидном геле и сравнения электрофоретических спектров и подсчета количества растений с одинаковым аллельным составом (UA 63265 (13) 2003.03.20)
Известен способ классификации ржи по типу молекулы ДНК на основе анализа молекулы ДНК, выделенной из тканей зерна, ростков или других тканей, определение чистоты повторения аллелей микросателлитных локусов в различных сортах ржи и определение генотипа сорта в виде формулы (UA 10230 (13) 2005.03.23).
Известен способ идентификации генотипов подсолнечника с помощью определения и записи набора аллелей по микросателлитным локусам генома путем их амплификации в полимеразной цепной реакции и электрофореза в полиакриламидном геле. (UA 68813 (13) 2003.10.30).
Недостатками известных способов является геном специфичность (подсолнечник, рожь) предлагаемых праймеров и, следовательно, их непригодность для сортовой идентификации картофеля, а также отсутствие праймеров и набора для идентификации и паспортизации сортов картофеля, невозможность созданиягенетическогопаспорта сортов картофеля.
Таким образом, изобретение - биологический ДНК маркер должен быть а) специфичен для картофеля, то есть быть пригодным для анализа генотипов картофеля; б) применим для идентификации и паспортизациисортов картофеля, то есть должен выявлять индивидуальныегенетическиеособенности сортов картофеля.
Изобретение также должно предполагать наличие набора пригодного для анализа генотипов картофеля, а также способа, обеспечивающего быстроту и точность анализа.
Задачей предлагаемого изобретения является определение сортовой принадлежности наиболее распространенных и практически значимых сортов картофеля (Solaum tuberosum), а также образцов родственных видов Solanum наиболее часто используемых в скрещиваниях при создании новых сортов картофеля и составление паспорта этих сортов картофеля.
В результате использования предлагаемого биологического маркера для сортовой идентификации картофеля, набора и способа идентификации появляется возможность создания молекулярно-генетическогопаспорта сорта и повышение эффективности сортовой идентификации в генетико-селекционных мероприятиях, связанных с выведением новых сортов и размножением уже имеющихся элитных растений сортов.
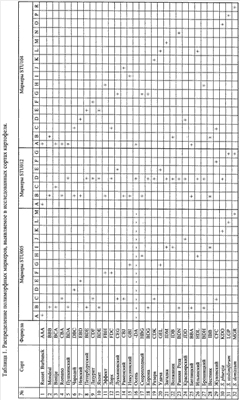
В таблице представлено распределение полиморфных биологических ДНК маркеров у сортов картофеля, а также аллельные формулы сортов, представляющие индивидуальные генетическиехарактеристики (паспорта) каждого сорта. С помощью предлагаемого набора биологических ДНК маркеров и способа получения индивидуальныхгенетическиххарактеристик осуществляется сортовая идентификация картофеля.
Вышеуказанный результат достигается тем, что биологический маркер, содержащий полиморфную ДНК, характеризует сортовую принадлежность наиболее распространенных и хозяйственно значимых сортов картофеля и представляет собой специфическую нуклеотидную последовательность, получаемую при использовании диагностического набора из следующих трех пар праймеров

Результат достигается также тем, что используют набор для определения сортовой принадлежности растений картофеля, содержащий биологический маркер, реакционную смесь, состоящую из 160 мкМ каждого динуклеотидтрифосфата dNTP, 1.6 мМ MgCl2, 0.3 мкМ каждого праймера из набора, 0.3 единицы фермента Taq-полимеразы, 1× буфера для Taq-полимеразы и эталонные ДНК растения картофеля в количестве 20-50 нг.
В способе определения сортовой принадлежности растений/клубней картофеля - выделение ДНК из свежего растительного материала и анализ геномной ДНК картофеля с помощью полимеразной цепной реакции (ПЦР).
ПЦР-анализ проводят, используя предлагаемый набор праймеров, получают полиморфные маркеры ДНК разного размера, визуализируют эти маркерные ДНК с помощью электрофореза в полиакриламидном или агарозном геле, затем для типирования выявленных в изученной группе аллелей проводят сравнение генетическогоразнообразия определяемой и контрольной групп образцов амплифицируемых фрагментов ДНК. Набор контрольных амплификатов содержит образцы амплифицированной ДНК, характерные для каждого из тестируемых сортов картофеля Solanum tuberosum. Сортовую принадлежность определяют по отсутствию или присутствию в исследуемой группе определенных сортоспецифических аллельных фенотипов. Размер амплифицируемых продуктов определяют с помощью маркера молекулярных масс (10 bp Ladder).
Результат достигается также тем, что для каждого сорта картофеля составляют молекулярно-генетическийпаспорт, представленный в таблице 1, в которой указано распределение выявленных с помощью трех пар праймеров (STU005F/STU005R, STU012F/STU012R, STU104F/STU104R) полиморфных микросателлитных маркеров, соответствующих локусам (STU005, STU012, STU104), по которым определяют сорт картофеля.
В предлагаемом способе верификацию сортовой принадлежности проверяют с помощью различных вариантов многомерного анализа (например, нейронных сетей Кахонена, кластерного анализа, метода главных компонент и т.п.), в которых в качестве обучающей выборки может служить выборка аллелей и аллельных фенотипов одного из сортов набора картофеля.
Таким образом, генетическая идентификация растений сортов картофеля как отечественной, так и зарубежной селекции с применением предлагаемых биологических маркеров и набора осуществляется в результате фингерпринтинга сортов или коллекционных образцов картофеля. Посредством предлагаемого способа получают молекулярно-генетическиехарактеристики сортов картофеля как отечественной, так и зарубежной селекции, а также коллекционных образцов близкородственных видов/подвидов/разновидностей картофеля рода Solarium ("секция Petota), определяют сортоспецифичные маркеры с цельюпаспортизациисортов. При изучении сортов картофеля и близкородственных видов/подвидов/разновидностей выявляют несколько сортоспецифичных маркеров, которые в дальнейшем используют в качестве эталона для определения сортовой принадлежности любого сорта картофеля.
На заключительном этапе для каждой идентифицируемой группы составляют таблицу-паспорт с указанием распределения выявленных аллельных фенотипов и их аллелей по трем полиморфным локусам. На основании этой таблицы с высокой вероятностью либо визуально, либо с помощью многомерного статистического анализа определяют принадлежность (или отсутствие принадлежности) исследуемого растения картофеля к одному из сортов.
Методикаиспользования изобретения
Для идентификации сортовой принадлежности сорта собирают живой растительный материал от 5-10 растений сорта (лучше молодые ткани - листья/почки). Наилучшим материалом для последующего выделения суммарной ДНК являются молодые ткани - листья/листовые или цветочные почки живых растений. Поэтому при наличии клубневого материала клубни предварительно проращивают до появления из глазковых почек 1-2 листков.
Выделение и хранение ДНК
1. Выделение ДНК из свежей растительной ткани
Выделение тотальной ДНК проводили по модифицированной методикеЭдвардса (Edwards et al., 1991) с дополнительными этапами депротеинизации смесью фенол/хлороформ (1:1). Выделение ДНК заключается в последовательном прохождении следующих фаз: лизис клеток и ядер, депротеинизация ДНК с помощью 5 М ацетата калия, фенол-хлороформная депротеинизация, осаждение и растворение ДНК.
2. Лизис клеток
К листовой высечке (~0.05 мг) в пробирке типа Eppendorf объемом 1,5 мл добавляют 400 мкл лизирующего буфера. Состав лизирующего буфера: 200 мМ трис-HCl, рН 7,5; 250 мМ NaCl; 25 мМ EDTA; 0,5% SDS. Растительный материал в буфере при комнатной температуре гомогенизируют с помощью специальных пестиков. Смесь инкубируют в термостате или водяной бане в течение 15-20 минут при 65°С.
3. Депротеинизация ДНК
В пробирку со смесью, содержащей лизированные клетки и ядра, добавляют 200 мкл предварительно охлажденного 5 М ацетата калия, смесь тщательно перемешивают и инкубируют на ледяной бане 10-20 мин. После инкубации смесь центрифугируют 15 мин при 16000 g. Полученный надосадочный супернатант (около 450 мкл) переносят в новую пробирку и подвергают двукратной очистке равным объемом смеси фенол/хлороформ. Смесь фенола и хлороформа приготавливают путем смешивания равных объемов фенола и хлороформа. Готовый фенол содержит 0.1% антиоксидантов 8-оксихинолина и 0.2% меркаптоэтанола. Готовый хлороформ представляет собой смесь хлороформа и изоамилового спирта (24:1). Супернатант и смеси фенол/хлороформ плавно перемешивают до образования эмульсии, а затем центрифугируют 5 минут при 16000 g. После чего верхнюю фазу с растворенной ДНК отбирают пипеткой, стараясь не захватывать интерфазу, содержащую белки и нижнюю фазу, содержащую фенол:хлороформ. Фенол-хлороформная депротеинизация повторяется 2-3 раза до полной очистки раствора ДНК от белков.
4. Осаждение ДНК
Осаждение ДНК производят путем добавления к очищенному супернатанту 0.6 V изопропанола; смесь перемешивают и центрифугируют в течение 15 мин при 16000 g. Спирт сливают и осадок промывают 70% этанолом не менее 3 раз, просушивают и растворяют в 20-25 мкл бидистилированной стерильной воды.
После определения концентрации и чистоты ДНК (на спектрофотометре/или в геле) концентрации ДНК в пробах стандартизируют путем разведения в стерильной бидистиллированной воды до 50 мкг/мкл.
Продолжительность выделения ДНК составляет 3 часа.
5. Оценка качества и количества экстрагированной ДНК
Оценку качества и количества экстрагированной ДНК производят путем измерения оптической плотности раствора ДНК на анализаторе типа Биофотометр (Bio Photometer Eppendorf) либо путем проведения аналитического электрофореза.
Измерение оптической плотности раствора ДНК при длине волны 260 нм позволяет установить концентрацию и чистоту нуклеиновых кислот. Оптическая плотность D=1 соответствует приблизительно 50 мкг/мл двуцепочечной ДНК. Отношение величины D при длине волны 260 нм к D при длине волны 280 нм позволяет судить о чистоте нуклеиновой кислоты. Чистые препараты ДНК имеют отношение D 260/D280, равное 1.6-1.8. Если препарат содержит примеси белка и/или фенола, то D260/D280значительно меньше этих значений. Соотношение D260/D230говорит о загрязнении полисахаридами. Для чистых препаратов ДНК это соотношение должно быть 1.8.
Определение концентрации и качества
выделенных образцов ДНК методом
электрофореза в агарозном геле проводится
в горизонтальной камере в 1хтрис-боратным буфере (ТВЕ), рН 7,5-7,8 (0,089
М трис, 0,089 М борной кислоты и 0,002 М ЭДТА).
При внесении ДНК в карманы геля
используется буфер для нанесения (0.25%
бромфеноловый синий, 40% (w/v) сахароза, 6
мМ ЭДТА). После электрофореза гель
окрашивают в растворе бромистого этидия
в течение 5-10 минут или добавляют бромистый
этидий непосредственно в гель. Вхождение
ДНК в гель осуществляют при 50 V в течение
15-20 минут, далее электрофорез ведут при
75-90 V в течение 1-2 часов. В качестве маркера
концентрации используется ДНК фага![]() ,
внесенная в разных количествах, например,
0,5; 1; 2 мкг.
,
внесенная в разных количествах, например,
0,5; 1; 2 мкг.
Полимеразная цепная реакция (ПЦР)
1. Проведение ПЦР
Выделенную ДНК используют в качестве матрицы в реакции амплификации. Амплификацию ДНК проводят в реакционной смеси объемом 15 мкл, содержащей 1х буфер из соответствующего набора, 0,16 мМ каждого dNTP, 1.2 mM MgCl2, 0.3 мкМ праймера, 0.3 ед. Taq полимеразы и 100 нг геномной ДНК в термоциклере «Applied Biosystems 2700» (США) в режиме: денатурация - 30 сек - 94°С; отжиг праймера - 45 сек - 50-60°С; синтез ДНК - 1 мин - 72°С (35 циклов) с предварительной денатурацией - 5 мин (94°С) и заключительной элонгацией PCR-фрагментов 10 мин - 72°С. Температура отжига для каждого праймера рассчитывалась отдельно по формуле Та=Tm-4, где температура плавления Tm=69.3+0.41(%GC)-650/L нуклеотидов, L - длина праймера (число нуклеотидов), %GC - процент GC нуклеотидов от длины праймера. Для проведения ПЦР предлагается использовать три пары праймеров следующего состава:

2. Электрофоретическое разделение продуктов и визуализация ПЦР-продуктов
Продукты амплификации подвергают
аналитическому электрофорезу в 1.7%
агарозном геле толщиной 5-7 мм, содержащем
бромистый этидий в 1х ТВЕ буфере. В
качестве маркеров молекулярного веса
используется O'Range Ruler![]() 100 bp DNA ladder Plus ("Fermentas", США). Вхождение
фрагментов в гель осуществляют при 50 V
в течение 10-20 минут, а разделение
фрагментов - в течение 1-2 часов при 75-90
V.
100 bp DNA ladder Plus ("Fermentas", США). Вхождение
фрагментов в гель осуществляют при 50 V
в течение 10-20 минут, а разделение
фрагментов - в течение 1-2 часов при 75-90
V.
В случае эффективной амплификации продукты амплификации подвергают электрофоретическому разделению в вертикальной камере (например, Sequi-Gen GT Sequencing Cell «Bio-Rad», США) в 6% ПААГе, содержащем 7% мочевину.
Визуализацию осуществляют путем
окрашивания гелей азотнокислым серебром
из набора Silver Sequence Staining Kit (Promega, США)
согласно инструкции производителя с
последующим фотографированием цифровым
фотоаппаратом Nicon либо любой другой
системы визуализации. Размер амплифицируемых
продуктов определяют с помощью маркера
молекулярных масс O'Range Ruler![]() 50 bp и DNA Ladder O'Range Ruler
50 bp и DNA Ladder O'Range Ruler![]() 10 bp DNA Ladder ("Fermentas", США).
10 bp DNA Ladder ("Fermentas", США).
Затем проводят статистическую обработку результатов и составляют сортовой молекулярно-генетическийпаспорт.
Для типирования аллельных вариантов (определения размера в п.н.), выявленных в изученной группе растений картофеля, необходимо провести обязательное сравнение генетическогоразнообразия данной и контрольной (эталонной) групп образцов амплифицируемой ДНК. Набор контрольных амплификатов может содержать в среднем от 5 до 20 образцов амплификатов, характерных для 5-20 микросателлитных аллельных вариантов. Отсутствие или присутствие в исследуемой группе генотипов растений определенных аллелей или аллельных сочетаний позволяет надежно определять сортовую принадлежность. Верификацию сортовой принадлежности проводят с помощью различных вариантов многомерного (кластерного анализа, нейронных сетей Кахонена, дискриминантного анализа или метода главных компонент), в котором в качестве обучающей выборки может служить выборка аллельных вариантов и аллельных фенотипов одного из ранее изученных сортов картофеля.
Список сортов картофеля, которые могут служить обучающей выборкой и для которых известно генетическоеразнообразие и сортоспецифические аллели и аллельные фенотипы, представлен в табл. 1 (Russet Burbunck, Mondeal, Bintije, Romano, Пушкинский, Чародей, Невский, Петербургский, Лазурит, Яхонт, Эффект, Лорх, Лукьяновский, Раменский, Никулинский, Осень, Скороплодный, Корона, Резерв, Удача, Загадка, Волжанин, Ранняя Роза, Красноярский, Беглицкий, Ильинский, Бронницкий, Вестник, Жуковский).
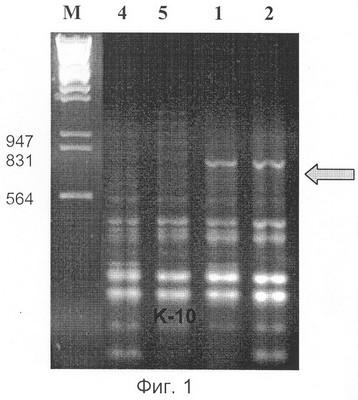
На фиг.1 представлен набор полиморфных ДНК, выявляемых с помощью праймера STU104 у различных сортов картофеля.
На фиг.2 представлен набор полиморфных ДНК, выявляемых с помощью праймера STU005 у различных сортов картофеля.
На фиг.3 представлен набор полиморфных ДНК, выявляемых с помощью праймера STU012 у различных сортов картофеля.
Примеры выполнения предлагаемого способа.
Пример 1. Способ определения сорта картофеля с помощью праймеров STU104F/ STU104R
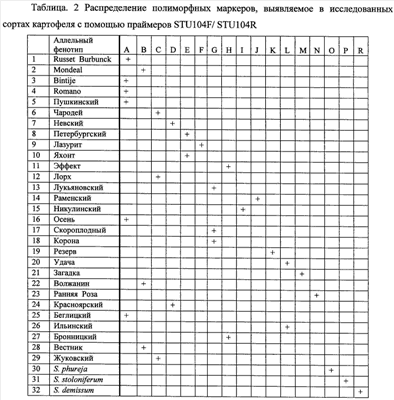
Из растительного материала сортов картофеля выделяют ДНК способом, указанным выше. На каждой из выделенных ДНК осуществляют ПЦР с одной из трех пар предлагаемых праймеров, а именно STU104F/ STU104R. Полученные продукты амплификации визуализируют путем электрофореза в геле, и после окрашивания геля нитратом серебра идентифицируют маркерные аллели различной длины, представляющие аллельные фенотипы сортов картофеля. На фиг.1 представлены полиморфные маркеры для каждого тестированного сорта картофеля, а также образцов, часто используемых в селекции сортов культурного картофеля видов S. stoloniferum, S. demissum и S. phureja.
Каждый из сортов характеризуется определенным набором или сочетанием аллелей, то есть сортоспецифичными аллельными фенотипами, представленными в таблице 2. Это распределение служит эталоном при сравнении и идентификации растений неизвестных сортов картофеля. Путем сравнения распределения полиморфных ДНК разной длины среди исследуемых и эталонных образцов определяют сортовую принадлежность
Пример 2. Способ определения сорта картофеля с помощью праймеров STU005F/STU005R
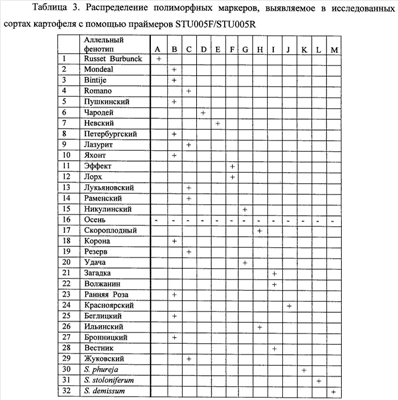
Из растительного материала сортов картофеля выделяют ДНК способом, указанным выше. На каждой из выделенных ДНК осуществляют ПЦР с одной из трех пар предлагаемых праймеров, а именно STU005F/STU005R. Полученные амплификационные продукты визуализируют путем электрофореза в геле, и после окрашивания геля нитратом серебра идентифицируют маркерные аллели различной длины, представляющие аллельные фенотипы сортов картофеля. На фиг.2 представлены полиморфные маркеры для каждого тестированного сорта картофеля, а также образцов часто используемых в селекции сортов культурного картофеля видов S. stoloniferum, S. demissum и S. phureja.
Каждый из сортов характеризуется определенным набором или сочетанием аллелей, то есть сортоспецифичными аллельными фенотипами, представленными в таблице 3, Это распределение служит эталоном при сравнении и идентификации растений неизвестных сортов картофеля. Путем сравнения распределения полиморфных ДНК разной длины среди исследуемых и эталонных образцов определяют сортовую принадлежность.
Пример 3. Способ определения сорта картофеля с помощью праймеров STU012F/STU012R
Из растительного материала сортов картофеля выделяют ДНК способом, указанным выше. На каждой из выделенных ДНК осуществляют ПЦР с одной из трех пар предлагаемых праймеров, а именно STU012F/STU012R. Полученные амплификационные продукты визуализируют путем электрофореза в геле, и после окрашивания геля нитратом серебра идентифицируют маркерные аллели различной длины, представляющие аллельные фенотипы сортов картофеля. На фиг.3 представлены полиморфные маркеры для каждого тестированного сорта картофеля, а также образцов часто используемых в селекции сортов культурного картофеля видов S. stoloniferum, S. demissum и S. phureja.
Каждый из сортов характеризуется определенным набором или сочетанием аллелей, то есть сортоспецифичными аллельными фенотипами, представленными в таблице 4. Это распределение служит эталоном при сравнении и идентификации растений неизвестных сортов картофеля. Путем сравнения распределения полиморфных ДНК разной длины среди исследуемых и эталонных образцов определяют сортовую принадлежность.
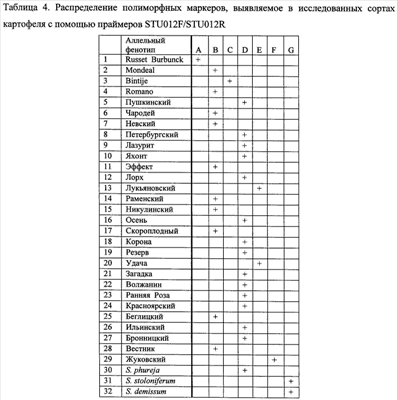
Молекулярно-генетическийпаспорт по всем трем парам праймеров составляется для каждого известного сорта и представлен в таблице 1. Аналогичный паспорт можно составить для любой выборки растений неизвестного происхождения.
Формула изобретения
1. Биологический маркер, содержащий полиморфную ДНК, характеризующий сортовую принадлежность наиболее распространенных и хозяйственно значимых сортов картофеля, и представляющий собой нуклеотидные последовательности из группы трех пар праймеров следующего состава:
|
Пары праймеров |
Первичная последовательность праймеров |
|
STU005 |
F 5'AATTCATGTTTGCGGTACGTC3' |
|
R 5'ATGCAGAAAGATGTCAAAATTGA3' |
|
|
STU012 |
F 5'AACATTACAACACATTAGCA3' |
|
R 5'AACTTATCTGAAACTCTCGT3' |
|
|
STU0104 |
F 5'GGGACATCACAGTCT3' |
|
R 5'GGTGCTCCTATTGGTG3 |
|
2. Набор для определения сортовой принадлежности растений картофеля, содержащий биологический маркер по п.1, реакционную смесь, состоящую из 160 мкМ каждого динуклеотидтрифосфата dNTP, 1.6 мМ MgCl2, 0.3 мкМ каждого праймера из набора, 0.3 единицы фермента Taq-полимеразы, 1· буфера для Taq-полимеразы и эталонные ДНК растения картофеля в количестве 20-50 нг.
3. Способ определения сортовой принадлежности растений картофеля, включающий выделение ДНК из свежего растительного материала, исследование геномной ДНК картофеля с помощью ПЦР, отличающийся тем, что ПЦР-анализ проводят, используя набор по п.2, получают полиморфные маркеры ДНК разного размера, визуализируют эти ДНК при электрофорезе в акриламидном или агарозном геле, затем для типирования выявленных в изученной группе аллелей проводят сравнение генетическогоразнообразия определяемой и контрольной групп образцов амплифицируемой ДНК, и по отсутствию или присутствию в исследуемой группе определенных единичных сортоспецифичных аллельных фенотипов определяют сортовую принадлежность.
4. Способ по п.3, отличающийся тем, что для каждого сорта картофеля составляют генетическийпаспорт, представленный в таблице 1, в которой указано распределение выявленных с помощью трех пар праймеров (STU005, STU012, STU104) полиморфных микросателлитных маркеров, соответствующих локусам (STU005, STU012, STU104), и по которым определяют сорт картофеля; размер амплифицируемых продуктов определяют с помощью маркера молекулярных масс (10 bp Ladder); набор контрольных амплификатов содержит образцы амплифицированной ДНК, характерные для каждого из изученных сортов картофеля.
5. Способ по п.3 или 4, отличающийся тем, что верификацию сортовой принадлежности проверяют с помощью различных вариантов многомерного анализа, например, путем кластерного анализа, дискриминантного анализа, метода главных компонент, или нейронных сетей Кахонена, в котором в качестве обучающей выборки может служить выборка аллелей и аллельных фенотипов одного из набора сортов картофеля.
РИСУНКИ
|
|
ПРИЛОЖЕНИЕ О
Заявка на изобретение: 2007134194
Дифференцирующий и специфический олиго-нуклеотид для идентификации последовательностей ДНК трансгенных растенийв пищевых продуктах, способ идентификации трансгенных продуктов, биочип, комбинация олигонуклеотидов (варианты) и набор для осуществления этого способа
1. Способ идентификации маркеровгенетически модифицированныхрастений,включающий обеспечение образца исследуемойДНК,прямых и обратных праймеров, специфичных кДНКодного или болеемаркеровГМИ, и биочипа, проведение ПЦР в присутствии образцаДНКи специфичных праймеров с зондами, специфичными к указанному одному или болеемаркерам, обработку продуктов ПЦР реакции ферментом, инкубацию ПЦР-продукта с биочипом в условиях гибридизации, определение наличия ГМИ детекцией образованных гибридизационных комплексов на биочипе, отличающийся тем, что биочип содержит дифференцирующие олигонуклеотиды, с последовательностями, представленными в SEQ ID NO:23-33, в качестве специфичных праймеров используют праймеры, последовательность которых выбирают из группы, состоящей из SEQ ID NO:1-22, в качестве ПЦР используют мультипраймерную симметричную ПЦР, продукты которой обрабатывают экзонуклеазой фага лямбда в концентрации от 5 до 20 единиц активности в течение от 30 до 90 мин, где в паре прямого и обратного праймеров, один праймер содержит метку, которую после гибридизацииДНК детектируют, регистрируют и используют для идентификациимаркеровГМИ, причем в качестве отрицательного контроля используют дифференцирующий олигонуклеотид SEQ ID NO:36, частично комплементарный олигонуклеотиду SEQ ID NO:8, а в качестве положительного контроля используют дифференцирующий олигонуклеотид SEQ ID NO:34, которому комплементарен специфический олигонуклеотид SEQ ID NO:35.
2. Способ по п.1, отличающийся тем, что проводят детекцию образцов на наличие в них, по крайней мере, одной ДНК, принадлежащей гену, входящему в переченьмаркеров ГМИ, приведенных в таблице 1.
3. Способ по п.1, отличающийся тем, что образец исследуемой ДНКвыделяют, по крайней мере, из одного образца, содержащего ГМИ, входящего в группу, состоящую из плодов или сокарастений, зерна, муки, или пищевых продуктов, в состав которых входят генетически модифицированные растения.
4. Способ по п.1, отличающийся тем, что определяемую метку выбирают из группы, состоящей из каталитических, лигандных, флуоресцентных или радиоактивных молекул, причем каталитическую молекулу выбирают из группы, включающей гемин, цианкобаламин или флавин, лигандную молекулу выбирают из группы, включающей биотин, диоксигенин или динитробензол, а флуоресцентную молекулу выбирают из группы, включающей FAM, TAMRA, Су3, Су5, Су7, R6G, R110, ROX или JOE.
5. Способ по п.1, отличающийся тем, что детектирование и/или идентификацию ГМИ осуществляют путем сравнительного анализа уровня сигналов исследуемого образца, положительного и отрицательного контролей.
6. Специфический олигонуклеотид в качестве праймера ПЦР для идентификации маркераГМИ в способе идентификации ГМИ по пп.1-5 с последовательностями, представленными от SEQ ID NO:1 по SEQ ID NO:22.
7. Дифференцирующий олигонуклеотид в качестве зонда для идентификации маркераГМИ в способе идентификации ГМИ по пп.1-5 с последовательностями, представленными от SEQ ID NO:23 по SEQ ID NO:33.
8. Биочип для идентификации ГМИ, представляющий собой несущий элемент, характеризующийся тем, что на его поверхности иммобилизированы от 1 до 11 кластеров с дифференцирующими олигонуклеотидами, выбранными из SEQ ID NO:23-33, один или более кластеров, включающих олигонуклеотид положительного контроля, представленный в SEQ ID NO:34, и один или более кластеров, включающих олигонуклеотид отрицательного контроля, представленный в SEQ ID NO:36.
9. Биочип по п.8, отличающийся тем, что несущий элемент чипа выполнен из материалов, входящих в группу, состоящую из: полимеров, стекла, металлов, керамики или их комбинаций.
10. Биочип по п.9, отличающийся тем, что полимеры выбирают из группы, состоящей из: полиметилметакрилата, полибутилметакрилата, поливинилхлорида, поликарбоната, сополимеров метилметакрилата и/или сополимеров бутилметакрилата с другими мономерами, такими как стирол, акрилонитрил.
11. Биочип по п.8, отличающийся тем, что несущий элемент чипа выполнен в виде плоских платформ, пленок с толщиной от 0,05 до 5,0 мм.
12. Биочип по п.8, отличающийся тем, что дополнительно содержит идентификатор выполненный в виде штрих-кода или элемента с магнитной записью.
13. Биочип по п.12, отличающийся тем, что идентификатор биочипа включает код номера партии и/или даты изготовления и информацию о перечне размещенных дифференцирующих олигонуклеотидов.
14. Набор для идентификации ГМИ, включающий:
а) по меньшей мере, один биочип для проведения разовой диагностики, содержащий один или более дифференцирующих олигонуклеотидов, специфичных к идентифицируемым маркерамтрансгенныхрастений, и представленных в SEQ ID NO:23-33, один или более дифференцирующих олигонуклеотидов в качестве положительного контроля, представленных в SEQ ID NO:34, и один или более дифференцирующих олигонуклеотидов в качестве отрицательного контроля, и представленных в SEQ ID NO:36,
б) реагенты, которые выбирают из реагентов для подготовки образца, реагентов для проведения ПЦР, реагентов для проведения гибридизации и их комбинации и фермента экзонуклеазы.
в) специфические олигонуклеотиды в качестве праймеров ПЦР для выявления маркеровГМИ с последовательностями, которые выбирают от SEQ ID NO:1 по SEQ ID NO:22.
15. Комбинация олигонуклеотидов для выявления маркера35S преимущественно для идентификации 35S промотора из вируса мозаики цветной капусты, отличающаяся тем, что включает олигонуклеотиды с последовательностями SEQ ID NO:23, и SEQ ID NO:1, и SEQ ID NO:2.
16. Комбинация олигонуклеотидов для выявления маркераNOS преимущественно для идентификации 3' нетранслируемой области гена нопалинсинтазы, отличающаяся тем, включает олигонуклеотиды с последовательностями SEQ ID NO:24, и SEQ ID NO:3, и SEQ ID NO:4.
17. Комбинация олигонуклеотидов для выявления маркераOCS преимущественно для идентификации 3' нетранслируемой области гена октопинсинтазы, отличающаяся тем, что включает олигонуклеотиды с последовательностями SEQ ID NO:25, и SEQ ID NO:5, и SEQ ID NO:6.
18. Комбинация олигонуклеотидов для выявления маркераNPTII преимущественно для идентификации гена кодирующего неомицин-фосфотрансферазу II, отличающаяся тем, что включает олигонуклеотиды с последовательностями SEQ ID NO:26, и SEQ ID NO:7, и SEQ ID NO:8.
19. Комбинация олигонуклеотидов для выявления маркераGUS преимущественно для идентификации гена кодирущего бета-глюкуронидазу, отличающаяся тем, что включает олигонуклеотиды с последовательностями SEQ ID NO:27, и SEQ ID NO:9, и SEQ ID NO:10.
20. Комбинация олигонуклеотидов для выявления маркераLectin преимущественно для идентификации гена семейства Lel из Glycine max, отличающаяся тем, что включает олигонуклеотиды с последовательностями SEQ ID NO: 28 в качестве зонда и два специфических SEQ ID NO:11 и SEQ ID NO:12.
21. Комбинация олигонуклеотидов для выявления маркераZein преимущественно для идентификации гена семейства Zein из Zea Mays, отличающаяся тем, что включает олигонуклеотиды с последовательностями SEQ ID NO:29, и SEQ ID NO:13, и SEQ ID NO:14.
22. Комбинация олигонуклеотидов для выявления маркераbar преимущественно для идентификации гена из Streptomyces hygroscopicus, кодирущего фосфотрицин-ацетилтрансферазу, отличающаяся тем, что включает олигонуклеотиды с последовательностями SEQ ID NO:30, и SEQ ID NO:15, и SEQ ID NO:16.
23. Комбинация олигонуклеотидов для выявления маркераcry1Ab преимущественно для идентификации гена из Bacillus thuringiensis, кодирующего Bt-токсин, отличающаяся тем, что включает олигонуклеотиды с последовательностями SEQ ID NO:31, и SEQ ID NO:17, и SEQ ID NO:18.
24. Комбинация олигонуклеотидов для выявления маркера EPSPS преимущественно для идентификации гена из Agrobacterium tumefaciens, кодирующего 5-енолпирувилшикимат-3-фосфатсинтазу, отличающаяся тем, что включает олигонуклеотиды с последовательностями SEQ ID NO:32, и SEQ ID NO:19, и SEQ ID NO:20.
25. Комбинация олигонуклеотидов для выявления маркераPatatin преимущественно для идентификации гена pat1 из Solanum tuberosum, кодирующего предшественник пататина, отличающаяся тем, что включает олигонуклеотиды с последовательностями SEQ ID NO:33, и SEQ ID NO:21, и SEQ ID NO:22.
ПРИЛОЖЕНИЕ П
Описание изобретения к патенту: 2294633

ПРИЛОЖЕНИЕ Р
Описание изобретения к патенту: 2413774

ПРИЛОЖЕНИЕ С
Заявка на изобретение: 2008114535
