
MolBiol_sivolob
.pdf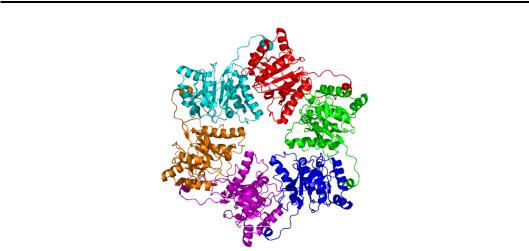
Розділ 9. Реплікація ДНК
Рис. 9.10. Структура реплікативної гелікази (1G8Y)
Інші реплікативні гелікази побудовані за аналогічними принципами. У розплітанні ДНК під час реплікації беруть також участь гелікази протилежного напрямку (3'-5' – транслокація вздовж лідируючого ланцюга), але, на відміну від DnaB, вони не є життєво необхідними. Геліказа DnaB – це невід'ємний елемент реплісоми, через інші структурні модулі вона зв'язана з ДНК-полімеразами та працює в тісній координації з ними. Швидкість розплітання ДНК окремою геліказою дорівнює ~35 нуклеотидів за секунду, швидкість руху окремої ДНК-полімерази вздовж одноланцюгової ДНК – ~1 тис. нуклеотидів за секунду, при їхньому об'єднанні сумісна швидкість реплікації становить ~750 нуклеотидів за секунду: геліказа трохи гальмує полімеразу, остання – штовхає геліказу.
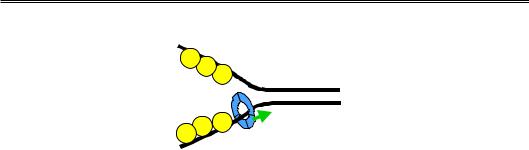
Енергія гідролізу АТР використовується геліказою для здійснення енергетично невигідного процесу розплітання дуплекса. Тобто одноланцюгова ДНК, що виникає в реплікативній вилці внаслідок активності гелікази, має бути тимчасово стабілізована в одноланцюговому вигляді. Цю функцію виконують білки SSB (Single Strand Binding), які мають високу спорідненість до одноланцюгової ДНК (рис. 9.11). SSB білки зв'язуються з полінуклеотидним ланцюгом кооперативно, взаємодіючи не тільки з ДНК, а й між собою. У результаті ланцюг ефективно вкривається білковою “шубою”. SSB білки взаємодіють також з іншими компонентами реплісоми, підсилюючи їхню активність – у тому числі активність ДНК-полімерази.
285
Сиволоб А.В. Молекулярна біологія
3'
ssbssbssb
5'

 3' ssbssb ssb геліказа
3' ssbssb ssb геліказа
5'
Рис. 9.11. Реплікативна вилка, що виникає
як результат дії гелікази та підтримується SSB білками
Синтез ланцюга, що запізнюється
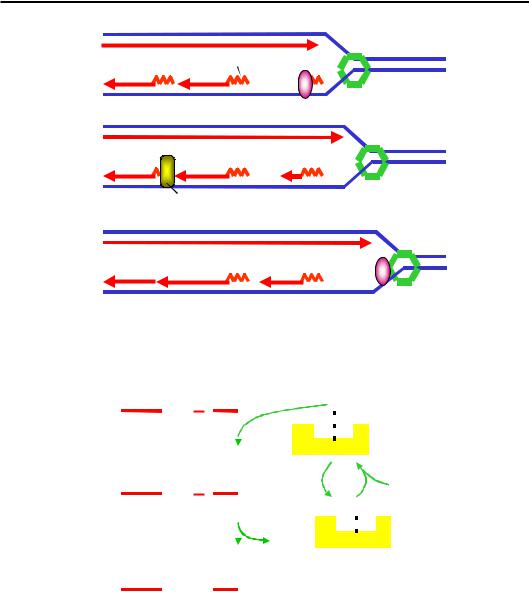
Лідируючий ланцюг при ініціації реплікації, а кожен фрагмент Оказакі під час реплікації, мають розпочинатися з РНК-праймера. Його синтез здійснює праймаза (білок DnaG E. coli). Праймаза рекрутується до основи реплікативної вилки геліказою (їхній комплекс називають праймосомою), робота праймази стимулюється геліказою та SSB білками. Розпочинаючи від основи реплікативної вилки, праймаза, яка має низьку процесивність, синтезує праймер – фрагмент РНК довжиною ~12 нуклеотидів (рис. 9.12). Після цього ДНК-полімераза ІІІ, подовжуючи 3'-кінець праймера, починає синтезувати решту фрагмента Оказакі, а праймаза (та сама чи інша) знову зв'язується з геліказою і згодом розпочинає синтез наступного праймера.
На відміну від ДНК-полімерази, праймаза може з однаковим успіхом включати до праймера як дезоксириботак і рибонуклеотиди. Праймер є фрагментом саме РНК просто тому, що внутрішньоклітинна концентрація рибонуклеозидтрифосфатів є вищою.
Зрозуміло, що РНК-праймер на початку кожного фрагмента Оказакі має бути замінений на відповідну послідовність ДНК. Цю роботу виконує ДНК-полімераза І (рис. 9.12): за рахунок своєї 5'-екзонук- леазної активності полімераза видаляє праймер і одночасно, використовуючи 3'-кінець попереднього фрагмента Оказакі, подовжує його, заповнюючи прогалину.
Після здійснення останньої операції між двома фрагментами Оказакі залишається одноланцюговий розрив (рис. 9.12, 9.13) – ДНК-полімераза не може його зшити, оскільки здатна тільки переносити нуклеотиди з NTP на 3'-кінцеву ОН-групу. Утворення фосфодіефірного зв'язку між сусідніми фрагментами Оказакі, яке потребує джерела енергії, здійснюється в АТР-залежній реакції, яка каталізується лігазою (ligase).
286
Розділ 9. Реплікація ДНК
Лідируючий  ланцюг
ланцюг
Фрагмент Праймер Оказакі
Геліказа
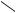 Праймаза
Праймаза
DNAP I
Рис. 9.12. Робота праймази та ДНК-полімерази І під час руху реплікативної вилки
3' |
|
|
|
|
|
|
5' |
|
|
|
|
|
|
|
|
|
|
|
|
|
|
O |
|
|
|
|
|
|
|
|
|
|
|||||
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
||||
5' |
OH |
O– |
|
|
|
3' |
|
|
AMP |
|||||||||
P |
||||||||||||||||||
|
|
|
|
|
|
|
|
|
|
NH |
|
|
||||||
|
|
|
|
|
|
|
|
|||||||||||
|
|
|
|
O– |
|
|
|
Lys |
|
|
|
|
|
|||||
3' |
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
5' |
|
|
|
|
|
|
|
|
|
|
||
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|||
|
|
|
O |
|
|
|
|
|
|
|
|
|
ATP |
|||||
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|||||
5' |
OH |
O– |
|
|
|
3' |
|
|
|
|
|
|
|
|
|
|||
P |
NH2 |
|||||||||||||||||
|
|
|
|
|
|
|
||||||||||||
|
|
|
|
|
|
AMP |
|
|
|
|
|
|
|
|
||||
|
|
|
|
|
|
|||||||||||||
|
|
|
|
|
|
|
|
|
|
|
|
|
Lys |
|
|
|||
|
|
|
|
|
|
|
|
AMP |
|
|
|
|
|
|
|
|
||
3' |
|
|
|
|
|
|
5' |
|
|
|
|
|
|
|
|
|||
|
|
|
|
|
|
|
|
Лігаза |
||||||||||
|
|
|
O |
|
|
|||||||||||||
|
|
|
|
|
|
|
||||||||||||
5' |
O |
|
|
|
|
|
3' |
|
|
|
|
|
|
|
|
|
|
|
|
|
P |
|
|
|
|
|
|
|
|
|
|
||||||
|
|
|
|
|
|
|
|
|
|
|
|
|||||||
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
O– |
|
|
|
|
|
|
|
|
|
|
|
|||
Рис. 9.13. Схема роботи лігази
Схему роботи лігази показано на рис. 9.13. До активного центру ферменту, ковалентно приєднуючись до аміногрупи Lys, переноситься АМР з молекули АТР. Далі АМР приєднується до 5'-кінцевого фос-
287

Сиволоб А.В. Молекулярна біологія
фату в місці розриву (своєрідна активація, що нагадує активацію амінокислоти АРСазою, див. розділ 8). Видалення АМР супроводжується утворенням фосфодіефірного зв'язку.
Голофермент ДНК-полімерази
Голофермент ДНК-полімерази, який є основою реплісоми, містить дві молекули ДНК-полімерази ІІІ (по три субодиниці в кожній – кор-фер- мент), дві копії τ-білка, γ-комплекс (п'ять субодиниць) та два β-білка, зв'язані з кожною полімеразою. Призначення компонентів голоферменту – утримання разом і забезпечення координації роботи компонентів реплісоми, підвищення процесивності ДНК-полімерази.
Проблема процесивності вирішується β-білком, який утворює так званий обруч (зажим), що ковзає (sliding clamp). Дві однакові субодиниці обруча, міцно взаємодіючи за принципом “голова до хвоста”, утворюють циркулярну тороподібну структуру з отвором приблизно 35 Å у діаметрі (рис. 9.14). Усередині такого кільця легко розміщується подвійна спіраль ДНК, яка практично не взаємодіє з β-білком. Кільце може вільно рухатись уздовж ДНК, але не може дисоціювати. С-кінцевий гідрофобний хвіст ДНК-полімерази ІІІ довжиною 11 амінокислот взаємодіє з гідрофобним карманом обруча. У результаті формується своєрідний поводок, що допускає певну свободу рухів полімерази, але міцно утримує її на ДНК.
Рис. 9.14. Обруч, що ковзає, – бактеріальний β-білок (2POL)
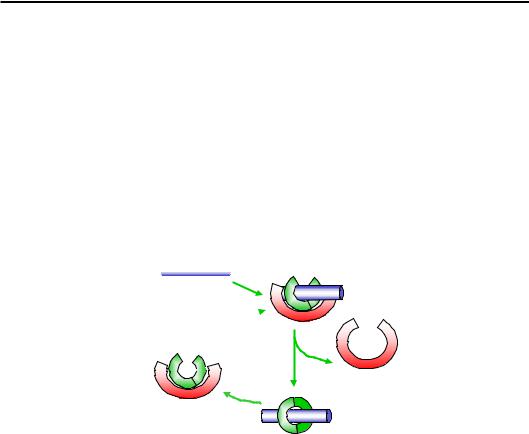
Оскільки обруч – білкове кільце, що оточує ДНК, його завантаження на подвійну спіраль виконується АТР-залежним шляхом, ця функція належить γ-комплексу. Відбувається завантаження на гібридній
288
Розділ 9. Реплікація ДНК
подвійній спіралі РНК-ДНК відразу після закінчення синтезу чергового праймера. γ-Комплекс, що складається з п'яти субодиниць та має U-подібну структуру (рис. 9.15), зв'язує АТР і змінює свою конформацію таким чином, що взаємодіє з обручем, порушуючи його цілісність – розмикаючи кільце. Далі γ-комплекс впізнає дуплекс, що утворений між праймером та матрицею, забезпечуючи посадку відкритого кільця на цю подвійну спіраль. Взаємодія з дуплексом індукує АТРазну активність γ-комплексу. У результаті гідролізу АТР γ-комплекс втрачає спорідненість до β-білка, що призводить до замикання обруча. Далі γ-комплекс знову зв'язує АТР, проте обруч має вищу спорідненість до ДНК-полімерази, ніж до γ-комплексу: обруч залишається на ДНК і взаємодіє з полімеразою та забезпечує її процесивність.
 ДНК
ДНК
обруч 
+
ADP
γ-комплекс•АТР
Рис. 9.15. Схема АТР-залежного завантаження обруча, що ковзає, γ-комплексом
Головну структурну роль в організації реплісоми виконує τ-білок, котрий взаємодіє з ДНК-полімеразою, геліказою, праймазою та утримує на собі γ-комплекс. Численні взаємодії τ-білка забезпечують функціональну координацію між елементами реплісоми.
Дві молекули τ-білка взаємодіють між собою і кожна утримує корфермент ДНК-полімерази ІІІ, кожна з яких, у свою чергу, зв'язана з обручем (рис. 9.16). Одна з полімераз здійснює синтез лідируючого ланцюга, інша – фрагмента Оказакі. Обидві полімерази орієнтовані в реплісомі однаково. Оскільки вони при цьому ведуть синтез на антипаралельних ланцюгах, той, що запізнюється, утворює реверсну петлю: конфігурація реплікативної вилки нагадує тромбон.
289

Сиволоб А.В. Молекулярна біологія
На рис. 9.16 зображено момент, коли праймаза почала синтез чергового праймера, а γ-комплекс – напоготові завантажити черговий обруч. Коли полімераза, яка працює на ланцюзі, що запізнюється, завершить синтез фрагмента Оказакі (зустріне попередній фрагмент), вона втратить свій зв'язок з обручем, але збереже взаємодію з τ-білком. Втрата спорідненості полімерази до обруча визначається одноланцюговим розривом між фрагментами Оказакі. Новий обруч буде при цьому вже завантажений на черговий праймер, полімераза зв'яжеться з ним і розпочне синтез нового фрагмента Оказакі. Обруч, який щойно втратив зв'язок із полімеразою, провзаємодіє з γ-комплексом – останній працює також і як “розвантажувач” обруча (див. рис. 9.15), знімаючи його з ДНК і забезпечуючи пізніше його посадку на праймер.
|
Геліказа |
|
t |
Праймаза |
|
|
|
|
? |
ß |
|
|
DNAP III |
5' |
|
|
Фрагмент
Оказакі
Лідуючий ланцюг
3'
Рис. 9.16. Схема організації реплісоми
Топологічні проблеми, пов'язані з реплікацією
Ще одним невід'ємним елементом системи реплікації є ДНК-топоізо- мерази (див. розділ 3). Рух реплікативної вилки з одночасним руйнуванням подвійної спіралі супроводжується прокруткою спіралі, що створює позитивні надспіральні витки – еластичні напруження попереду від вилки (див. рис. 3.24). Це стосується як еукаріотичних систем, де петлі ДНК зчеплені своїми кінцями з ядерним матриксом, так і прокаріотич-
290
Розділ 9. Реплікація ДНК
них циркулярних хромосом: зустрічний рух двох реплікативних вилок (рис. 9.3) створює позитивні напруження на ділянці, яка ще не є реплікованою. Еластична напруга має час від часу зніматися, інакше врештірешт вона повністю зупинить рух реплісоми. Відповідно, процес реплікації потребує допоміжної дії топоізомераз, основна функція яких як раз і полягає в релаксації надспіралізованої ДНК: штучне вимикання активності топоізомераз повністю блокує реплікацію.
В еукаріотичній клітині завдання релаксації можуть виконувати як топоізомерази І, так і топоізомерази ІІ. Прокаріотична топоізомераза І (топоізомераза Іа) не підходить для виконання цієї ролі, оскільки здатна знімати тільки негативну надспіралізацію. Релаксація позитивної надспіралізації при реплікації у бактерій забезпечується гіразою – ферментом, що відноситься до класу топоізомераз ІІ. Гіраза вносить негативну надспіралізацію в циркулярну ДНК, що й компенсує позитивні напруження.
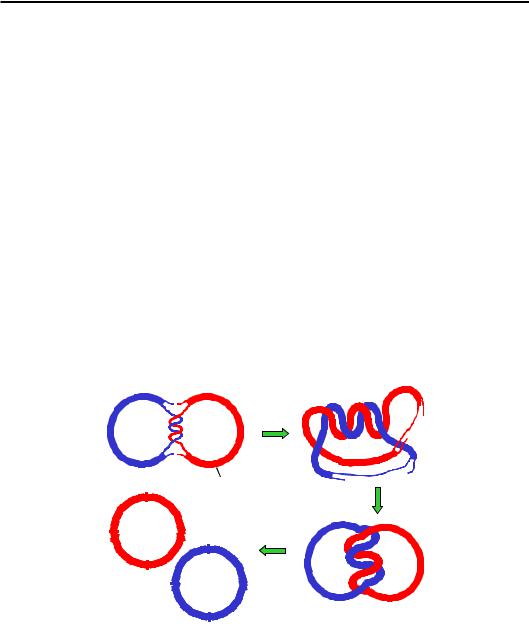
Ще одна топологічна проблема виникає при закінченні процесу реплікації в циркулярному репліконі. Коли дві реплікативні вилки зустрічаються, вони зупиняються за кілька витків подвійної спіралі одна від одної: еластичну напругу на маленькій ділянці зняти вже неможливо (рис. 9.17).
Дволанцюгова
ДНК
Катенан
Рис. 9.17. Термінація реплікації у циркулярному репліконі
Відбувається руйнування короткої подвійної спіралі, яка не піддалася реплікації, але це неможливо зробити без відповідного перекручення двох дочірніх кільцевих молекул ДНК десь у іншому місці. Одноланцю-
291

Сиволоб А.В. Молекулярна біологія
гові прогалини далі заповнюються ДНК-полімеразами. Але після цього дві остаточно синтезовані циркулярні молекули так і залишаються топологічно зчепленими одна з одною – утворюється так званий катенан (рис. 9.17). Таке саме перекручення двох дочірніх молекул ДНК реалізується при закінченні реплікації на двох сусідніх еукаріотичних репліконах (див. рис. 9.2).
Зрозуміло, що декатенація – розділення двох молекул – є неможливим без дволанцюгового розриву в одній із них. За цю операцію також відповідають топоізомерази класу ІІ (еукаріотична топоізомераза ІІ, яка зв'язана з ядерним матриксом під час реплікації, і гомологічна до неї топоізомераза ІV у прокаріотів), що роблять тимчасовий дволанцюговий розріз.
Ініціація реплікації у бактерій
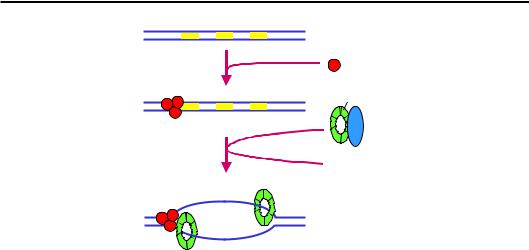
Сайт ініціації реплікації E. coli – ориджин oriC – є ділянкою довжиною 245 пар основ, яка містить чотири сайти по дев’ять пар основ кожний, що мають спорідненість до білка DnaA, а також кілька АТ-збагачених ділянок по 13 пар основ зі зниженою стабільністю подвійної спіралі. Передумовою ініціації реплікації є створення за рахунок активності гірази певного рівня негативної надспіралізації в циркулярній хромосомі. По-перше, негативна надспіралізація сприяє локальному руйнуванню подвійної спіралі в межах ориджина. По-друге, оскільки введення надспіралізації є можливим тільки за умови цілісності полінуклеотидних ланцюгів (хоча б один одноланцюговий розрив знімає будь-яку еластичну напругу), це є ефективною перевіркою готовності матриці (по всій довжині) до реплікації.
На першій стадії ініціації кілька копій DnaA (у комплексі з АТР) зв'язуються з ориджином (рис. 9.18) та індукують АТР-залежне локальне плавлення подвійної спіралі. Білок DnaС завантажує на одноланцюгові ділянки геліказу DnaВ, яка, у свою чергу, рекрутує праймазу та інші елементи реплісоми. Праймаза здійснює синтез праймерів на лідируючих ланцюгах і замінюється на ДНК-полімеразу ІІІ: починається рух двох реплікативних вилок у протилежних напрямках. Напрямок руху задається напрямком транслокації гелікази по ланцюгу, що запізнюється. Присутність гелікази саме на цьому ланцюзі забезпечує рекрутування до нього праймази під час елонгації реплікації на початку синтезу кожного фрагмента Оказакі.
292
Розділ 9. Реплікація ДНК
5' |
3' |
3' |
5' |
5' |
3' |
3' |
5' |
5' |
3' |
3' |
5' |
 Dna A
Dna A
Dna B

 Dna C
Dna C
Рис. 9.18. Ініціація реплікації в E. coli. Жовтим позначено 13-мерні ділянки ДНК зі зниженою стабільністю
На діаметрально протилежному щодо oriC боці циркулярної хромосоми розташовано 6 ділянок по 22 пари основ – сайтів термінації реплікації (Ter A, B, C, D, E, F), які утворюють комплекс із білком Tus (Terminus utilization substance). На сайтах термінації, зв'язаних із цим білком, реплікативні вилки зупиняються. Зона між ними піддається далі локальному плавленню і після цього відбувається добудова прогалин і декатенація (див. рис. 9.17).
Особливості еукаріотичної системи реплікації
Загальні принципи й механізми реплікації є спільними для прота еукаріотів, розрізняючись у деталях:
•Еукаріотична реплікативна вилка містить дві ДНК-поліме- рази, процесивність яких забезпечується ковзним циркулярним обручем. Різниця лише в тому, що обруч (PCNA – Proliferating Cell Nuclear Antigene) складається з трьох субодиниць. Крім забезпечення процесивності ДНК-полімерази, PCNA взаємодіє ще з багатьма білками, залученими до виконання різноманітних функцій при реплікації.
•Одна з полімераз веде синтез лідируючого ланцюга, інша синтезує фрагменти Оказакі довжиною 200–400 пар основ. РНК-прай- мер на початку кожного фрагмента Оказакі видаляється спе-
293

Сиволоб А.В. Молекулярна біологія
цифічною нуклеазою. Прогалина потім заповнюється ДНК-полі- меразами, одноланцюговий розрив зшивається лігазою.
•Основним структурним модулем реплісоми є мультибілковий комплекс RF-C (Replication Factor C), що виконує функції, аналогічні до бактеріального τ-білка та γ-комплексу.
•Вважається, що основною реплікативною геліказою еукаріотів є моногексамерний білок МСМ (MiniChromosome Maintenance). Одноланцюговий стан ДНК у реплікативній вилці підтримується білками RPA (Replication Protein A – аналог білків SSB).
Еукаріотичні ДНК-полімерази
П'ять основних типів еукаріотичних ДНК-полімераз прийнято позначати грецькими літерами.
Полімераза α складається з чотирьох субодиниць: одна виконує структурну роль, друга має ДНК-полімеразну активність, ще дві субодиниці відповідають за активність РНК-полімеразну. Відповідно, полімераза α виконує роль праймази: саме вона синтезує короткий РНК-праймер (6–10 нуклеотидів), який далі трохи подовжує як ДНК. Полімераза α характеризується низькою процесивністю і, на відміну від інших ДНК-полімераз, не має екзонуклеазної активності.
Полімераза β – мономерний білок, основна роль якого полягає в заповненні однонуклеотидних прогалин при ексцизійній репарації основ (див. нижче).
Полімераза γ – реплікативна ДНК-полімераза мітохондрій. Складається з двох субодиниць, крім ДНК-полімеразної має обидві (3'- та 5'-) екзонуклеазні активності.
Полімерази δ та ε – основні реплікативні ДНК-полімерази еукаріотів. Складаються з чотирьох (ε) або трьох–п’яти в різних видів (δ) субодиниць. Обидві полімерази можуть працювати в реплікативній вилці (у комплексі з PCNA та RF-C) на обох ланцюгах (лідируючому та тому, який запізнюється). Функціональну різницю між двома полімеразами остаточно не з'ясовано. Вважається, що полімераза δ є суто ДНК-синтезуючою машиною, тоді як полімераза ε використовується також як “сенсор” пошкоджень ДНК, сприяючи активації репараційних процесів. Крім реплікативного синтезу ДНК, полімерази δ і ε здійснюють заповнення довгих багатонуклеотидних прогалин як при з'єднанні сусідніх фрагментів Оказакі, так і при репараційних процесах, які розглядатимуться далі.
294
