
- •Лекція № 5. Транскрипція: прокаріоти
- •Структура рнк-полімерази
- •Ініціація транскрипції
- •Елонгація транскрипції
- •Термінація транскрипції
- •Регуляція транскрипції
- •Лактозний оперон
- •Антитермінація
- •Атенюація
- •Регуляція транскрипції бактеріофага λ
- •Неспецифічна регуляція загального рівня транскрипції
- •Контрольні запитання
- •Рекомендована література
Елонгація транскрипції
При елонгації транскрипції РНК-полімераза рухається вздовж матриці разом із розплавленими 12 – 14 парами основ у транскрипційному міхурі (рис. 4, 8). При цьому на кожному кроці полімерази одна пара основ руйнується попереду міхура й одна відновлюється позаду. Розділення подвійної спіралі ДНК перед міхуром полегшується так званою G-петлею РНК-полімерази (Gtrigger loop, рис. 8).У зоні міхура з полімеразою завжди зв’язаний ДНК-РНК-гібрид довжиною вісім – дев’ять пар основ: вісім – дев’ять нуклеотидів на 3′-кінці РНК залучені до утворення подвійної спіралі з матричним ланцюгом, наступні п’ять – шість нуклеотидів локалізовані в каналі виходу РНК, решта нуклеотидів на 5′-кінці виходять за межі полімеразного комплексу. Поділу гібрида, а отже, і відновленню ДНК-дуплекса, сприяє L-петля (lid-loop, рис. 8).

Рис. 8. Вихідна конфігурація системи транскрипціїна початку елонгаційного циклу.
Наявність ДНК-РНК-гібрида при транскрипції є дуже важливою обставиною. Цей гібрид існує завдяки специфічним взаємодіям, які реалізують з ним певні елементи полімерази. Висока спорідненість РНК-полімерази до гібрида забезпечує високу процесивність ферменту – здатність працювати на матриці без дисоціації. Інший фактор підвищення процесивності – взаємодія транскрипту з каналом виходу РНК. Дисоціація полімерази, яка була можливою під час синтезу первинного транскрипту, поки гібрид був недостатньо довгим (абортивна ініціація, див. вище), під час елонгації стає малоймовірною подією. На рис. 8 схематично зображено вихідну конфігурацію системи напочатку кожного елонгаційного циклу перед зв’язуванням чергового NTP.
Сайт зв’язування формується 3′-кінцевим нуклеотидом РНК, неспареним нуклеотидом матричного ланцюга ДНК (позначеним як і +1), активним центром полімерази та його оточенням, зокрема F-спіраллю (рис. 3, 8). ОН-група на 3′-кінці РНК фіксується в активному центрі поряд з іоном Mg2+ (рис. 3, 9). NTP потрапляє до сайту зв’язування через вторинний канал разом з другим іоном Mg2+.
Перший етап елонгаційного циклу – відбір і зв'язування нуклеозидтрифосфату, яке контролюється матрицею (нуклеотидом і +1).Спорідненість усіх чотирьох типів NTP до РНК-полімерази є досить невисокою, що дозволяє їм здійснювати швидке зв’язування / дисоціацію. Реалізація комплементарних взаємодій NTP із відповідним нуклеотидом матриці (поява нового ліганду в сайті зв’язування – комплементарної нуклеотидної пари) викликає конформаційну зміну із замиканням NTP та жорсткою фіксацією субстратів у активному центрі (рис. 9, 10).
Другий етап – хімічна реакція приєднання чергового нуклеотиду до 3′-кінця транскрипту (рис. 9). За умови реалізації комплементарних взаємодій між NTP і нуклеотидом у складі матриці, NTP жорстко фіксується в активному центрі в певній напруженій реакційноздатній конформації, яка може розглядатися як інтермедіат реакції.
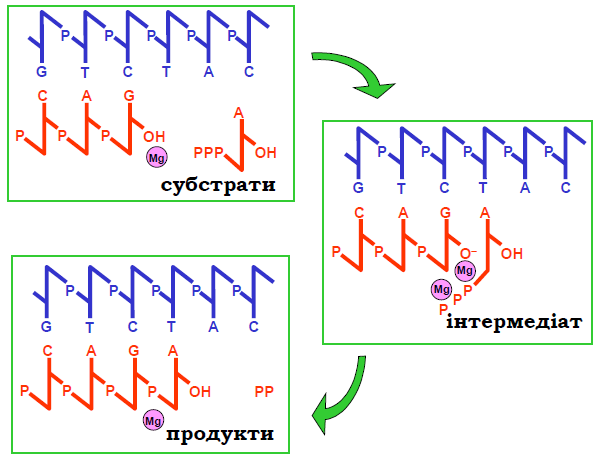
Рис. 9. Хімічна схема процесу приєднання нуклеотиду.
Найважливішу роль у фіксації інтермедіату відіграють два іони Mg2+ (один утримується в активному центрі завжди, інший, який потрапив туди разом з NTP – тимчасово, через взаємодію з двома із трьох залишків Asp активного центру). Перший іон взаємодіє з α-фосфатом NTP і 3′ ОН-групою, забезпечуючи зокрема її іонізацію (рис. 9). Другий іон стабілізує певну напружену конформацію всіх трьох фосфатних залишків. Спорідненість активного центру до інтермедіату і жорстка фіксація субстратів забезпечує зниження активаційного бар’єра реакції, тобто її каталіз.
Результатом реакції є подовжений на один нуклеотид 3′-кінець РНК і відщеплений пірофосфат. Крім того, унаслідок реакції змінюється розташування ліганду (3′-кінця) відносно активного центру (рис. 9; 10).
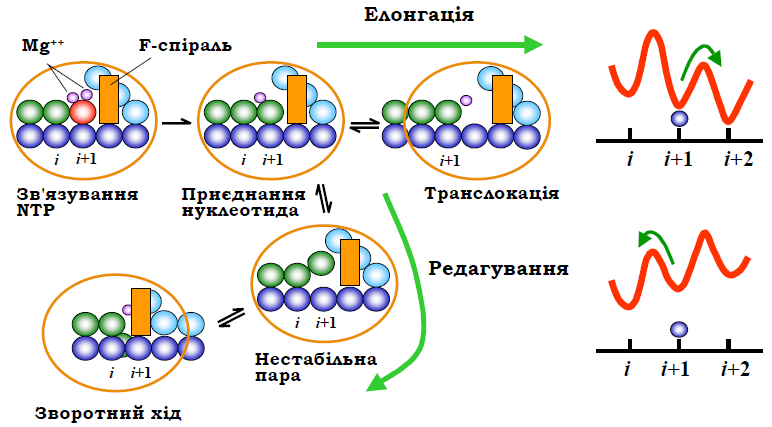
Рис. 10. Послідовність подій в елонгаційному циклі (збільшений фрагмент схеми на рис. 8). Показано також альтернативний шлях при редагуванні помилок. Праворуч: енергетичні профілі, що відповідають руху полімерази вперед відносно матриці при елонгації (угорі) і назад при редагуванні (унизу).
Зміна ліганду та дисоціація пірофосфату спричиняє "розмикання" структури в зоні активного центру, яке дозволяє рух полімерази.
Відбувається третій етап – транслокація у напрямку оптимального розташування подовженого 3′-кінця відносно активного центру (рис. 10). Рух полімерази при транслокації відбувається як одномірна дифузія вздовж матриці за рахунок енергії теплових флуктуацій, тобто можливим є рух як "уперед", так і "назад". Пересування полімерази вперед супроводжується руйнуванням однієї нуклеотидної пари у складі РНК-ДНК гібрида та відновленням однієї пари ДНК позаду від транскрипційного міхура. Одночасно руйнується одна пара ДНК-попереду від міхура. Якщо взяти до уваги, що на попередньому етапів наслідок хімічної реакції відбулося утворення пари в гібриді, то маємо практично ізоенергетичний процес – дві нуклеотидні пари утворюються замість двох зруйнованих. Аналогічно, пересування полімерази назад буде супроводжуватись руйнуванням пари в гібриді на 3′-кінці РНК (3′-кінцевий нуклеотид при цьому опиняється у вторинному каналі) і пари ДНК позаду від міхура з одночасним утворенням пари ДНК попереду та гібрида позаду.
Різниця між кінцевими результатами двох рухів – в енергії взаємодій РНК-полімерази з нуклеїновими кислотами. У вихідному претранслокаційному стані (положення і + 1, рис. 10) реалізуються певні нуклео-протеїнові взаємодії, що зумовлює знаходження системи в локальному мінімумі вільної енергії. Із двох інших локальних мінімумів, що відповідають положенням і та і + 2, другий є більш глибоким, оскільки для нього реалізуються найсприятливіші взаємодії між активним центром та 3′-кінцем РНК. Максимуми енергії (бар’єри) між мінімумами на рис. 10 відповідають проміжним станам при пересуванні полімерази (часткове порушення взаємодій, конформаційні зміни полімерази). Ці максимуми є досить невисокими, тобто легко долаються зарахунок теплової енергії. Отже, полімераза спонтанно переміщується вперед, і система опиняється в більш глибокому мінімумі вільної енергії(рис. 10), звідки починається наступний елонгаційний цикл.
Якщо напрямок переміщення полімерази визначається положенням мінімумів на шкалі вільної енергії, швидкість переміщення залежить від висоти максимумів вільної енергії: чим нижчим є максимум, тим легше він долається. Висота максимумів залежить від послідовності пар основ у складі гібрида, а також може змінюватись під дією додаткових факторів (фактори елонгації, шпильки в мРНК тощо). Під час елонгації здійснюється також редагування – помилок. Забезпечити стовідсоткову точність операцій з такими невеликими молекулами, як нуклеотиди, практично неможливо. Зокрема, джерелом (одним із основних) помилкового приєднання нуклеотидів під час транскрипції є таутомерія азотистих основ. Спонтанні перебудови електронних систем гетероциклів приводять до того, що кожна основа існує у вигляді двох таутомерних форм: аміно- чи іміноформи для A, C; енольної чи кетоформи для G, U, T (рис. 11). Рівновага зсунута в бік аміно- та кетоформ, які присутні у складі подвійних спіралей і для яких реалізуються правила комплементарності AT, GC. Але спарювання основ підпорядковується іншим правилам для мінорних таутомерних форм: наприклад, іміноформа А та аміноформа С утворюють між собою два водневі зв’язки, що може відбутися під час упізнання матриці нуклеозидтрифосфатом. Тоді після хімічної реакції, у результаті швидкого повернення до мажорної таутомерноїформи, у складі ДНК-РНК гібрида на 3′-кінці транскрипту залишиться некомплементарна пара нуклеотидів.
Нестабільність гібрида на 3′-кінці у випадку, коли щойно приєднаний нуклеотид є помилковим, приводить до зміни енергетичного балансу між різними позиціями РНК-полімерази відносно матриці (рис. 10). Найсприятливішою (такою, що відповідає найнижчому мінімуму енергії) тепер є позиція і, оскільки при цьому гібрид стає стабільним по всій довжині. Відповідно, відбувається зворотний рух полімерази з виходом 3′-кінця транскрипту у вторинний канал. При цьому ніщо більше не фіксує 3′-кінець транскрипту, і всі позиції позаду від і (і, і – 1, і – 2, ...) є майже ізоенергетичними (варіації залежать від стабільності гібрида, тобто від послідовності нуклеотидів). Отже, зворотний рух відбувається на невизначену випадкову відстань.

Рис. 11. Таутомерні форми азотистих основ.
Коли 3′-кінцевий фрагмент транскрипту опиняється у вторинному каналі, з полімеразою в тому ж вторинному каналі взаємодіє особливий фактор елонгації транскрипції, який можна назвати фактором редагування, і який існує у двох варіантах: GreA та GreB (еукаріотичним аналогом цих факторів є фактор TFIIS). Gre-фактори, через конформаційні зміни РНК-полімерази, індукують нуклеазну активність її активного центру. У результаті відбувається відщеплення 3′-кінцевого фрагмента транскрипту і в активному центрі залишається новий 3′-кінець (зі стабільною нуклеотидною парою гібрида), який використовується для відновлення елонгаційного процесу. Загальна точність процесу транскрипції дорівнює приблизно 10–6 (частота помилково включених нуклеотидів, що залишаються у складі РНК).
З викладеного видно, що РНК-полімераза є одним із прикладів молекулярної машини. Конформаційна рухливість полімерази забезпечує їй можливість існувати в кількох структурних станах. Ці стани мають різну спорідненість до певних лігандів (NTP, пари основ у складі РНК-ДНК гібрида, фактори елонгації). Взаємодії з лігандами фіксують певні стани. Хімічні реакції, які каталізуються машиною, приводять до заміни лігандів, а відповідно – і до переходу в інший структурний стан. Рушійною силою для переміщення полімерази є тепловий рух: структурні блоки полімеразного комплексу рухаються відповідно до конструкції машини; зв’язування лігандів та їхня заміна внаслідок реакцій каталізують ці рухи в певних напрямах. Результатом структурних перебудо в є переміщення полімерази вздовж матриці з одночасним синтезом транскрипту із середньою швидкістю 40 нуклеотидів/с.
