
Лаб 2 / Лб2
.docМІНІСТЕРСТВО ОСВІТИ ТА НАУКИ УКРАЇНИ
ХАРКІВСЬКИЙ НАЦІОНАЛЬНИЙ УНІВЕРСИТЕТ РАДІОЕЛЕКТРОНІКИ
Кафедра СТ
Звіт
про виконання лабораторної роботи № 2
з дисципліни Інтелектуальна обробка даних в розподілених інформаційних середовищах
на тему “ Аналіз методів на основі самонавчання. Кластеризація даних”
Виконав: ст. гр. СПРм-19-1 Мизніков Р.І. |
Перевірила: Перова І. Г. |
Харків 2020
АНАЛИЗ МЕТОДОВ НА ОСНОВЕ САМООБУЧЕНИЯ. КЛАСТЕРИЗАЦИЯ ДАННЫХ
Цель работы: получение навыков работы с методами кластеризации данных.
Ход работы:
1. Загрузить в DataFrame результаты 1-й лабораторной работы.
import numpy as np
import math
import copy as copy
import pandas as pd
import matplotlib.pyplot as plt
from sklearn.preprocessing import StandardScaler
from sklearn.cluster import KMeans
from sklearn import metrics
from sklearn.decomposition import PCA
from seaborn import scatterplot as scatter
from sklearn.manifold import TSNE
# Check if value is nan
def isNa(value):
if isinstance(value, int) or isinstance(value, float):
if math.isnan(value) and value != 0:
return True
return False
# Drop maxNaPercents percents of nan values in DataFrame
def dropNaRowByPercent (df, maxNaPercents):
for y, colObject in df.iteritems():
countOfNa = 0;
countOfRows = len(colObject);
for key in colObject:
if isNa(key):
countOfNa += 1
percentNaInRow = (countOfNa / countOfRows) * 100;
if percentNaInRow > maxNaPercents:
#print('deleting', colObject.name)
df = df.drop(colObject.name, axis=1)
return df
# Generate value instead nan
def generateValue(naPosX, naPosY, rowCount):
distances = []
for x, naFreeRow in df_dropNa.iterrows():
res = 0.0000000000001 #divide zero
for y, value in df_res.iloc[naPosX].iteritems():
if (isNa(value) != True) and (isinstance(value, int) or isinstance(value, float)):
res += abs(naFreeRow[y] - value)
distances.append(res/rowCount)
print(distances)
inverseDistancesSum = 0
for distance in distances:
inverseDistancesSum += 1/distance
affiliationLevels = []
for distance in distances:
affiliationLevels.append((1/distance)/inverseDistancesSum)
naValue = 0
iterator = 0
for x, value in df_dropNa[naPosY].iteritems():
naValue += value * affiliationLevels[iterator]
iterator += 1
return naValue
2. Разделить данные и диагноз с использованием метода .loc () и .values (). Провести нормирование или стандартизации данных с использованием метода StandardScaler () или MinMaxScaler () из библиотеки sklearn.
features = resultData.columns[1:].values.tolist()
x = resultData.loc[:, features].values
y = resultData.loc[:,['Діагноз']].values
from sklearn.preprocessing import StandardScaler
x = StandardScaler().fit_transform(x)
3. Провести кластеризацию даных с помощью метода Kmeans. Оценить качество кластеризации с использованием методов библиотеки klearn metrics.classification_report() и metrics.confusion_matrix() для трёх диагнозов одновременно.
mainData = resultData.loc[:].drop(columns=['№иб']).drop(columns=['Діагноз']).values
diagnoses = resultData.loc[:,['Діагноз']].replace('ХОЗЛ', 2).replace('БА', 1).replace('Пневмонія', 0).values
x_stan = StandardScaler().fit_transform(mainData)
kmeans = KMeans(n_clusters = 3, init = 'k-means++', random_state = 42)
y_kmeans = kmeans.fit_predict(x_stan)
print(y_kmeans)
expected = diagnoses
predicted = y_kmeans
print(metrics.classification_report(expected, predicted))
print(metrics.confusion_matrix(expected, predicted))
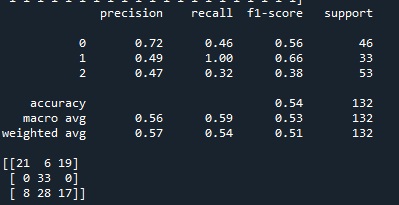
Рисунок 1 — метрики результатов кластеризации методом Kmeans
4. Провести визуализацию даных при помощи метода PCA() на двух радом расположенных рисунках: левый — даные, размеченные согласно значения диагноза; правый — данные, размеченные согласно работе метода Kmeans.
pca = PCA(n_components=2)
pca_diagnosis = pca.fit_transform(x_stan)
principalDf = pd.DataFrame(data = pca_diagnosis [:, 0:2], columns = ['principal component 1', 'principal component 2'])
pcaDF = pd.concat([principalDf, pd.DataFrame(data = diagnoses)], axis=1)
centers = kmeans.cluster_centers_
f, axes = plt.subplots(1, 2, figsize=(15,7))
scatter(pcaDF.loc[:, 'principal component 1'], pcaDF.loc[:, 'principal component 2'], ax=axes[0], hue=diagnoses[:,0])
scatter(pcaDF.loc[:, 'principal component 1'], pcaDF.loc[:, 'principal component 2'], ax=axes[1], hue=y_kmeans)
scatter(centers[:,0], centers[:,1], ax=axes[1], marker='s',s=200)
plt.show()
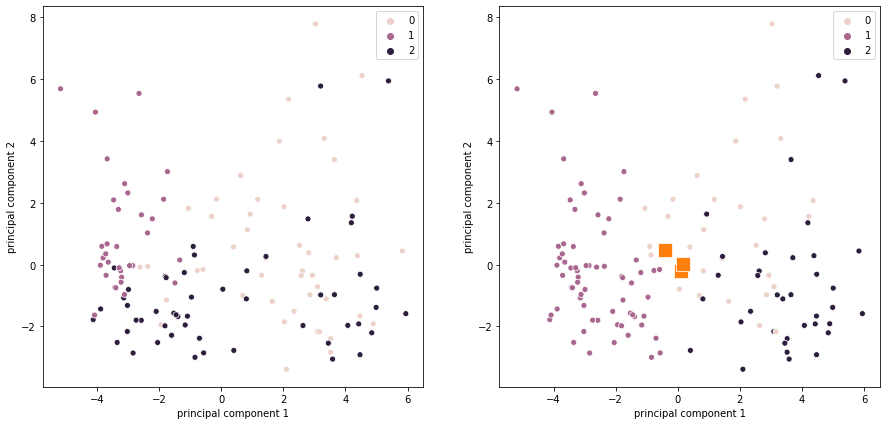
Рисунок 2 — Результат визуализации PCA по диагнозу(слева) и по результатам KMeans(справа)
5. Провести визуализацию даных при помози метода TSNE() на двух расположенных рядом рисунках: левый — даные, размеченные согласно значения диагноза; правый — данные, размеченные согласно работе метода Kmeans. Подобрать значения параметров метода TSNE() для лучшей визуализации. Сделать выводы.
#TSNE
diagnose + kmeans
#%%
tsne
= TSNE (n_components = 2, perplexity = 18, n_iter=1000, random_state
= 43, learning_rate = 100)
x_2D
= tsne.fit_transform(x_stan)
tsneDf
= pd.DataFrame(data = x_2D, columns = ['dim 1', 'dim 2'])
final_tsneDf
= pd.concat([tsneDf, pd.DataFrame(data = diagnoses)], axis = 1)
f,
axes = plt.subplots(1, 2, figsize=(15,7))
scatter(final_tsneDf.loc[:,
'dim 1'], final_tsneDf.loc[:, 'dim 2'], ax=axes[0],
hue=diagnoses[:,0])
scatter(final_tsneDf.loc[:,'dim
1'], final_tsneDf.loc[:, 'dim 2'], ax=axes[1], hue=y_kmeans)
scatter(centers[:,0],
centers[:,1], ax=axes[1], marker="s",s=200)
plt.show()
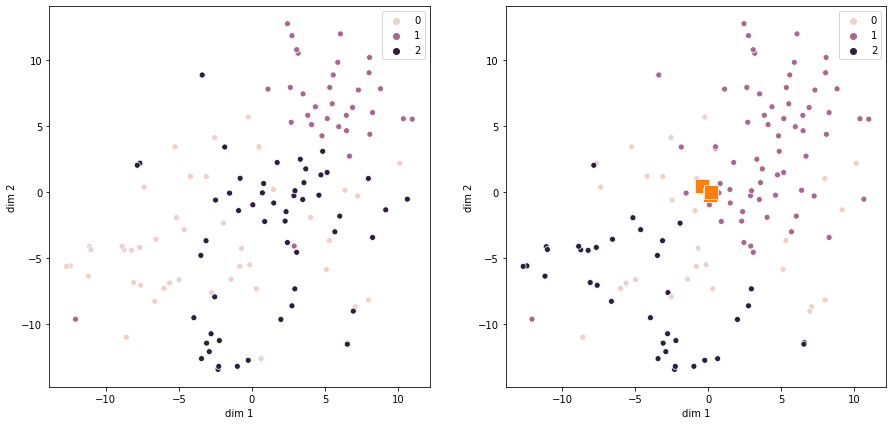
Рисунок 3 — Результат визуализации TSNE по диагнозу(слева) и по результатам KMeans(справа)
6. Провести кластеризацию даных при помощи метода DBSCAN(). Повторить шаги 4-5 для результатов работы DBSCAN().
#PCA
diagnose + DBSCAN
#%%
from
sklearn.cluster import DBSCAN
dbscan =
DBSCAN(eps=9, min_samples=3)
dbscan.fit(x_stan)
f, axes =
plt.subplots(1, 2, figsize=(15,7))
scatter(pcaDF.loc[:,
'principal component 1'], pcaDF.loc[:, 'principal component 2'],
ax=axes[0], hue=diagnoses[:,0])
scatter(pcaDF.loc[:,
'principal component 1'], pcaDF.loc[:, 'principal component 2'],
ax=axes[1], hue=dbscan.labels_)
plt.title("PCA
DBSCAN")
plt.show()
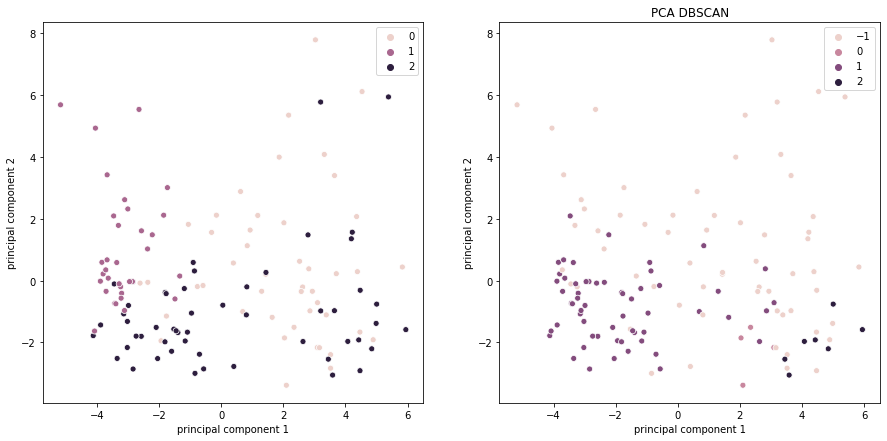
Рисунок 4 — Результат визуализации PCA по диагнозу(слева) и по результатам DBSCAN(справа)
#TSNE
diagnose + DBSCAN
#%%
tsne = TSNE
(n_components = 2, perplexity = 18, n_iter=1000, random_state = 43,
learning_rate = 100)
x_2D =
tsne.fit_transform(x_stan)
tsneDf =
pd.DataFrame(data = x_2D, columns = ['dim 1', 'dim 2'])
final_tsneDf
= pd.concat([tsneDf, pd.DataFrame(data = diagnoses)], axis = 1)
f, axes =
plt.subplots(1, 2, figsize=(15,7))
scatter(final_tsneDf.loc[:,
'dim 1'], final_tsneDf.loc[:, 'dim 2'], ax=axes[0],
hue=diagnoses[:,0])
scatter(final_tsneDf.loc[:,'dim
1'], final_tsneDf.loc[:, 'dim 2'], ax=axes[1], hue=dbscan.labels_)
plt.show()
plt.show()
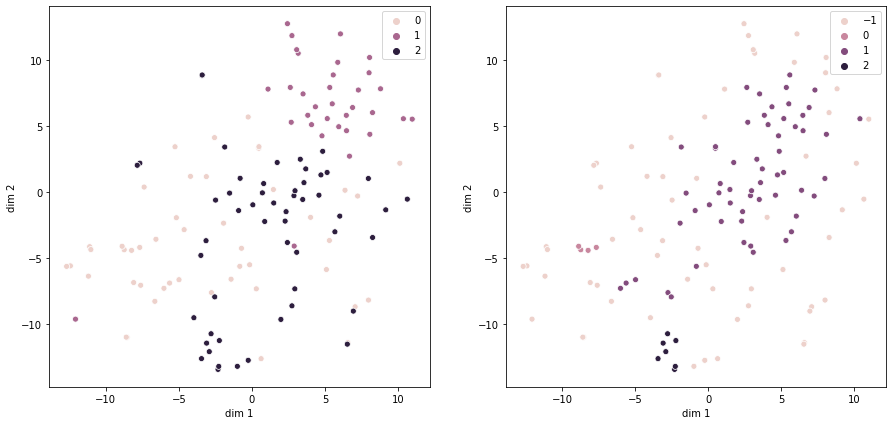
Рисунок
5
— Результат
визуализации TSNE
по диагнозу(слева) и по результатам
DBSCAN(справа)
7.
Провести
оценку
работы
методов
кластеризации
с
использованием
adjusted_rand_score() , adjusted_mutual_info_score() ,
homogeneity_score() , completeness_score(), v_measure_score(),
silhouette_score().
predicted =
dbscan.labels_
print(metrics.adjusted_rand_score(expected[:,0],
predicted))
print(metrics.adjusted_mutual_info_score(expected[:,0],
predicted))
print(metrics.homogeneity_score(expected[:,0],
predicted))
print(metrics.completeness_score(expected[:,0],
predicted))
print(metrics.v_measure_score(expected[:,0],
predicted))
print(metrics.silhouette_score(x,expected[:,0]))
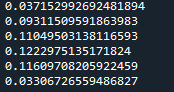
Рисунок 6 – Результаты работы методов кластеризации
Выводы: сравнивая методы визуализации многомерных данных (РСА, TSNE), можно отметить, что РСА был более показательным, так с помощью него можно было увидеть группы данных, объединённых в кластер. Метод кластеризации KMeans показал лучше себя, чем DBSCAN при работе с данными, если сравнивать алгоритмы кластеризации на данном наборе данных. И хоть KMeans был более показательным, нельзя говорить что он лучше во всех случаях, нужно учитывать что DBSCAN не предназначен для работы с данными которые имеют пропуски. Можно отметить что оба алгоритма можно использовать для первичного анализа данных.
Если внимательно изучить результаты подсчёта метрик(рис.6), то можно утверждать, что данная выборка плохо потдаётся кластеризации в принципе: калстеры накладываются друг на друга, изначально данные не полные и самих данных мало.
