
Лаб 2 / Лб2 Віка
.docМІНІСТЕРСТВО ОСВІТИ ТА НАУКИ УКРАЇНИ
ХАРКІВСЬКИЙ НАЦІОНАЛЬНИЙ УНІВЕРСИТЕТ РАДІОЕЛЕКТРОНІКИ
Кафедра СТ
Звіт
про виконання лабораторної роботи № 2
з дисципліни Інтелектуальна обробка даних в розподілених інформаційних середовищах
на тему “ Аналіз методів на основі самонавчання. Кластеризація даних”
Виконав: ст. гр. СПРм-19-1 Шемчук В. Н. |
Перевірила: Перова І. Г. |
Харків 2020
АНАЛИЗ МЕТОДОВ НА ОСНОВЕ САМООБУЧЕНИЯ. КЛАСТЕРИЗАЦИЯ ДАННЫХ
Цель работы: получение навыков работы с методами кластеризации данных.
Ход работы:
1. Загрузить в DataFrame результаты 1-й лабораторной работы.
file = pd.ExcelFile('Dataset.xlsx')
df = pd.read_excel(file, sheet_name='Лист1', header=1);
df_res = dropNaRowByPercent(df, 4);
df_dropNa = df_res.dropna()
resultData = df_res.copy()
for x, row in df_res.iterrows():
for y, value in row.iteritems():
if isNa(value):
resultData.loc[x, y] = generateValue(x, y, len(df_dropNa.index) + 1)
2. Разделить данные и диагноз с использованием метода .loc () и .values (). Провести нормирование или стандартизации данных с использованием метода StandardScaler () или MinMaxScaler () из библиотеки sklearn.
mainData = resultData.loc[:].drop(columns=['№иб']).drop(columns=['Діагноз']).values
diagnoses = resultData.loc[:,['Діагноз']].replace('ХОЗЛ', 0).replace('БА', 2).replace('Пневмонія', 1).values
x_norm = StandardScaler().fit_transform(mainData) from sklearn.preprocessing import StandardScaler
x = StandardScaler().fit_transform(x)
3. Провести кластеризацию даных с помощью метода Kmeans. Оценить качество кластеризации с использованием методов библиотеки klearn metrics.classification_report() и metrics.confusion_matrix() для трёх диагнозов одновременно. symptoms = resultData.loc[:].drop(columns=['№иб']).drop(columns=['Діагноз']).values
diagnoses = resultData.loc[:,['Діагноз']].replace('ХОЗЛ', 0).replace('БА', 2).replace('Пневмонія', 1).values
x_norm = StandardScaler().fit_transform(symptoms)
kmeans = KMeans(n_clusters = 3, init = 'k-means++', random_state = 33)
kMeansPredicted = kmeans.fit_predict(x_norm)
expected = diagnoses
predicted = kMeansPredicted
print(metrics.classification_report(expected, predicted))
print(metrics.confusion_matrix(expected, predicted))
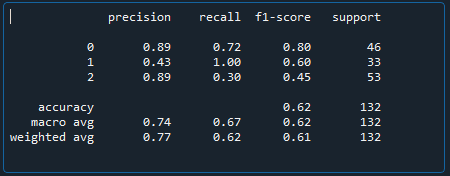

Рисунок 1 — метрики результатов кластеризации методом Kmeans
4. Провести визуализацию даных при помощи метода PCA() на двух радом расположенных рисунках: левый — даные, размеченные согласно значения диагноза; правый — данные, размеченные согласно работе метода Kmeans.
def showPCA(data_right, title_right):
pca = PCA(n_components=2)
pca_diagnosis = pca.fit_transform(x_norm)
principalDf = pd.DataFrame(data = pca_diagnosis [:, 0:2], columns = ['principal component 1', 'principal component 2'])
pcaDF = pd.concat([principalDf, pd.DataFrame(data = diagnoses)], axis=1)
f, axes = plt.subplots(1, 2, figsize=(15,7))
scatter(pcaDF.loc[:, 'principal component 1'], pcaDF.loc[:, 'principal component 2'], ax=axes[0], hue=diagnoses[:,0])
scatter(pcaDF.loc[:, 'principal component 1'], pcaDF.loc[:, 'principal component 2'], ax=axes[1], hue=data_right)
axes[0].set_title('Diagnoses')
axes[1].set_title(title_right)
plt.show()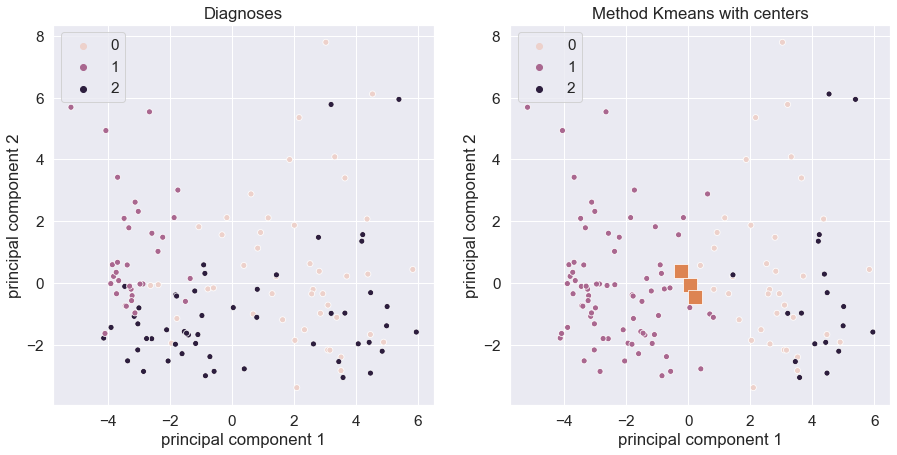
Рисунок 2 — Результат визуализации PCA по диагнозу(слева) и по результатам KMeans(справа)
5. Провести визуализацию даных при помози метода TSNE() на двух расположенных рядом рисунках: левый — даные, размеченные согласно значения диагноза; правый — данные, размеченные согласно работе метода Kmeans. Подобрать значения параметров метода TSNE() для лучшей визуализации. Сделать выводы.
tsne
= TSNE (n_components = 2, perplexity = 18, n_iter=1000, random_state
= 33, learning_rate = 100)
x_2D
= tsne.fit_transform(x_norm)
tsneDf
= pd.DataFrame(data = x_2D, columns = ['dim 1', 'dim 2'])
final_tsneDf
= pd.concat([tsneDf, pd.DataFrame(data = diagnoses)], axis = 1)
f,
axes = plt.subplots(1, 2, figsize=(15,7))
scatter(final_tsneDf.loc[:,
'dim 1'], final_tsneDf.loc[:, 'dim 2'], ax=axes[0],
hue=diagnoses[:,0])
scatter(final_tsneDf.loc[:,'dim
1'], final_tsneDf.loc[:, 'dim 2'], ax=axes[1], hue=kMeansPredicted)
scatter(centers[:,0],
centers[:,1], ax=axes[1], marker="s",s=200)
plt.show()
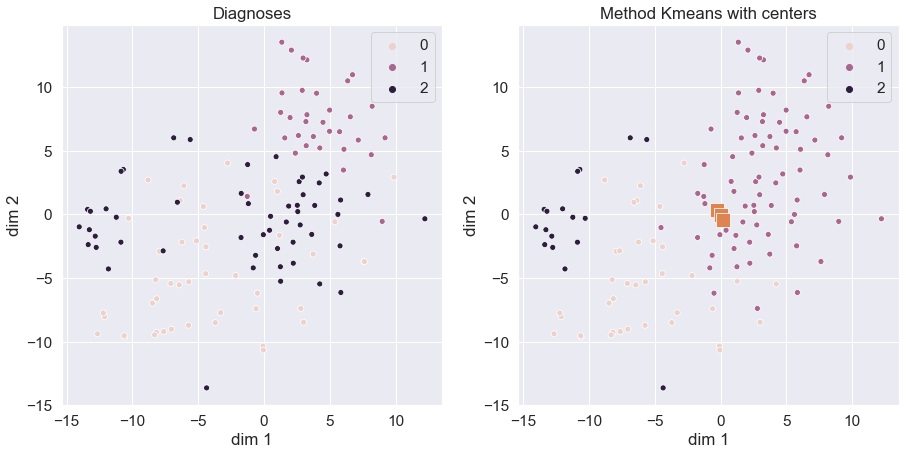
Рисунок 3 — Результат визуализации TSNE по диагнозу(слева) и по результатам KMeans(справа)
6. Провести кластеризацию даных при помощи метода DBSCAN(). Повторить шаги 4-5 для результатов работы DBSCAN().
from
sklearn.cluster import DBSCAN
dbscan
= DBSCAN(eps=9, min_samples=3)
dbscan.fit(x_norm)
showPCA(dbscan.labels_,
'Method DBSAN')
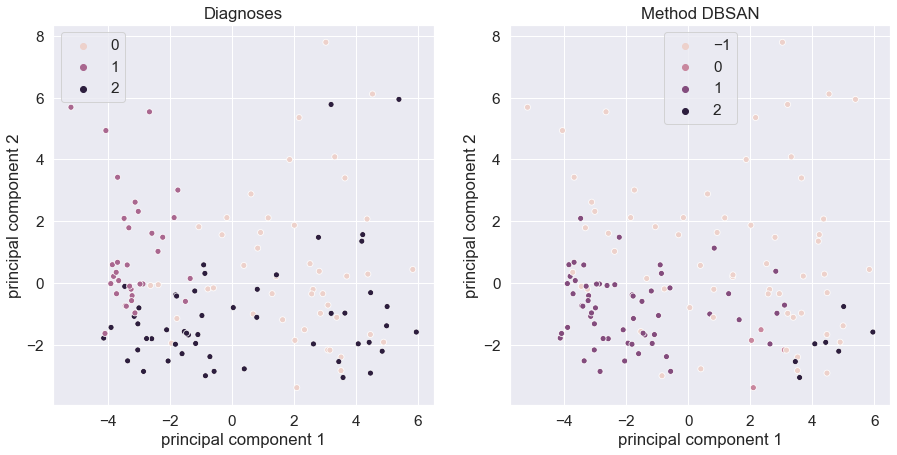
Рисунок 4 — Результат визуализации PCA по диагнозу(слева) и по результатам DBSCAN(справа)
tsne
= TSNE (n_components = 2, perplexity = 18, n_iter=1000, random_state
= 33, learning_rate = 100)
x_2D
= tsne.fit_transform(x_norm)
tsneDf
= pd.DataFrame(data = x_2D, columns = ['dim 1', 'dim 2'])
final_tsneDf
= pd.concat([tsneDf, pd.DataFrame(data = diagnoses)], axis = 1)
f,
axes = plt.subplots(1, 2, figsize=(15,7))
scatter(final_tsneDf.loc[:,
'dim 1'], final_tsneDf.loc[:, 'dim 2'], ax=axes[0],
hue=diagnoses[:,0])
scatter(final_tsneDf.loc[:,
'dim 1'], final_tsneDf.loc[:, 'dim 2'], ax=axes[1],
hue=dbscan.labels_)
axes[0].set_title('Diagnoses')
axes[1].set_title('Method
DBSAN')
plt.show()
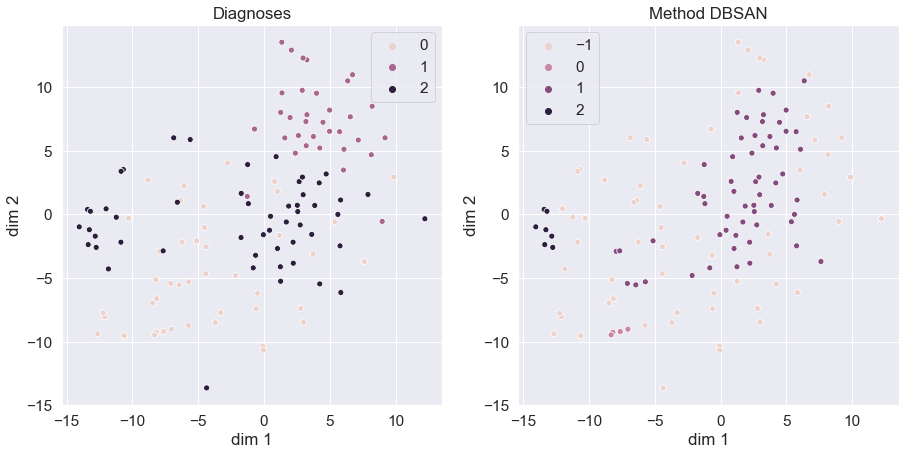
Рисунок
5
— Результат
визуализации TSNE
по диагнозу(слева) и по результатам
DBSCAN(справа)
7.
Провести
оценку
работы
методов
кластеризации
с
использованием
adjusted_rand_score() , adjusted_mutual_info_score() ,
homogeneity_score() , completeness_score(), v_measure_score(),
silhouette_score().
dbscan_predicted
= dbscan.labels_
def
getMetrics(methodName, predictedData):
print('-------------------------------------',
methodName, '-------------------------------------')
print('adjusted_rand_score:
', metrics.adjusted_rand_score(expected[:,0], predictedData))
print('adjusted_mutual_info_score:
', metrics.adjusted_mutual_info_score(expected[:,0], predictedData))
print('homogeneity_score:
', metrics.homogeneity_score(expected[:,0], predictedData))
print('completeness_score:
', metrics.completeness_score(expected[:,0], predictedData))
print('v_measure_score:
', metrics.v_measure_score(expected[:,0], predictedData))
print('silhouette_score:
', metrics.silhouette_score(x_norm, predictedData))
getMetrics('DBSCAN',
dbscan_predicted)
getMetrics('KMEANS',
kMeansPredicted)
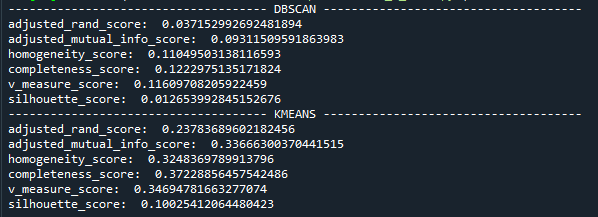
Рисунок 6 – Результаты работы методов кластеризации
Выводы: сравнивая методы визуализации многомерных данных (РСА, TSNE), можно отметить, что РСА был более показательным, так с помощью него можно было увидеть группы данных, объединённых в кластер. Метод кластеризации KMeans показал лучше себя, чем DBSCAN при работе с данными, если сравнивать алгоритмы кластеризации на данном наборе данных. И хоть KMeans был более показательным, нельзя говорить что он лучше во всех случаях, нужно учитывать что DBSCAN не предназначен для работы с данными которые имеют пропуски. DBSCAN необходимо использовать с данными, которые могут иметь шум. Можно отметить, что оба алгоритма можно использовать для первичного анализа данных.
Если внимательно изучить результаты подсчёта метрик(рис.6), то можно утверждать, что данная выборка плохо поддаётся кластеризации в принципе: кластеры накладываются друг на друга, изначально данные не полные и самих данных мало.
