
- •Эффективность пцр-маркеров генов устойчивости пшеницы к бурой ржавчине
- •5.3.3 Обсуждение результатов исследования маркеров
- •1.1.3 Симптомы болезни
- •1.2 Использование молекулярных маркеров для идентификации генов устойчивости пшеницы к бурой ржавчине
- •1.2.1 Гены устойчивости пшеницы к бурой ржавчине (Lr-гены)
- •1.2.2 Молекулярные маркеры Lr-генов
- •1.2.2.1 Restriction Fragment Length Polymorphism (rflp) и Randomly Amplified Polymorphic dna (rapd) – маркеры
- •1.2.2.2 Sequence Tagged Site (sts) маркеры
- •1.2.2.3 Sequence-Characterized Amplified Regions (scar) маркеры
- •1.2.2.4 Cleaved Amplified Polymorphic Sequences (caps) маркеры
- •1.2.2.5 Микросателлитные маркеры (ssr – маркеры)*
- •1.2.2.6 Single Nucleotide Polymorphism (snp) маркеры
- •1.2.3 Использование молекулярных маркеров для идентификации генов устойчивости пшеницы к бурой ржавчине Lr24 и Lr34
- •1.2.3.1 Идентификация гена Lr24
- •1.2.3.2 Идентификация гена Lr34
- •2 Патентный поиск
- •3 Цель и задачи работы
- •4 Экспериментальная часть
- •4.1 Материалы исследования
- •4.2 Методы исследований
- •4.2.1 Выделение днк из пшеницы
- •4.2.2 Электрофорез в горизонтальных агарозных гелях [75]
- •4.2.3 Проведение полимеразной цепной реакции [75]
- •5 Результаты и обсуждения
- •5.1 Определение концентрации днк в рабочем растворе
- •5.2 Изучение маркера гена Lr24
- •5.2.1 Оценка специфичности пяти днк-маркеров гена Lr24
- •5.2.2 Оценка специфичности пяти днк-маркеров гена Lr24 на сортах мягкой пшеницы
- •5.2.3 Обсуждение результатов исследования маркеров гена Lr24
- •5.3 Изучение маркеров гена Lr34
- •5.3.1 Оценка специфичности трех днк-маркеров гена Lr34
- •5.3.2 Идентификация гена Lr34 у сортов мягкой пшеницы
- •5.3.3 Обсуждение результатов исследования маркеров гена Lr34
- •6 Выводы
- •7 Список использованных источников
5.3.2 Идентификация гена Lr34 у сортов мягкой пшеницы
Для наглядной демонстрации практического использования трех вышеперечисленных маркеров гена Lr34 была проведена идентификация гена Lr34 у новых российских сортов озимой пшеницы, рекомендуемых к выращиванию в РФ в 2010 году.
При использовании STS маркера csLV34 для идентификации гена Lr34 у российских сортов характерный фрагмент амплификации размером 150 п.о. был выявлен у 8 из них (№ 1, 4, 6, 8, 9, 10, 11, 14) (Рисунок 19).

М – маркер молекулярного веса 100bp, 1 – АНТОНИВКА, 2 – БЕРЕЗИТ, 3 – ГРОМ, 4 – ДОН 107, 5 - ДОНЭКО, 6 – КАМЫШАНКА 4, 7 - КРАЛЯ, 8 - КСЕНИЯ, 9 - ЛЫТАНИВКА, 10 – НОВОСИБИРСКАЯ 40, 11 – РАПСОДИЯ, 12 – САМУРАЙ, 13 - СИЛА, 14 – СКАРБНИЦА, 15 – ТОРРИЛД, 16 - ЮМПА
Рисунок 19 — Использование STS - маркера csLV34 для идентификации гена Lr34 у российских сортов мягкой пшеницы.
При использовании двух других маркеров Xbarc352 и Xgwm130 характерные фрагменты амплификации размером 248 и 130 п.о. соответственно, были выявлены у всех исследуемых сортов (Рисунок 20, 21).
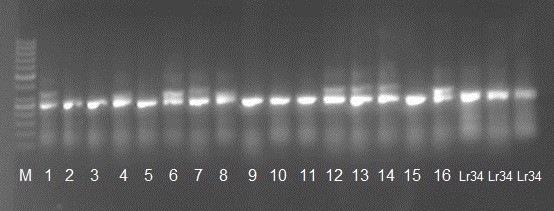
Рисунок 20 — Использование микросателлитного маркера Xbarc352 для идентификации гена Lr34 у российских сортов мягкой пшеницы.

Рисунок 21 — Использование микросателлитного маркера Xgwm130 для идентификации гена Lr34 у российских сортов мягкой пшеницы.
5.3.3 Обсуждение результатов исследования маркеров гена Lr34
Проведенный анализ эффективности маркеров гена Lr34 показал более высокую эффективность при использовании STS маркера. Не смотря на то, что при анализе на Lr-линиях фрагмент амплификации был выявлен как у линии с геном Lr34, а так и у сортов Pavon 76 (Lr46), CSP 44 (Lr48) и VL 404 (Lr49), эти данные не противоречат друг другу. Контроли генов Lr46 , Lr48 и Lr49 представлены не моногенными линиями, а сортами или селекционными образцами пшеницы и теоретически могут нести данный ген. Наличие фрагмента гена Lr34 у сорта с геном Lr46, также несущего ген частичной устойчивости, может указывать на то, что это одни и те же гены, либо наличие в генотипе сорта Pavon 76 (Lr46),кроме гена Lr34 дополнительно гена Lr46.
Использование маркера csLV34 для идентификации Lr34 у сортов пшеницы показало широкое распространение данного гена в российских сортах пшеницы. Среди 16 проанализированных сортов, восемь (50%) несли этот ген.
По данным литературы микросаттелиты Xgwm130 и Xbarc352 фланкируют количественный локус (QTL) гена Lr34. Эта пара маркеров была создана для конкретных гибридных комбинаций и протестирована на нескольких линиях, несущих ген Lr34, что, вероятно и объясняет их низкую эффективность для массового применения.
Микросателлитный локус Xgwm130 расположен проксимально относительно участка количественного локуса гена Lr34 [73]. По данным Schnurbusch и соавторов основная проблема при использовании данного маркера состоит в том, что он может обнаруживать более чем один локус. В связи с этим, информацию, полученную с использованием данного маркера, авторы рекомендуют расценивать как предварительную.
Таким образом, проведенный анализ показал абсолютную неэффективность двух микросателлитных маркеров для практической идентификации. Они абсолютно не идентифицировали генотипы с Lr34 и без него, как у контрольных линий, так и у сортов взятых для анализа. Вероятно, что отсутствие положительных результатов с этим маркером обусловлено использованием агарозного геля вместо полиакриламидного. Однако на практике, данный вид электрофореза не всегда представляется возможным провести, что еще раз указывает на ограничение в использовании данного маркера.
Таким образом, в результате проведенного анализа для практического широкого использования может быть рекомендован STS маркер csLV34.
