
Манило (метода)
.pdf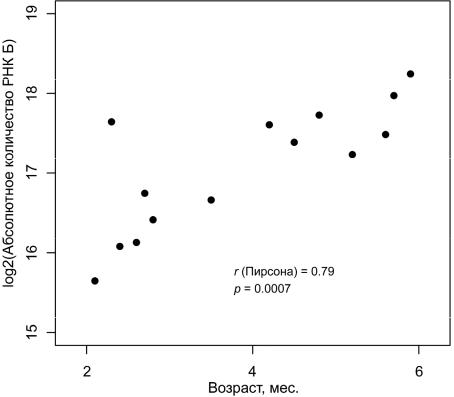
Рис. 3.8. Скатерограмма, иллюстрирующая зависимость между уровнем РНК Б и возрастом экспериментальных животных
> p l o t ( a g e 1 2 , l A Q _ d o 1 2 , x l i m = c ( 0 . 3 , 3 . 1 ) , y l i m = c ( 1 5 , 1 9 ) ,
x a x t = " n " , |
p c h = 1 9 , |
x l a b = " Возраст, |
мес. " , y l a b |
= |
|
" l o g 2 ( Абсолютное количество РНК Б) " ) |
|
|
|||
> |
a x i s ( s i d e = 1 , a t = c ( 1 , 2 , 3 ) ) |
|
|
||
> |
l e g e n d ( 2 , |
1 6 , c ( " r |
( Пирсона) = 0 . 7 8 " , |
" p = 0 . 0 0 1 " ) , |
c e x |
=1 , b t y = " n " , y . i n t e r s p = 1 . 3 )
3.5.4.ROC-анализ для сравнения свойств диагностических признаков
Проверим, можно ли использовать измеренные уровни РНК А и РНК Б в качестве классификаторов, чтобы различать животных из первой и второй групп. Для этого используем ROC-анализ, а именно построим ROC-кривые для каждой РНК и сравним эффективности данных классификаторов.
Для ROC-анализа в программной среде R удобно воспользоваться дополнительным пакетом R O C R . Загрузим и установим его:
>i n s t a l l . p a c k a g e s ( " R O C R " )
>l i b r a r y ( R O C R )
Для каждой РНК зададим новую переменную, которая будет содержать объединение групп соответствующих значений Cq до и после воздействия:
71
>g e n e A = c ( g e n e A _ d o , g e n e A _ p o s )
>g e n e B = c ( g e n e B _ d o , g e n e B _ p o s )
Введем вектор меток групп. При этом элементам в группе с большими значениями присвоим метку 1, а элементам из другой группы – метку 0. Для обеих РНК большие значения Cq принадлежат группе до воздействия, которая занимает в объединении первое место.
> m e t = c ( r e p ( 1 , 2 5 ) , r e p ( 0 , 2 5 ) )
Произведем расчет параметров для ROC-кривых:
> |
r o c A = p e r f o r m a n c e ( p r e d i c t i o n ( g e n e A , m e t ) , " t p r " , |
" f p r " ) |
|
> |
r o c B = p e r f o r m a n c e ( p r e d i c t i o n ( g e n e B , m e t ) , " t p r " , |
" f p r " )
Построим графики, иллюстрирующие отношение чувствительности и специфичности для каждого классификатора, и рассчитаем площади под соответствующими ROC-кривыми (рис. 3.9):
> p l o t ( r o c A , c e x = 4 , l t y = 1 , x l a b =
" 1 -C пецифичность" , y l a b = " Чувствительность" , m a i n = " " )
> p l o t ( r o c B , c e x = 4 , l t y = 2 , a d d = T R U E )
Ниже приведена функция для расчета площади под ROC-кривой в пакете ROCR. Значения AUC присвоим переменным aucA и aucB:
> a u c A |
= u n l i s t ( s l o t ( p e r f o r m a n c e ( p r e d i c t i o n ( g e n e A , m e t ) , |
" a u c " ) , |
" y . v a l u e s " ) ) |
> a u c B |
= u n l i s t ( s l o t ( p e r f o r m a n c e ( p r e d i c t i o n ( g e n e B , m e t ) , |
" a u c " ) , |
" y . v a l u e s " ) ) |
Добавим кполученным графикам легенду срассчитанными значениями AUC:
> |
l e g e n d ( 0 . 4 , 0 . 3 , c e x = |
0 . 9 , c ( " " , " РНК А" , |
" РНК Б" ) , |
l t y = c ( 0 , 1 , 2 ) , b t y = ' n ' ) |
|
||
> |
l e g e n d ( 0 . 5 7 , 0 . 3 , c e x |
= 0 . 9 , c ( " Площадь |
под кривой |
( A U C ) " , r o u n d ( a u c A , 2 ) , r o u n d ( a u c B , 2 ) ) , b t y = ' n ' , y . i n t e r s p = 1 . 3 )
72
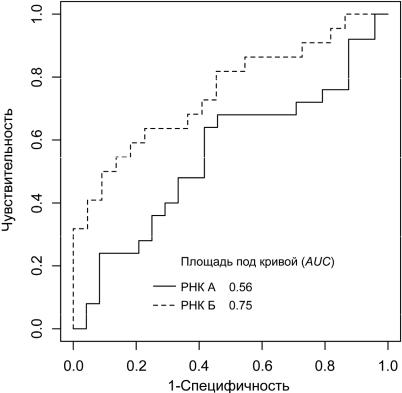
Рис. 3.9. Графики отношения чувствительности и специфичности для РНК А и Б
Результаты показывают, что в качестве параметра для разделения животных на группы до и после воздействия лучше подходит значение Cq для РНК Б, так как площадь под соответствующей ROC-кривой больше площади под ROC-кривой для РНК А.
73
СПИСОК ЛИТЕРАТУРЫ
1.Zar J. H. Biostatistical Analysis. 5th ed. PearsonEducation, 2013.
2.Статистический анализ данных, моделирование и исследование вероятностных закономерностей. Компьютерный подход / Б. Ю. Лемешко, С. Б. Лемешко, С. Н. Постовалов, Е. В. Чимитова. Новосибирск: Изд-во НГТУ, 2011.
3.Кобзарь А. И. Прикладная математическая статистика. Для инженеров
инаучных работников. М.: Физматлит, 2006.
4.Гублер Е. В., Генкин А. А., Применение непараметрических критериев статистики в медико-биологических исследованиях. Л.: Медицина, 1973.
5.Лагутин М. Б. Наглядная математическая статистика: учеб. пособие. 2-е изд. М.: Бином. Лаборатория знаний, 2009.
6.Гланц С. Медико-биологическая статистика / пер. с англ. М.: Практи-
ка, 1998.
7.Мастицкий С. Э., Шитиков В. К. Статистический анализ и визуализация данных с помощью R. М.: ДМК Пресс, 2015.
8.Pritchard C. C., Cheng H. H., Tewari M. MicroRNA profiling: approaches and considerations // Nat. Rev. Genet. 2012. Vol. 13. P. 358–369.
9.ПЦР «в реальном времени» / Д. В. Ребриков, Г. А. Саматов, Д. Ю. Трофимов и др.; под общ. ред. Д. В. Ребрикова. М.: Бином. Лаборатория знаний, 2009.
10.Enzymatic amplification of beta-globin genomic sequences and restriction site analysis for diagnosis of sickle cell anemia / R. K. Saiki, S. Scharf, F. Faloona et al. // Science. 1985. Vol. 230. P. 1350–1354.
11.Кабаков Р. И. R в действии: анализ и визуализация данных в программе R / пер. с англ. М.: ДМК Пресс, 2014.
12.Circulating microRNAs as stable blood-based markers for cancer detection / P. S. Mitchell, R. K. Parkin, E. M. Kroh et al. // Proc. Natl. Acad. Sci. USA. 2008. Vol. 105. P. 10513–10518.
13.Livak K. J., Schmittgen T. D. Analysis of relative gene expression data using real-time quantitative PCR and the 2(-Delta Delta C(T)) Method // Methods. 2001. Vol. 25. P. 402–408.
74
ПРИЛОЖЕНИЯ
1. ПРИМЕРЫ РАСПРОСТРАНЕННЫХ СПОСОБОВ ВИЗУАЛИЗАЦИИ ДАННЫХ ЭКСПЕРИМЕНТА С ПОМОЩЬЮ СРЕДЫ R
1.1.Альтернативные способы графического представления средних
ииндивидуальных значений параметра
Рассмотрим эксперимент, в котором измерены значения некоторого параметра в образцах, принадлежащих двум исследуемым группам. Экспериментальные данные в виде таблицы введем с клавиатуры:
> e x p _ d a t a < - r e a d . t a b l e ( h e a d e r = T R U E , t e x t = '
Номер_ образца |
Номер_ группы |
Значение_ параметра |
1 |
1 |
5 . 5 |
2 |
1 |
8 |
3 |
1 |
4 |
4 |
1 |
3 . 5 |
5 |
2 |
7 . 5 |
6 |
2 |
8 |
7 |
2 |
9 |
8 |
2 |
7 ' ) |
Представим результаты измерений графически четырьмя разными способами. Загрузим и установим в R дополнительные пакеты, необходимые для использования функций b a r p l o t 2 ( ) и p l o t C I ():
>i n s t a l l . p a c k a g e s ( " g p l o t s " )
>l i b r a r y ( g p l o t s )
Найдем для каждой группы средние значения и стандартные отклонения:
> m 1 = m e a n ( e x p _ d a t a |
[ 1 : 4 , 3 ] ) |
||
> m 2 = m e a n ( e x p _ d a t a |
[ 5 : 8 , 3 ] ) |
||
> |
s t d e v 1 = s d ( e x p _ d a t a |
[ 1 : 4 , 3 ] ) |
|
> |
s t d e v 2 = s d ( e x p _ d a t a |
[ 5 : 8 , 3 ] ) |
|
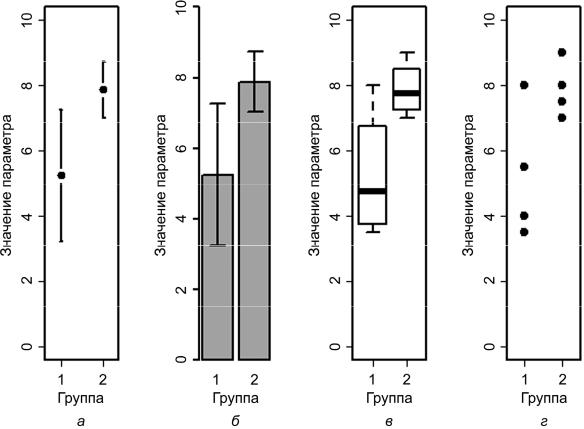
На рис. П1.1, а представлена точечная диаграмма, где для каждой группы точкой обозначено полученное среднее значение, а «усы» соответствуют величине стандартного отклонения:
> p a r ( m f r o w = c ( 1 , 4 ) )
75
> p l o t C I ( x = c ( 1 , 2 ) , y = c ( m 1 , m 2 ) , u i w =
c ( s t d e v 1 , s t d e v 2 ) , l i w = c ( s t d e v 1 , s t d e v 2 ) , x l a b = " Группа" , y l a b = " Значение параметра" ,
x l i m = c ( 0 . 7 , 2 . 3 ) , y l i m = c ( 0 , 1 0 ) , x a x t = " n " , p c h = 1 9 , c e x = 1 . 5 , l w d = 2 , c e x . l a b = 1 . 3 , m a i n = " " )
> a x i s ( 1 , a t = s e q ( 1 , 2 , b y = 1 ) )
На рис. П1.1, б те же данные представлены в виде столбчатой диаграммы (длина «усов» также соответствует стандартному отклонению для каждой группы):
> p a r ( l w d = 2 )
> b a r p l o t 2 ( c ( m 1 , m 2 ) , p l o t . c i = T R U E , c i . l =
c ( m 1 + s t d e v 1 , m 2 + s t d e v 2 ) , c i . u = c ( m 1 - s t d e v 1 , m 2 - s t d e v 2 ) ,
n a m e s . a r g = c ( " 1 " , " 2 " ) , c i . l w d = 2 , |
x l a b |
= " Группа" , y l a b |
|
= |
" Значение параметра" , y l i m = c ( 0 |
, 1 0 ) , |
l w d = 2 , c e x . l a b |
= |
1 . 3 , m a i n = " " ) |
|
|
Рис. П1.1. Способы представления результатов измерений:
а– точечная диаграмма; б – столбчатая диаграмма;
в– боксплот; г – диаграмма рассеяния
76
На рис. П1.1, в боксплоты иллюстрируют распределение данных в группах, при этом жирные горизонтальные линии соответствуют медианам, а границы «усов» минимальным и максимальным значениям в группах (при условии отсутствия выбросов в выборках):
> b o x p l o t ( v a l u e _ a l l ~ g r o u p I D , |
x l a b = " Группа" , |
y l a b = " Значение параметра" , |
y l i m = c ( 0 , 1 0 ) , l w d = 2 , |
c e x . l a b = 1 . 3 , m a i n = " " ) |
|
На рис. П1.1, г значения параметра в индивидуальных образцах представлены в виде диаграммы рассеяния:
> s t r i p c h a r t ( v a l u e _ a l l ~ g r o u p I D , m e t h o d = " s t a c k " ,
v e r t i c a l = T R U E , x l a b = " Группа" , y l a b = " Значение параметра" , x l i m = c ( 0 . 6 , 2 . 4 ) , y l i m = c ( 0 , 1 0 ) , p c h = 1 9 , c e x = 1 . 7 , c e x . l a b = 1 . 3 , m a i n = " " )
Кроме того, полученные диаграммы можно совмещать, например диаграмму рассеяния и боксплот. Для этого необходимо последовательно использовать соответствующие функции для диаграмм и в аргументах второй из них указать параметр add = TRUE.
1.2. Анализ связи между значениями нескольких параметров
Рассмотрим эксперимент, в котором значения некоторых четырех параметров измерены в образцах, принадлежащих двум группам. Экспериментальные данные в виде таблицы введем с клавиатуры:
> e x p _ d a t a < - r e a d . t a b l e ( h e a d e r = T R U E , c o l C l a s s e s = c ( " N U L L " , " N U L L " , N A , N A , N A , N A ) , t e x t = '
Номер |
Номер |
Значение |
Значение |
Значение |
Значение |
образца |
группы |
параметра1 |
Параметра2 |
Параметра3 |
Параметра4 |
1 |
1 |
2 0 |
1 4 |
1 0 |
5 9 |
2 |
1 |
2 1 |
1 9 |
1 2 |
6 5 |
3 |
1 |
2 4 |
2 6 |
1 3 |
5 0 |
4 |
1 |
2 2 |
2 9 |
2 4 |
4 6 |
5 |
2 |
2 3 |
7 6 |
5 2 |
7 |
6 |
2 |
2 1 |
4 8 |
4 1 |
1 8 |
7 |
2 |
2 6 |
5 7 |
6 7 |
3 |
8 |
2 |
2 1 |
6 4 |
6 6 |
1 ' ) |
77

Загрузим и установим в R дополнительный пакет, необходимый для ис-
пользования функции c h a r t . C o r r e l a t i o n ( ) :
> |
i n s t a l l . p a c k a g e s ( " P e r f o r m a n c e A n a l y t i c s " ) |
> |
l i b r a r y ( P e r f o r m a n c e A n a l y t i c s ) |
Применим корреляционный анализ, для того чтобы количественно оценить связь исследуемых параметров и значимость полученной оценки. Рассчитаем коэффициент корреляции Спирмена и его фактический уровень значимости (р-значение) для каждой пары параметров. Представим результаты расчета с помощью матричной скатерограммы (рис. П1.2):
> c h a r t . C o r r e l a t i o n ( e x p _ d a t a , h i s t o g r a m = F A L S E , p c h = 1 9 , l w d = 2 , m e t h o d = " s p e a r m a n " , m a i n = " " )
Рис. П1.2. Матричная скатерограмма
На рис. П1.2 в нижней левой части матрицы для каждой пары параметров изображены скатерограммы. Кроме этого, для каждой пары параметров цифры в ячейках верхней правой части матрицы представляют коэффициенты корреляции Спирмена, а звездочки указывают соответствующие уровни значимости (р-значения) – “***”, “**”, “*” <=> 0, 0.001, 0.01.
78
1.3.Графическое представление значений параметров
спомощью теплокарт
Вслучае если в эксперименте измерены значения многих однотипных параметров (например, уровней экспрессии генов) или/и измерение значений параметра произведено во многих группах, для графического представления всего массива таких данных удобно использовать теплокарты. При этом способе визуализации измерению конкретного параметра в конкретном образце соответствует одна клетка на карте, а значение параметра определяет цвет клетки. При построении теплокарт образцы или/и параметры могут быть дополнительно сгруппированы друг с другом на основании степени близости значений параметров, а результаты определения степени сходства визуализированы с помощью дендрограмм.
Загрузим и установим в R дополнительные пакеты, необходимые для использования функций h e a t m a p . 2 ( ) :
>i n s t a l l . p a c k a g e s ( " g p l o t s " )
>l i b r a r y ( g p l o t s )
Визуализируем с помощью теплокарты данные эксперимента, описанного в 1.2. Для этого запишем таблицу данных в виде матрицы:
> e x p _ d a t a _ m a t r i x < - d a t a . m a t r i x ( e x p _ d a t a )
Сгруппируем строки матрицы:
> h r < - h c l u s t ( a s . d i s t ( 1 - c o r ( t ( e x p _ d a t a _ m a t r i x ) , m e t h - o d = " s p e a r m a n " ) ) , m e t h o d = " c o m p l e t e " )
Сгруппируем столбцы матрицы:
> h c < - h c l u s t ( a s . d i s t ( 1 - c o r ( e x p _ d a t a _ m a t r i x , m e t h - o d = " s p e a r m a n " ) ) , m e t h o d = " c o m p l e t e " )
Зададимпалитруцветов, используемуюдлявизуализациизначенийпараметров: > p a l e t t e < - c o l o r R a m p P a l e t t e ( c ( " w h i t e " , " b l a c k " ) ) ( 1 0 0 )
Построим теплокарту (рис. П1.3, а):
> h e a t m a p . 2 ( e x p _ d a t a _ m a t r i x , R o w v = a s . d e n d r o g r a m ( h r ) , C o l v = a s . d e n d r o g r a m ( h c ) , s c a l e = " n o n e " , c o l = p a l e t t e ,
d e n s i t y . i n f o = " n o n e " , t r a c e = " n o n e " , R o w S i d e C o l o r s =
c ( r e p ( " g r a y 3 0 " , 4 ) , r e p ( " g r a y 8 0 " , 4 ) ) , m a r g i n s = c ( 1 0 , 5 ) , c e x R o w = 1 . 8 , c e x C o l = 1 . 8 )
79
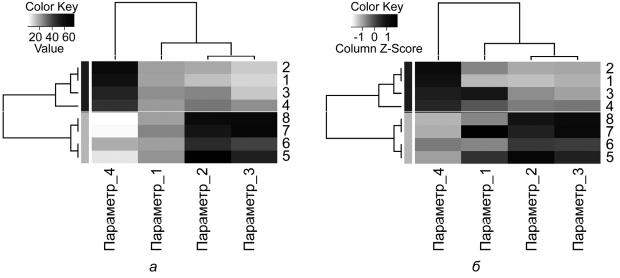
Рис. П1.3. Теплокарты: а – при значении параметра scale="none"; б – при значении параметра scale="col"
Функция h e a t m a p . 2 ( ) позволяет визуализировать данные, предварительно выполнив нормировку значений параметров в каждом столбце или каждой строке, для этого параметру s c a l e присваивается значение " c o l " или " r o w " . Построим теплокарту, выполнив нормировку значений параметров в каждом столбце (рис. П1.3, б). На рис. П1.3 показан вид полученных теплокарт. Более светлые клетки соответствуют меньшим значениям параметров. Дендрограмма сверху от теплокарт определяет группировку параметров, а дендрограмма слева определяет группировку образцов. Цвет полосы слева от теплокарт указывает на принадлежность образцов группам: темно-серый – группа 1, светло-серый – группа 2.
1.4. Изменение параметров осей и надписей на рисунках
Графические функции R позволяют контролировать параметры осей и надписей на рисунках. На рис. П1.4 приведен пример, иллюстрирующий способы изменения названия и масштаба осей, расположения делений и значений по осям, расположения заголовков и условных обозначений, изменения размера и стиля шрифтов, а также способы добавления верхних и нижних индексов, специальных символов и греческих букв.
>x < - 1 : 1 0
>y < - x * x
>p 1 = 0 . 0 2 3 4
>p 2 = 6 . 7 6 5 2
80
