
Манило (метода)
.pdfГлава 3. ПРИМЕР СТАТИСТИЧЕСКОГО АНАЛИЗА ДАННЫХ ПЦР
ВРЕАЛЬНОМ ВРЕМЕНИ С ПОМОЩЬЮ СРЕДЫ R
3.1.Описание условий эксперимента и задач исследования
Целью эксперимента было определение влияния экспериментального препарата на экспрессию генов, а именно, уровни РНК этих генов, участвующих в развитии ожирения, на модели лабораторных мышей. Чтобы дополнительно определить зависимость уровней РНК от возраста, в эксперимент были включены животные разных возрастов – молодые (<3 мес.), взрослые (3–6 мес.) и старые (>12 мес.). Возраст каждой мыши на момент начала эксперимента был точно известен. Образцы цельной крови забирали у каждой мыши до и после введения препарата. Из цельной крови выделяли образцы общей РНК. Концентрация РНК была измерена спектрофотометрическим методом. Уровни исследуемых РНК А и РНК Б количественно определяли в образцах общей РНК с помощью ПЦР в реальном времени, при этом для каждой реакции обратной транскрипции использовали одинаковое количество общей РНК. Кроме того, ПЦР в реальном времени применяли для определения уровней РНК А и РНК Б в последовательных десятикратных разведениях растворов, содержащих известные концентрации фрагментов синтетической РНК идентичные анализируемым участкам РНК А и РНК Б. Данные, полученные с применением растворов синтетических РНК, будут использованы для расчета абсолютных количеств РНК Б, а также для определения линейных диапазонов и эффективностей амплификации систем для детекции РНК А и РНК Б. При анализе растворов синтетических РНК амплификация была проведена в двух технических репликатах, все остальные реакции ПЦР, а также все реакции обратной транскрипции выполняли без технических репликатов. Для каждого образца обратная транскрипция РНК А и РНК Б проходила в одной и той же пробирке, а амплификация – в разных пробирках. При этом амплификация конкретных РНК во всех образцах, включая растворы синтетических РНК известных концентраций, была проведена в ходе одного запуска амплификатора и полученные таким образом результаты не требовали межплашечной калибровки.
Технические задачи эксперимента:
1)определить диапазон линейной зависимости Cq от логарифма концентрации матрицы для систем детекции РНК А и РНК Б;
2)выяснить, может ли РНК А быть использована в качестве референсной, т. е.
51
–не меняются ли ее уровни в экспериментальных группах,
–совпадают ли эффективности амплификаций РНК А и РНК Б;
3)выявить и исключить «выбросы»;
4)проверить выполнение условий нормальности распределения и однородности дисперсий полученных данных.
Биологические задачи эксперимента:
1)изучитьвлияние экспериментального препарата на уровень РНК Б, т. е.
–проверить гипотезу о равенстве средних значений в группах образцов до и после воздействия,
–рассчитать абсолютные значения РНК Б в каждом из экспериментальных образцов;
2)изучить зависимость уровней РНК Б до воздействия от возраста, т. е.
–проверить гипотезу о равенстве средних значений в трех возрастных группах,
–рассчитать средние относительные количества РНК Б в трех возрастных группах;
3)изучить связи между параметрами экспериментальной системы, т. е. проверить наличие связи между возрастом животных и уровнем РНК Б до воздействия;
4)c помощью ROC-анализа выполнить характеристику измеренных уровней РНК А и РНК Б в качестве диагностических признаков для классификации экспериментальных образцов.
3.2. Инструменты анализа
Для проведения статистического анализа и визуализации результатов вычислений использована открытая среда R (www.r-project.org). Для облегчения работы с кодом можно дополнительно установить удобный графический интерфейс R-studio (www.r-studio.com). R – это язык программирования и среда для выполнения статистических расчетов с хорошо развитой графикой. Базовая версия программы не перегружена и содержит только широко используемые функции. Однако в зависимости от требований пользователя функциональность программы может быть значительно расширена посредством подключения дополнительных специализированных пакетов, широкий спектр которых ориентирован на анализ медико-биологических данных. Для более подробного знакомства с R можно рекомендовать следующую литературу: [7], [11]. Кроме того, информацию о назначении и аргументы всех функций легко получить, набрав непосредственно в рабочем поле программы знак вопроса и имя интересующей функции, например: ? t.test.
52
3.3.Ввод данных
Впервую очередь, важно записать информацию об экспериментальных образцах и группах, а также измеренные значения Cq в экспериментальных образцах в таблицу и сохранить в виде текстового файла exp.txt (табл. 3.1).
Таблица. 3.1
Данные о возрасте животных и значениях Cq в экспериментальных образцах
1. Номер |
2. Номер |
3. Возраст, |
4. Cq РНК А |
5. Cq РНК Б |
6. Cq РНК А |
7. Cq РНК Б |
образца |
группы |
мес. |
до воздействия |
до воздействия |
после |
после |
воздействия |
воздействия |
|||||
1 |
1 |
2.1 |
20.61 |
32.07 |
21.26 |
29.16 |
2 |
1 |
2.7 |
21.90 |
30.95 |
21.13 |
29.43 |
3 |
1 |
2.9 |
21.77 |
NA |
20.29 |
30.23 |
4 |
1 |
2.6 |
21.48 |
36.85 |
21.64 |
29.16 |
5 |
1 |
2.3 |
21.56 |
30.03 |
21.13 |
30.37 |
6 |
1 |
2.4 |
21.14 |
31.63 |
20.36 |
29.75 |
7 |
1 |
2.6 |
20.45 |
31.58 |
21.64 |
30.14 |
8 |
1 |
2.8 |
21.76 |
31.29 |
21.46 |
29.02 |
9 |
2 |
3.5 |
21.21 |
31.03 |
20.67 |
28.25 |
10 |
2 |
3.8 |
21.22 |
30.07 |
29.73 |
37.13 |
11 |
2 |
4.2 |
21.22 |
30.07 |
20.87 |
28.15 |
12 |
2 |
4.5 |
20.54 |
30.29 |
20.59 |
28.43 |
13 |
2 |
5.7 |
20.34 |
29.70 |
21.16 |
29.25 |
14 |
2 |
5.9 |
21.75 |
29.42 |
22.12 |
28.65 |
15 |
2 |
5.2 |
22.09 |
30.45 |
21.01 |
27.48 |
16 |
2 |
5.6 |
20.57 |
30.19 |
21.35 |
28.23 |
17 |
2 |
4.8 |
21.35 |
29.95 |
20.88 |
28.63 |
18 |
3 |
18.0 |
20.33 |
29.13 |
21.13 |
27.43 |
19 |
3 |
18.3 |
20.88 |
28.93 |
21.72 |
28.22 |
20 |
3 |
18.5 |
21.35 |
28.83 |
20.40 |
29.14 |
21 |
3 |
19.0 |
21.46 |
28.44 |
21.88 |
28.93 |
22 |
3 |
19.5 |
21.20 |
28.22 |
21.49 |
27.96 |
23 |
3 |
19.9 |
20.57 |
28.04 |
20.60 |
29.95 |
24 |
3 |
19.3 |
21.37 |
28.75 |
21.00 |
27.68 |
25 |
3 |
19.7 |
21.72 |
27.94 |
21.28 |
28.34 |
Столбец 1 будет содержать номера образцов, столбец 2 – номера экспериментальных групп (1 – молодые, 2 – взрослые, 3 – старые), столбец 3 – возраст мышей в месяцах, столбец 4 – значения Cq для РНК A до воздействия, столбец 5 – значения Cq для РНК Б до воздействия, столбец 6 – значения Cq для РНК А после воздействия, столбец 7 – значения Cq для РНК Б после воздействия.
Затем нужно записать данные о значениях Cq в образцах РНК А и РНК Б известной концентрации в таблицу и сохранить в виде текстового файла dop.txt (табл. 3.2). Столбец 1 будет содержать концентрацию синтетической
53
матрицы (количество копий в микролитре реакции обратной транскрипции), столбец 2 – значения Cq при амплификации синтетической РНК А, столбец 3 – значения Cq при амплификации синтетической РНК Б. Два значения Cq для каждой концентрации синтетической матрицы соответствуют двум техническим репликатам.
Таблица. 3.2
Данные о значениях Cq при амплификации РНК А и РНК Б из образцов синтетической РНК известной концентрации
1. Количество копий в микролитре |
2. Cq при амплификации |
3. Cq при амплификации |
реакции обратной транскрипции |
синтетической РНК А |
синтетической РНК Б |
100 |
37.16 |
35.93 |
100 |
36.54 |
35.99 |
1000 |
36.61 |
32.96 |
1000 |
35.84 |
32.92 |
10 000 |
35.25 |
29.54 |
10 000 |
34.56 |
29.57 |
100 000 |
31.58 |
25.99 |
100 000 |
31.62 |
26.38 |
1 000 000 |
27.61 |
22.98 |
1 000 000 |
27.45 |
22.84 |
10 000 000 |
24.03 |
19.20 |
10 000 000 |
23.96 |
19.54 |
100 000 000 |
20.88 |
15.55 |
100 000 000 |
20.74 |
15.56 |
1 000 000 000 |
17.65 |
13.75 |
1 000 000 000 |
17.60 |
13.81 |
Для начала работы в R следует определить путь к рабочей папке программы. Это можно сделать следующим образом:
> s e t w d ( " C : / рабочая папка программы" )
Загружаемые в R файлы с данными, представленными в табличном виде, могут иметь формат .txt или .xls. В качестве десятичного разделителя следует использовать точку. Отметим, что в исходных данных отсутствует значение Cq РНК Б у третьего образца в группе до воздействия. Такие пропущенные значения следует заменить на NA (not available), для того чтобы R воспринимал эти значения именно как пропущенные, а не как равные нулю. Для загрузки текстового файла с данными, представленными в виде таблицы, в R можно использовать функцию r e a d . t a b l e ( ) . Загружаемые данные удобно присвоить некоторой переменной (в данном случае матричного типа), имя которой пользователь задает произвольно. Чтобы указать, что первая строка таблиц содержит не данные, а названия столбцов, параметру h e a d e r присваивается значение TRUE. В данном примере первые две строки таблиц не
54
содержат данных. Чтобы это учесть, параметру s k i p присвоим значение 2. Итак, разместим файлы с данными проведенного эксперимента в рабочей папке и загрузим их в программу:
> |
e x p = r e a d . t a b l e ( s k i p = 2 , " e x p . t x t " ) |
> |
d o p = r e a d . t a b l e ( s k i p = 2 , " d o p . t x t " ) |
Проверить введенные данные можно, отобразив в рабочем поле программы содержание соответствующих переменных. Для этого введем имена этих переменных:
>e x p
>d o p
Для дальнейшего анализа понадобятся данные из определенных строк и столбцов загруженных таблиц. Например, чтобы обратиться к элементу таблицы, располагающемуся на пересечении строки i и столбца j переменной e x p , используем exp [i, j]. Таким образом, определим необходимые для дальнейшего анализа переменные, записав в них данные из соответствующих строк или столбцов загруженных таблиц:
> |
g r o u p |
= e x p [ , 2 ] |
# номера групп образцов |
> |
a g e = |
e x p [ , 3 ] # |
значения параметра |
> g e n e A _ d o = e x p [ , 4 ] # значения C q РНК А до воздействия
> g e n e B _ d o = e x p [ , 5 ] # значения C q РНК Б до воздействия
>g e n e A _ p o s = e x p [ , 6 ] # значения C q РНК А после воздействия
>g e n e B _ p o s = e x p [ , 7 ] # значения C q РНК Б после воздействия
>c o n c = d o p [ , 1 ] # известные значения концентраций синтетических РНК А и РНК Б
> c q A = d o p [ , 2 ] # значения C q РНК А в образцах с известной концентрацией
> c q B = d o p [ , 3 ] # значения C q РНК Б в образцах с известной концентрацией
В следующем разделе с помощью методов предварительного анализа данных будут определены их характеристики, которые необходимо учитывать при выборе адекватных методов статистического анализа.
55
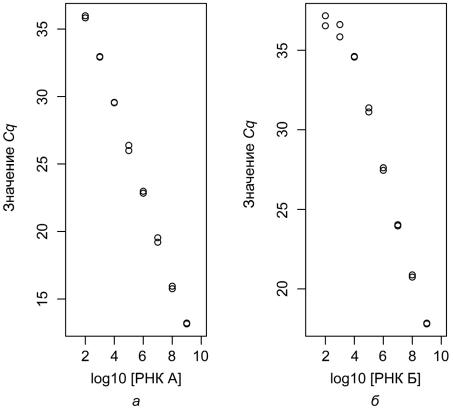
3.4.Предварительный анализ
3.4.1.Характеристика систем детекции
Для того чтобы оценить границы линейных участков систем детекции РНК А и РНК Б построим графики зависимостей значений Cq от логарифма концентраций соответствующих РНК (калибровочные графики) (рис. 3.1). Чтобы разместить оба графика на одном рисунке, предварительно сделаем разбивку графической области, указав, сколько строк и столбцов должно быть в матрице графиков, с помощью функции p a r ( ) . Затем последовательно построим два графика с помощью функции p l o t ( ) .
> p a r ( m f r o w = c ( 1 , 2 ) ) |
|
|
|
|
||||
> |
p l o t ( l o g 1 0 ( c o n c ) , |
c q A , x l i m = c ( 1 , 1 0 ) , |
||||||
y l a b |
= |
" Значение C q " , |
x l a b |
= |
" l o g 1 0 |
[ РНК А] " ) |
||
> |
p l o t ( l o g 1 0 ( c o n c ) , |
c q B , x l i m = c ( 1 , 1 0 ) , |
||||||
y l a b |
= |
" Значение C q " , |
x l a b |
= |
" l o g 1 0 |
[ РНК Б] " ) |
||
Из рис. 3.1 видно, что при использовании системы детекции РНК А нет существенных отклонений от линейной зависимости, а при детекции РНК Б ли-
Рис. 3.1. Оценка границ линейных участков зависимости значений Cq от логарифма концентрации РНК:
а– зависимость для системы детекции РНК А;
б– зависимость для системы детекции РНК Б
56
нейность связи нарушается в двух образцах с минимальными концентрациями. Поэтому удаляем из данных значения Cq, соответствующиеэтим образцам:
> c q B = c q B [ - 1 : - 4 ]
Используя обновленную переменную cqB, можно будет выполнить характеристику системы детекции РНК Б в диапазоне концентраций, где сохраняется линейная зависимость Cq от логарифма концентрации РНК.
Далее построим калибровочные графики для линейных диапазонов зависимости Cq от логарифма концентраций РНК А и РНК Б. Для этого используем линейный регрессионный анализ. Найти параметры модели линейной регрессии в R для исследуемой зависимости можно с помощью функции l m ( ) . Удобно присвоить некоторой переменной массив параметров рассчитанной линейной модели, для того чтобы использовать эти параметры в дальнейших расчетах. Таким образом, получим модель линейной регрессии для РНК А:
> m A = l m ( c q A ~ l o g 1 0 ( c o n c ) )
Определим необходимые параметры полученной модели линейной регрессии и найдем их значения (где Y _ i n t – координаты точки пересечения прямой с осью Y, S l o p e – угол наклона прямой, R – коэффициент детерминации):
> Y _ i n t = c o e f ( m A ) [ [ 1 ] ] ; Y _ i n t
[ 1 ] 4 2 . 7 1
> S l o p e = c o e f ( m A ) [ [ 2 ] ] ; S l o p e
[ 1 ] - 3 . 3 1
> R = s u m m a r y ( m A ) [ [ 8 ] ] ; R
[ 1 ] 0 . 9 9 9
Используя найденные значения, рассчитаем эффективность реакции амплификации в процентах для РНК А:
> E = ( 1 0 ^ ( - 1 / c o e f ( m A ) [ [ 2 ] ] ) - 1 ) * 1 0 0 ; E
[ 1 ] 1 0 0
Нанесем на график зависимости значений Cq от логарифма концентрации РНК А соответствующую регрессионную прямую (рис. 3.2, а):
> |
p l o t ( l o g 1 0 ( c o n c ) , c q A , x l i m = c ( 1 , 1 0 ) , y l i m = |
c ( 1 0 , 4 0 ) , |
||||
c e x |
= |
0 |
. 6 , |
p c h = 2 , c o l = " b l a c k " , x l a b = " l o g 1 0 |
[ РНК |
|
А] " , |
y l a b |
= " Значение C q " ) |
|
|||
> |
l i n e s ( l o g 1 0 ( c o n c ) , c o l = " b l a c k " , m A $ f i t t e d , l w d = 1 , |
|||||
c e x |
= |
0 |
. 8 ) |
|
|
|
57
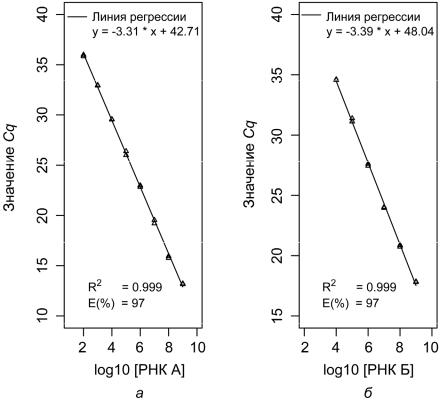
Рис. 3.2. Калибровочные графики: а – для РНК А; б – для РНК Б
Добавим на график уравнение линейной регрессии:
> l e g e n d ( " t o p l e f t " , c ( " Линия регрессии" , " y = - 3 . 3 1 * x + 4 2 . 7 1 " ) , l t y = c ( 1 , 1 ) , c o l = c ( 1 , 0 ) , c e x = 0 . 8 , b t y = ' n ' , y . i n t e r s p = 1 . 3 )
Нанесем на график рассчитанные значения коэффициента детерминации и значение эффективности амплификации, предварительно выполнив округление значений с помощью функции r o u n d ( ) :
> l e g e n d ( " b o t t o m l e f t " , c ( e x p r e s s i o n ( R ^ 2 ) , " E ( % ) " , e v a l ( s u b s t i t u t e ( p a s t e ( " = " , f l ) , l i s t ( f l = r o u n d ( s u m m a r y ( m A ) [ [ 8 ] ] , 3 ) ) ) ) , e v a l ( s u b s t i t u t e ( p a s t e ( " = " , f l ) , l i s t ( f l =
r o u n d ( E , 0 ) ) ) ) ) , n c o l = 2 , b t y = ' n ' , t e x t . w i d t h = 0 . 3 5 , y . i n t e r s p = 1 . 5 )
Аналогично для РНК Б выполним необходимые расчеты и построим калибровочный график (рис. 3.2, б):
> m B = l m ( c q B ~ l o g 1 0 ( c o n c ) [ 5 : 1 6 ] )
> |
Y _ i n t |
= |
c o e f ( m B ) [ [ 1 ] ] |
> |
S l o p e |
= |
c o e f ( m B ) [ [ 2 ] ] |
> R = s u m m a r y ( m B ) [ [ 8 ] ]
58
> E = ( 1 0 ^ ( - 1 / c o e f ( m B ) [ [ 2 ] ] ) - 1 ) * 1 0 0
> p l o t ( l o g 1 0 ( c o n c ) [ 5 : 1 6 ] , c q B , x l i m = c ( 2 , 1 0 ) , y l i m =
c ( 1 5 , 4 0 ) , c e x = 0 . 6 , p c h = 2 , c o l = " b l a c k " , x l a b = " l o g 1 0 [ РНК Б] " , y l a b = " Значение C q " , c e x . m a i n = 0 . 8 )
> l i n e s ( l o g 1 0 ( c o n c ) [ 5 : 1 6 ] , c o l = " b l a c k " , m B $ f i t t e d , l w d = 1 )
> l e g e n d ( " t o p l e f t " , c ( " Линия регрессии" , " y = - 3 . 3 9 * x
+ |
4 8 . 0 4 " ) , l t y = c ( 1 , 1 ) , c o l = c ( 1 , 0 ) , c e x = 0 . 8 , b t y = ' n ' ) |
> |
l e g e n d ( " b o t t o m l e f t " , c ( e x p r e s s i o n ( R ^ 2 ) , " E ( % ) " , e v a l |
( s u b s t i t u t e ( p a s t e ( " = " , f l ) , l i s t ( f l = R , 3 ) ) ) ) , e v a l ( s u b s t i - t u t e ( p a s t e ( " = " , f l ) , l i s t ( f l = r o u n d ( E , 0 ) ) ) ) ) , n c o l = 2 , b t y = ' n ' , t e x t . w i d t h = 0 . 3 5 , y . i n t e r s p = 1 . 5 )
Таким образом, в результате этого этапа анализа установлено, что системы для детекции РНК А и РНК Б обеспечивают линейную зависимость значений Cq от логарифма концентрации РНК в диапазонах концентраций 102...109 и 104...109 молекул в реакции обратной транскрипции соответственно. При этом коэффициенты детерминации систем больше 0.99, а эффективности реакций амплификации лежат в диапазоне 90–110 %. Таким образом, в указанных диапазонах концентраций обе системы пригодны для измерения количества копий РНК в биологических образцах. Кроме этого, построенный калибровочный график для РНК Б будет использован далее для определения абсолютных уровней РНК Б в экспериментальных образцах.
Исключение из анализа значений, выходящих за пределы линейно-
сти системы детекции РНК Б. Для этого возьмем переменную, содержащую значения Cq для стандартных разведений РНК Б, которые еще принадлежат линейному диапазону:
> c q B
Определим максимальное значение этой переменной, которое и есть граница линейного участка:
> m a x B = m a x ( c q B )
Исключим из анализа значения Cq, выходящие за пределы линейного участка (присвоим им значение NA):
>g e n e B _ d o [ g e n e B _ d o > m a x B ] = N A
>g e n e B _ p o s [ g e n e B _ p o s > m a x B ] = N A
59
3.4.2. Характеристика референсной РНК
Проверка условия равенства эффективностей амплификации РНК А и исследуемой РНК Б. Используем только те значения концентрации, для которых сохраняется линейность калибровочного графика РНК Б, поэтому удалим значения, соответствующие образцам с двумя минимальными концентрациями:
> s = d o p [ - 1 : - 4 , ]
Найдем значения разности между соответствующими Cq РНК А и РНК Б для каждого значения концентрации:
> r = s [ , 3 ] – s [ , 2 ]
Получим модель линейной регрессии для зависимости между разностями Cq и десятичным логарифмом концентрации РНК в образцах и присвоим параметры полученной модели переменной m:
> m = l m ( r ~ l o g 1 0 ( s [ , 1 ] ) )
Определим параметры линейной модели:
> |
Y _ i n t = c o e f ( m ) [ [ 1 ] ] ; Y _ i n t |
|
[ 1 ] |
5 . 2 4 |
|
> |
S l o p e = c o e f ( m ) [ [ 2 ] ] ; S l o p e |
|
[ 1 ] |
- 0 . 0 6 |
|
Построим соответствующий график и нанесем на него уравнение модели линейной регрессии (рис. 3.3):
> p l o t ( l o g 1 0 ( s [ , 1 ] ) , r , x l i m = c ( 3 . 5 , 9 . 5 ) , y l i m = c ( 4 , 6 ) , y a x t = " n " , x l a b = " l o g 1 0 [ РНК] " , y l a b = " Разность C q " , p c h = 1 9 )
> a x i s ( s i d e = 2 , a t = c ( 4 , 5 , 6 ) )
> l i n e s ( l o g 1 0 ( s [ , 1 ] ) , c o l = " b l a c k " , m $ f i t t e d , l w d = 1 )
> l e g e n d ( " t o p l e f " , c ( " Линия регрессии" , " y = - 0 . 0 6 * x + 5 . 2 4 " ) , l t y = c ( 1 , 1 ) , c o l = c ( 1 , 0 ) , c e x = 0 . 8 , b t y = ' n ' ,
y . i n t e r s p = 1 . 3 )
Результаты расчетов показывают, что абсолютное значение наклона линии регрессии меньше 0.1. Таким образом, эффективности амплификации РНК А и Б можно признать равными.
60
