
Алексеева, Бруснигина (2014)_МПС
.pdf
www.medial-journal.ru
АНАЛИТИЧЕСКИЕ ОБЗОРЫ
УДК 578:616.9-036.22-07
ВОЗМОЖНОСТИ И ПЕРСПЕКТИВЫ ПРИМЕНЕНИЯ МЕТОДОВ МАССИВНОГО ПАРАЛЛЕЛЬНОГО СЕКВЕНИРОВАНИЯ В ДИАГНОСТИКЕ И ЭПИДЕМИОЛОГИЧЕСКОМ НАДЗОРЕ ЗА ИНФЕКЦИОННЫМИ ЗАБОЛЕВАНИЯМИ
А.Е. Алексеева, Н.Ф. Бруснигина,
ФБУН «Нижегородский научно-исследовательский институт эпидемиологии и микробиологии
им. академика И.Н. Блохиной»
Алексеева Анна Евгеньевна – e-mail: alanyutka@yandex.ru
ВВЕДЕНИЕ
В настоящее время все большее значение в лабораторной диагностике и эпидемиологическом надзоре за инфекционными заболеваниями придается молекулярногенетическим методам исследования. Особое место занимают методы, основанные на секвенировании нуклеиновых кислот (мультилокусное секвенирование (MLST), мультилокусный анализ областей генома с вариабельным числом тандемных повторов (MLVA), полногеномное секвенирование), позволяющие выявить и описать генетическую структуру новых, различия в геноме уже известных инфекционных агентов, осуществлять мониторинг генетической вариабельности патогенов, их распространенности и происхождения [1, 2, 3, 4, 5, 6, 7, 8, 9, 10,].
Массивное параллельное секвенирование, иначе называемое секвенированием следующего поколения «nextgeneration sequencing (NGS)», относится к методам, которые появились в последнее десятилетие. Ранее для секвенирования использовался метод терминации растущей
|
цепи, разработанный Фредериком Сэнгером [11]. |
можности использования различных платформ NGS как в |
|
Секвенирующие платформы, использующие метод |
научных исследованиях, так и в повседневной практике |
|
|
|
6 |
№ 2 (12) май 2014 |
|

лечебных учреждений для решения проблем диагностики инфекционных заболеваний с использованием метагеномного подхода [24, 25, 26, 27, 28, 29], мониторинга мутаций, приводящих к лекарственной устойчивости вирусов и бактерий [9, 10, 30, 31]. Имеется опыт использования полногеномного секвенирования в эпидемиологическом расследовании вспышек холеры, туберкулеза и эшерихиоза (диареи с гемолитико-уремическим синдромом), обусловленного энтерогеморрагическим штаммом E. coli O104:H4 [21, 23, 32]. Представлены результаты использования платформ NGS в метагеномных исследованиях различных биотопов, позволяющих охарактеризовать микробиом и виром человека [33, 34, 35, 36, 37, 38, 39, 40, 41, 42]. Исследования микробиома человека во всем мире координируются главным международным объединением – International Human Microbiome Consortium (http://www.human-microbiome.org).
Опыт отечественных исследователей в использовании платформ NGS для изучения возбудителей инфекционных заболеваний остается достаточно незначительным, о чем свидетельствует ограниченное количество публикаций, посвященных в основном изучению генома человека, растений и животных.
Основными точками приложения методов NGS в области микробиологии, вирусологии и эпидемиологии являются:
•открытие новых бактерий и вирусов с использованием метагеномных подходов;
•изучение микробных сообществ различных биотопов тела здорового человека и в состоянии болезни;
•анализ вариабельности геномов возбудителей инфекционных заболеваний.
ГЛАВА 1. МАССИВНОЕ ПАРАЛЛЕЛЬНОЕ СЕКВЕНИРОВАНИЕ (NGS)
Секвенирование нуклеиновых кислот – метод определения нуклеотидной последовательности, позволяющий получить описание первичной структуры линейной макромолекулы в виде последовательности мономеров (нуклеотидов). Первые подходы для проведения секвенирования были разработаны Эдманом, затем Максамом, Гилбертом [43] и Сэнгером [11]. Наибольшее распространение получил метод Сэнгера, который относится к секвенированию первого поколения и считается «золотым стандартом», поскольку позволяет определять нуклеотидную последовательность исследуемой ДНК с высокой точностью [13, 15, 16]. В основе метода лежит терминирование синтеза цепи ДНК ДНК-полимеразой с помощью дидезоксирибонуклеозид трифосфатами. Полученные фрагменты ДНК разделяют электрофорезом в полиакриламидном геле по величине фрагмента [11]. К настоящему времени разработаны автоматические капиллярные секвенаторы
(«GE – MegaBACE», «Beckman Coulter – CEQ», «Applied Biosystems»), использующие метод Сэнгера. Наибольшей популярностью пользуются приборы производителя «Applied Biosystems». Их последние модели с 48 и 96 капиллярами позволяют за 2 ч прочитать до 1100 нуклеотидов каждого образца, что суммарно составляет около 5 Мб/день (www.appliedbiosystems.com.). Широкое распространение получило использование флуоресцентных меток, каждая из которых соответствует одному из четырех нуклеотидов, что позволяет проводить детекцию результатов реакции с помощью лазера [12, 13, 14, 15, 18].
Международный проект «Геном Человека» (Human Genome Project – HGP) позволил разработать новый способ определения более длинных последовательностей ДНК, названный «shotgun-sequencing» (метод «дробовика»), при котором геномная ДНК энзиматическим или химическим способом гидролизуется на короткие фрагменты, клонируемые и используемые в дальнейшем для определения нуклеотидной последовательности методом Сэнгера. Полную последовательность исследуемого фрагмента ДНК определяют путем выравнивания и объединения полученных нуклеотидных последовательностей за счет их частичного перекрывания. Этот способ позволил впервые определить полную нуклеотидную последовательность генома человека. Метод shotgun-sequencing послужил основой для массивного параллельного секвенирования, используемого в NGS [16].
Массивное параллельное секвенирование или высокопроизводительное параллельное секвенирование (NGS) является новым этапом в совершенствовании технологий определения нуклеотидных последовательностей. Принципиальное отличие технологий NGS состоит в возможности параллельного определения нуклеотидных последовательностей множества различных нитей ДНК и чтения миллиардов нуклеотидов в день [14, 15, 16]. На платформах NGS проводят одновременное секвенирование пулированных нуклеиновых кислот, выделенных из большого количества различных образцов. Для дифференцировки исследуемых образцов используют наборы штрих-кодов или индексов (до 96 вариантов), представляющих собой олигонуклеотиды известной последовательности [12, 14, 16].
Процесс секвенирования на платформах NGS состоит из нескольких этапов (рис. 1). На первом этапе осуществляют процесс подготовки библиотеки ДНК, который включает фрагментирование ДНК ферментативно или с помощью ультразвука с последующим присоединением к полученным фрагментам ДНК универсальных олигонуклеотидных адаптеров известной последовательности и индексов с помощьюполимеразнойцепнойреакции(ПЦР).Адаптеры необходимы для дальнейшей амплификации фрагментов.
7 |
№ 2 (12) май 2014 |
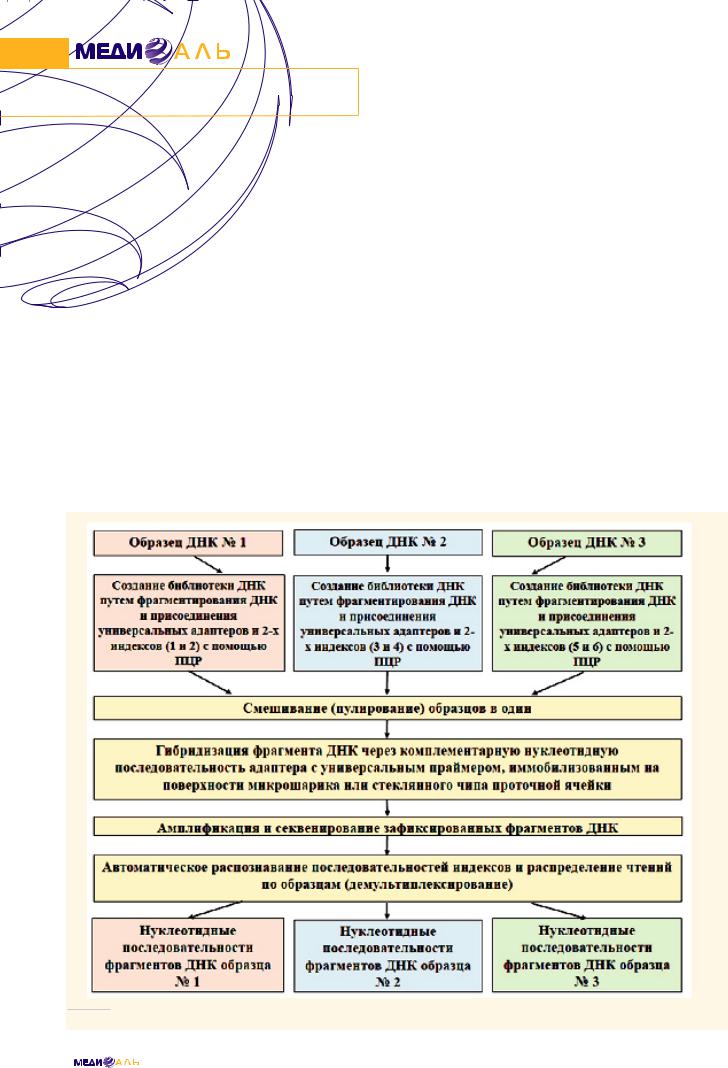
Второй этап заключается в проведении амплификации каждого фрагмента ДНК методом ПЦР. Фрагмент ДНК с помощью последовательности адаптера гибридизуется с одним или двумя праймерами, иммобилизованными на твердой поверхности (микрошарик или стеклянный чип) и участвующими в ПЦР. Через чип (проточная ячейка) пропускается реакционная смесь, содержащая набор ферментов для секвенирования. Далее происходит автоматическое пошаговое считывание каждого типа нуклеотида и детекция результата [12, 15, 18].
1.1. Технологии NGS
В настоящее время на мировом рынке представлены различные платформы NGS производителей Roche (Швейцария), Illumina (США), Life Technologies (США), которые используют следующие подходы: секвенирование путем синтеза (Sequencing by Synthesis), секвенирование путем лигирования (Sequencing by Oligonucleotide Ligation and Detection).
1.1.1. Секвенирование путем синтеза
Секвенирование путем синтеза применяется на платформах, выпускаемых фирмами Roche (Швейцария)
(http://www.454.com/), Illumina (США) (http://www. illumina.com), Ion Torrent/ Life Technologies (США) (http:// www.iontorrent.com/). Различия между этими платформами заключаются в подходах, используемых для детекции определенного нуклеотида, который присоединяется ДНК-полимеразой к растущей цепи ДНК, и способах получения разобщенных фрагментов ДНК.
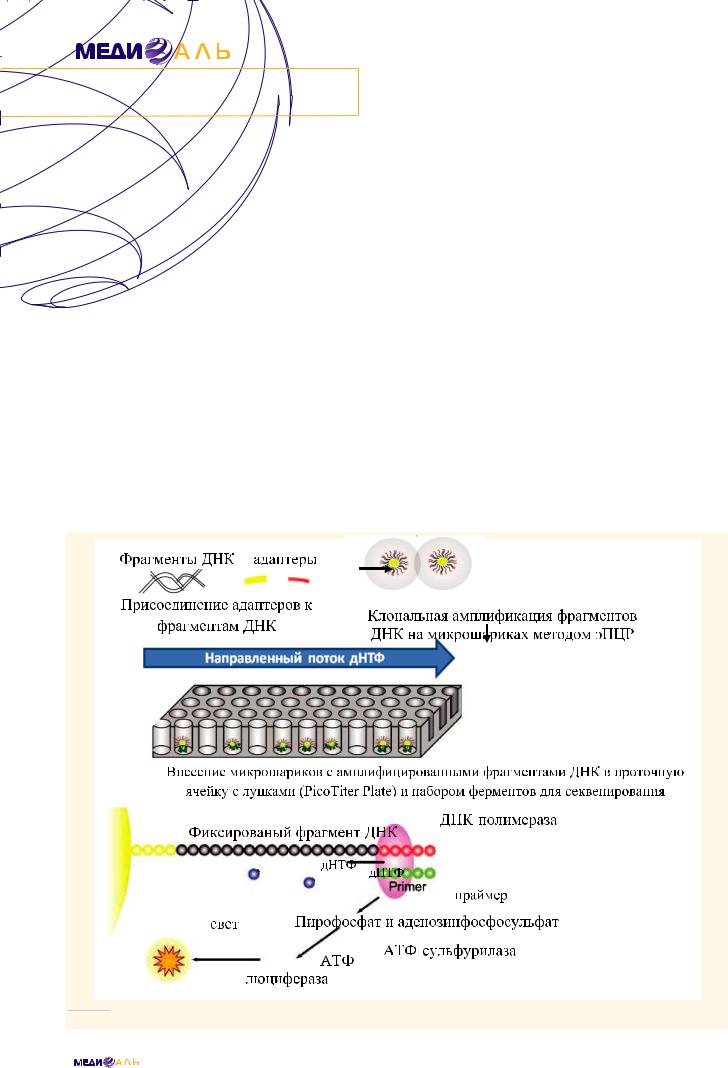
Пиросеквенирование. Принцип технологии основан на детекции хемилюминесцентного сигнала, полученного в процессе синтеза комплементарной цепи ДНК.
Одноцепочечные фрагменты ДНК гибридизуют с праймером и инкубируют с ферментами ДНК-полимеразой, АТФ-сульфурилазой, люциферазой и апиразой, а также с субстратами аденозин-5'-фосфосульфатами и люциферином. При добавлении дезоксирибонуклеозидтрифосфатов (дНТФ) ДНК-полимераза встраивает соответствующий дезоксинуклеотид, в результате чего происходит высвобождение пирофосфата. Световой сигнал формируется в результате каскадного превращения дНТФ® пирофосфат®АТФ®свет, интенсивность люминесценции пропорциональна количеству встроившихся нуклеотидов.
РИС. 1.
Этапы секвенирования на платформах NGS.
8 |
№ 2 (12) май 2014 |
|
|
|
|
|
|
|
|
|
Праймер, с которым происходит гибридизация исследуе- |
ностью люминесцентного сигнала при одновременном |
|||||||
мой нити ДНК, присоединен к отдельному микрошарику. |
встраивании в цепь ДНК большого числа дНТФ одного |
|||||||
Для физической изоляции микрошариков друг от друга |
типа [12, 13, 16, 18]. |
|
|
|||||
используется эмульсионная полимеразная цепная реак- |
|
Флуоресцентная |
технология |
секвенирования. |
||||
ция (эПЦР). На поверхности каждого микрошарика |
Технология основана на детекции флуоресцентного сиг- |
|||||||
амплифицируется только один фрагмент ДНК. |
нала, полученного при встраивании ДНК-полимеразой в |
|||||||
Микрошарики затем помещаются в фиксированное коли- |
растущую цепь ДНК одного из четырех типов дНТФ, отме- |
|||||||
чество лунок на поверхности проточного чипа (рис. 2) [12, |
ченных соответствующим флуорофором. Наличие флуо- |
|||||||
13, 14, 16]. |
ресцентной метки, с одной стороны, не позволяет встраи- |
|||||||
|
Данный подход используется на платформах 454 |
ваться следующему нуклеотиду, а с другой, дает возмож- |
||||||
Genome Sequencer (GS) 20 и 454 Genome Sequencer FLX |
ность идентифицировать нуклеотид по флуоресценции, |
|||||||
titanium, 454 Genome Sequencer Junior (Roche). 454 GS 20 |
которая соответствует конкретному кластеру на поверх- |
|||||||
является первой коммерческой NGS платформой, запу- |
ности чипа. После детекции сигналов происходит обра- |
|||||||
щенной в 2004 г. Первичная длина чтений пиросеквенато- |
ботка реагентами, которые удаляют флуоресцентные |
|||||||
ра 454 GS 20 составляла 100 нуклеотидов, в настоящее |
метки, позволяя начать новый цикл чтения. Фрагменты |
|||||||
время – 400 нуклеотидов [16]. Максимально возможный |
ДНК с присоединенными адаптерами гибридизуются с |
|||||||
размер нуклеотидных последовательностей для новых |
универсальными праймерами, иммобилизованными на |
|||||||
моделей 454 пиросеквенаторов составляет около 700 |
поверхности стеклянного чипа в виде плотного «газона». |
|||||||
нуклеотидов, что является наиболее длинными среди всех |
Стеклянный чип называется проточной ячейкой, где про- |
|||||||
коротких чтений, осуществляемых NGS технологиями |
исходят оба процесса: амплификация и секвенирование. |
|||||||
(www.454.com). К недостаткам платформ 454 относятся |
Кластерная амплификация проводится методом «bridge |
|||||||
проблемы с чтением гомоповторов, связанных с нелиней- |
amplification» (амплификация мостом) с использованием |
|||||||
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
РИС. 2.
Схема пиросеквенирования [13].
9 |
№ 2 (12) май 2014 |
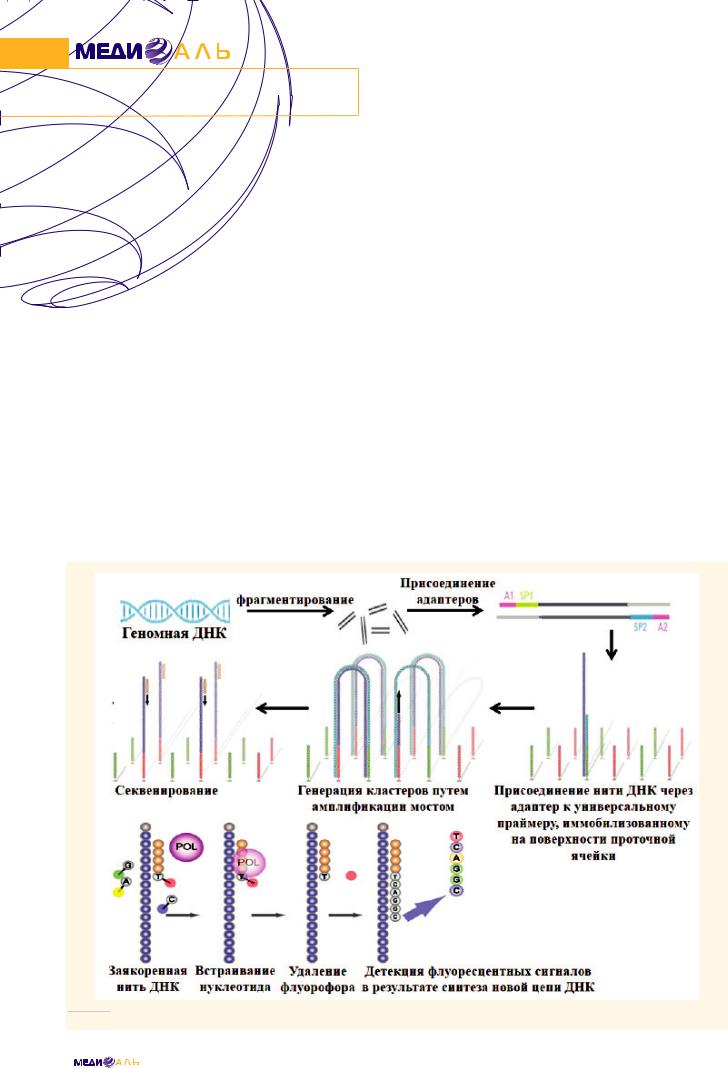
универсального праймера, специфичного последовательности адаптера. В результате амплификации на поверхности чипа формируются кластеры из фрагментов ДНК (рис. 3).
Данная технология используется на платформах, выпускаемых компанией Illumina/Solexa (США). Genome Analyzer IIX (Solexa) был второй платформой NGS, вышедшей на рынок [12, 14, 15, 16]. В 2011 году был выпущен секвенатор HiSeq 2000 Genome Analyzer (Illumina), который способен производить отдельные чтения длиной в 100 нуклеотидов и генерировать до 600 Гб данных в виде коротких последовательностей за пробег. Уровень достоверности больше, чем 99,95%. Данный прибор является наиболее производительным среди платформ NGS. В конце 2011 года был также выпущен компактный настольный секвенатор MiSeq. Инструмент очень легок в обращении, снабжен сенсорной панелью с интуитивно понятным интерфейсом и пошаговой инструкцией. Реагенты раскапаны в едином картридже, что избавляет от необходимости подготовки реакционных смесей. В приборе автоматизированы все стадии секвенирования, начиная от генерации кластеров и заканчивая компьютерным анализом данных. MiSeq способен генерировать чтения длиной в
250 нуклеотидов в количестве до 8,5 Гб/день. С целью ускорения и упрощения процесса анализа результатов исследователями компанией Illumina для своих платформ (MiSeq и HiSeq) разработано программное обеспечение BaseSpace, позволяющее хранить и обрабатывать полученные результаты в так называемом цифровом облаке, то есть полученные данные можно хранить и обрабатывать прямо в сети Интернет. Перенос данных секвенирования на BaseSpace происходит автоматически, полученные данные могут быть доступны для всех соисполнителей в любое время. BaseSpace уже включает все необходимые инструменты для анализа последовательностей после выравнивания и объединения (www.Illumina.com.).
Полупроводниковая технология секвенирования Принцип технологии основан на детекции изменения
рН при выделении одного протона водорода в результате встраивания дНТФ ДНК-полимеразой в реальном времени. Этапы подготовки библиотеки также включают в себя фрагментирование ДНК, присоединение адаптеров для дальнейшей амплификации на микрошариках с использованием эПЦР. Носителями микроклонов служат микрошарики размером около 3 мкм, распределяемые по лункам рН-сенсорного чипа (рис. 4) [14, 44].
РИС. 3.
Схема флуоресцентной технологии секвенирования [13].
10 |
№ 2 (12) май 2014 |

1.1.2. Секвенирование путем лигирования
Отличительной особенностью секвенирования нуклеиновых кислот путем лигирования является использование ДНК-лигазы. ДНК-лигаза – фермент, образующий ковалентную связь между 5'-фосфатом и 3'-гидроксилом в одноцепочечном разрыве ДНК-дуплекса. Подобная технология используется платформами ABI 5500/5500xl SOLiD, ABI SOLiD4, ABI 5500W/Wxl SOLiD (http://www. appliedbiosystems.com). Все иммобилизованные на поверхности микрошариков одноцепочечные фрагменты ДНК первоначально формируют комплементарный комплекс с универсальным адаптером. Для чтения нуклеотидной последовательности используется набор олигонуклеотидов следующего вида: 3'-XYNNNZZZ-F-5', где XY – один из 16 возможных динуклеотидов; N – любой нуклеотид (вырожденная буква); Z – универсальное основание; F – один из четырех флуорофоров. Один флуорофор соответствует четырем различным динуклеотидам (XY, X'Y', YX, Y'X', где X < > Y и отдельная группа XX, X'X'), например AG, TC, GA и CT. Из добавленного набора зондов с ДНК гибридизуется олигонуклеотид, содержащий комплементарные димер (XY) и тример (NNN). ДНК-
лигаза формирует фосфодиэфирную связь между универсальным праймером и комплементарным зондом. Далее происходит регистрация флуоресцентных сигналов и соотнесение их с пространственным расположением иммобилизованных на чипе микрошариков. Для начала нового шага удаляются три нуклеотида и флуорофор с 5'-конца (ZZZ-F). После нескольких этапов лигирования проводится денатурация и удаление комплементарной цепи с микрошариков, что позволяет начать новый цикл секвенирования с использованием адаптера, смещенного на один нуклеотид (рис. 5) [12, 13, 18].
В настоящее время активно продолжается совершенствование платформ, использующих NGS технологии, с целью увеличения производительности и снижения стоимости расходных материалов. Основные технические характеристики платформ NGS, существующих на мировом рынке в настоящее время, представлены в таблице 1.
1.2. Анализ данных, полученных в результате секвенирования на платформах NGS
Платформы NGS обеспечивают определение нуклеотидных последовательностей длиной в среднем 50–500 нуклеотидов, что намного короче, чем получаемые путем
РИС. 4.
Схема полупроводниковой технологии секвенирования (http://bioinformatics.ru/Misc/genseq-roadmap.html).
11 |
№ 2 (12) май 2014 |
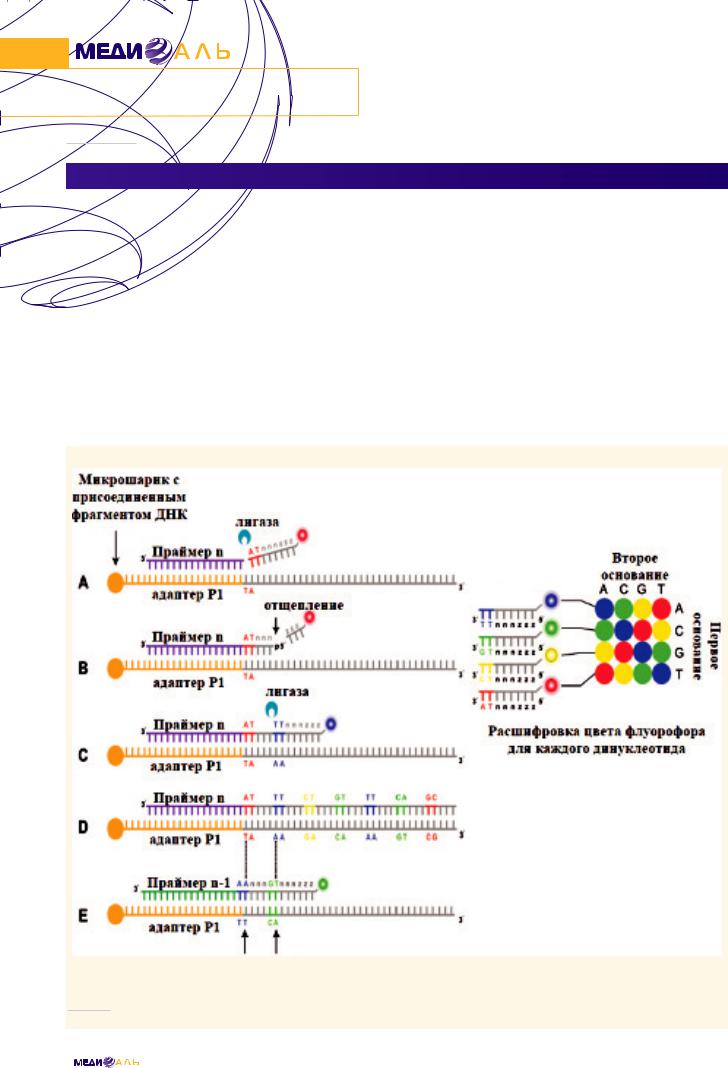
ТАБЛИЦА 1.
Технические характеристики основных платформ NGS
Производитель |
Roch |
Illumina (Solexa) |
Life Technologies |
Life Technologies |
|
(Applied Biosystems) |
(Ion Torrent) |
||||
|
|
|
|||
Адрес сайта |
http://www.454.com/ |
https://www.illumina.com/ |
http://www.appliedbiosystems. |
http://www.iontorrent.com/ |
|
com/ |
|||||
|
|
|
|
||
|
GS FLX Titanium |
GA IIX,HiScanSQ, |
ABI 5500xl SOLiD, |
|
|
Платформы |
HiSeq1000/2000,MiSeq, |
ABI 5500 SOLiD, |
Ion PGM, Ion Proton |
||
GS Junior |
|||||
|
HiSeq1500/2500 |
ABI 5500W/Wxl SOLiD |
|
||
|
|
|
|||
Амплификация |
Клональная эПЦР |
Клональная амплификация |
Клональная эПЦР на |
Клональная эПЦР |
|
на поверхности микрошариков |
«мостом» на поверхности чипа |
поверхности микрошариков |
на поверхности микрошариков |
||
|
|||||
|
Регистрация хемилюминесцен- |
Регистрация флуоресцентного |
Регистрация флуоресцентного |
Регистрация изменения |
|
Метод детекции |
ции в результате образования |
сигнала при встраивании |
сигнала при лигировании |
рН в результате освобождения |
|
|
пирофосфата |
меченных нуклеотидов |
меченных олигонуклеотидов |
протонов водорода |
|
Длина чтений |
400-700 нуклеотидов |
36, 100, 150 и 250 нуклеотидов |
до 75 нуклеотидов |
35-400 нуклеотидов |
|
Максимальное |
До 132 |
до 96 (MiSeq, HiSeq1000, |
до 1152 |
До 384 |
|
количество образцов |
HiScanSQ) до 192 (HiSeq2000) |
||||
|
|
|
|||
|
|
1,5-11 дней (GA IIX, HiScanSQ, |
|
|
|
Время секвенирования |
От 10 до 23 часов |
HiSeq1000/2000), |
1-7 дней |
2-4 часа |
|
|
|
4-49 часов (MiSeq) |
|
|
|
Производительность за прогон |
До 1Гб |
от 8,5Гб (MiSeq) |
от 90Гб (ABI 5500/5500xl SOLiD) |
от 20Гб (Ion PGM) |
|
до 600Гб (HiSeq1000/2000) |
до 300Гб (ABI 5500W/Wxl SOLiD) |
до 100Гб (Ion Proton) |
|||
|
|
||||
Уровень достоверности |
99,99% |
99,95% |
99,99% |
99% |
РИС. 5.
Схема секвенирования путем лигирования [13].
12 |
№ 2 (12) май 2014 |
секвенирования по Сэнгеру. В связи с этим, очень важным параметром секвенирования является степень покрытия (coverage) короткими чтениями. Покрытие определяется числом чтений, перекрывающих друг друга внутри определенного региона генома. Например, 30-кратное покрытие гена CYP2D6 означает, что каждый нуклеотид внутри этого гена представлен минимально в 30 разных и перекрывающихся коротких чтениях. Значительный показатель покрытия является необходимым условием для достоверного построения последовательности генома. Таким образом, в результате секвенирования на платформах NGS создается огромный массив данных и возникает необходимость в автоматизации процесса обработки с помощью вычислительной техники. Разработка программного обеспечения для оценки качества секвенирования, выравнивания, объединения и дальнейшей обработки данных осуществляется с помощью биоинформатики, находящейся на стыке двух наук: биологии и информатики [16, 45, 46, 47, 48, 49, 50].
Процесс обработки данных включает следующие основные шаги: фильтрация последовательностей и коррекция ошибок, выравнивание и объединение, анализ результатов [3, 50]. На этапе фильтрации из набора данных исключаются последовательности низкого качества. Для оценки качества секвенирования разработан показатель Phred Quality Scores (Q), который связан логарифмически с
вероятностью P ошибочного определения нуклеотида: Q= __10 lgP
В таблице 2 представлены варианты значений показателя Q, вероятности некорректного определения нуклеотида и достоверности секвенирования.
ТАБЛИЦА 2.
Варианты значений показателя Q, вероятности некорректного определения нуклеотида и достоверности секвенирования
Phred Quality |
Вероятность некорректного |
Достоверность |
|
Score Q |
определения нуклеотида |
||
|
|||
10 |
1 из 10 |
90% |
|
20 |
1 из 100 |
99% |
|
30 |
1 из 1000 |
99.9% |
|
40 |
1 из 10000 |
99.99% |
|
50 |
1 из 100000 |
99.999% |
Значение Q10 показывает, что вероятность некорректного определения нуклеотида составляет 1 из 10 (достоверность 90%), если Q20, то вероятность 1 из 100 (достоверность 99%), Q30 – один неправильный нуклеотид из 1000 нуклеотидов (достоверность 99,9%) и т. д. Показатель Q20 является допустимым или пороговым уровнем для определения достоверности результатов.
Для различных платформ NGS характерны различные типы ошибки. Например, платформы 454 Roch осуществляют чтения с ошибками в виде инсерций и делеций, в
то время как платформы SOLiD и Illumina склонны к ошибкам в виде замен [51]. Исправление ошибок помогает достичь высокого качества полученных нуклеотидных последовательностей, что значительно сокращает дальнейшие алгоритмы объединения. Разработаны различные программы для коррекции ошибок секвенирования и фильтрации чтений низкого качества. В 2011 году L. Salmela и J. Schrоder создали программу Coral (CORrection with Alignments) для коррекции ошибок коротких чтений, осуществляемых различными секвенирующими платформами, которая легко адаптируется к определенным (разным) моделям ошибок [51]. Р. Skums et al. (2012) предложили две новые программы: k-mer- based error correction (KEC) и empirical frequency threshold (ET) для коррекции ошибок при секвенировании вирусных геномов [49].
Второй этап – выравнивание и объединение (aligment and assembly). В процессе выравнивания осуществляется сопоставление нуклеотидных последовательностей с целью обнаружения совпадающих участков [52, 53].
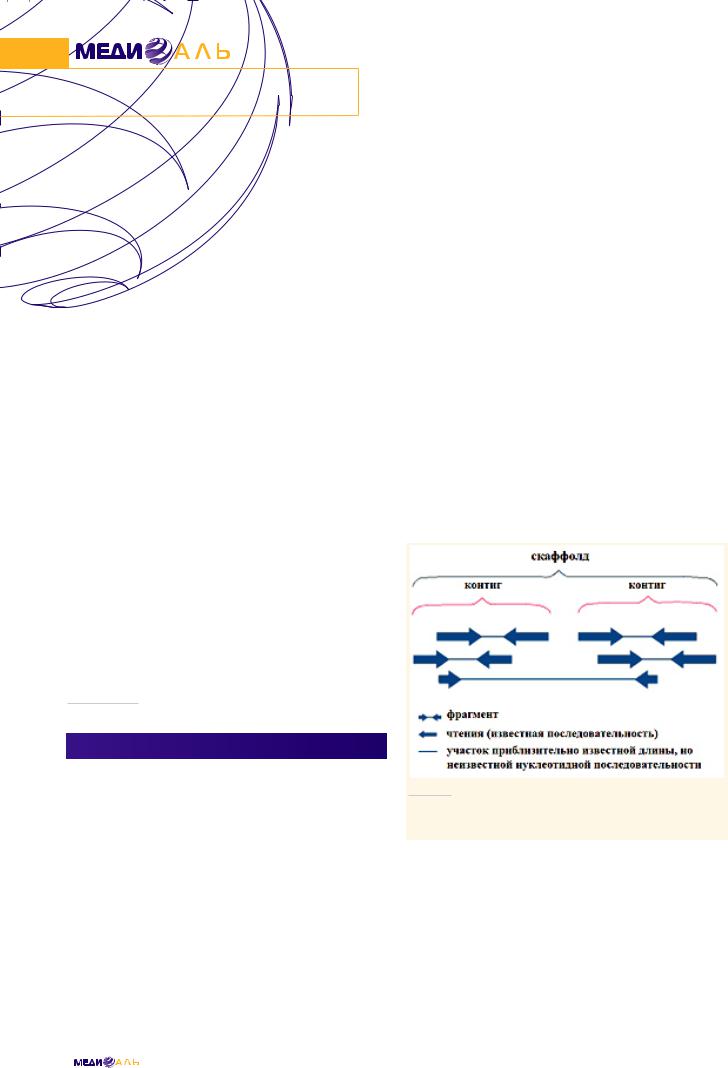
После выравнивания происходит непосредственно сборка коротких нуклеотидных цепочек в одну или несколько длинных последовательностей (рис. 6).
РИС. 6.
Принцип объединения чтений в контиги и скаффолды (http:// commons.wikimedia.org/wiki/File:PET_contig_scaffold. png?uselang=ru).
Сначала формируются более длинные контиги, представляющие собой набор перекрывающихся фрагментов ДНК, которые в совокупности представляют собой консенсусную область ДНК. Контиги с промежутками известной длины объединяются в скаффолды.
Существует множество программ для объединения и сборки генома, большинство используют алгоритмы, основанные:
• на перекрытии (overlap-layout-consensus) и применяются для длинных фрагментов;
13 |
№ 2 (12) май 2014 |

• на графах де Брейна (de Bruijn Graph) и применяются для коротких фрагментов, получаемых при секвенировании на платформах NGS [53].
Процесс выравнивания и объединения может осуществляться с использованием референсного (проверочного) генома или референсных нуклеотидных последовательностей и de novo (заново).
В случае, если исследуемый микроорганизм известен, то возможно использование референсного генома близкородственного микроорганизма. В этом случае определяется наиболее вероятная позиция полученных коротких чтений на референсном геноме. Для выравнивания и объединения коротких чтений по отношению к референсному геному используются такие программы, как Clustal, MAQ (Mapping and Assembly with Quality) [54], ELAND (Efficient Large-Scale Alignment of Nucleotide Databases) (www. illumina.com), BLAST (Basic Local Alignment Search Tool) [33], SOAP (Short Oligonucleotide Alignment Program) [56], SeqMap [57], MUSCLE [58], MAFFT [59] и др.
По сравнению со сборкой коротких последовательностей на основе референсной последовательности объединение de novo является более сложным процессом. Построение de novo представляет собой реконструкцию в чистой форме, без сопоставления с ранее полученными последовательностями геномов или транскриптов. Короткие чтения дают меньше информации, создавая трудности при объединении последовательностей в одну хромосому [53]. В настоящее время построение de novo на основе данных, полученных на платформах NGS, ограничивается, как правило, проектами по изучению микробного генома в связи с малыми размерами бактериальных хромосом [60, 61]. Для объединения нуклеотидных последовательностей de novo используются такие программные обеспечения, как SOAP de novo [62], Velvet [52], Euler [46] и др.
Третий этап обработки данных секвенирования – непосредственный анализ набора последовательностей ДНК, полученного после объединения. В метагеномных исследованиях на заключительном этапе определяется таксономическая принадлежность (taxonomic assignment) микроорганизмов, присутствующих в образце. В основе анализа набора полученных нуклеотидных последовательностей лежит определение филогенетических связей с уже известными нуклеотидными последовательностями целого генома или участками генома (например, гена 16S рРНК) микроорганизмов, относящихся к различным таксонам [17, 45]. Для этих целей используется программное обеспечение RITA, UniFrac, Naïve Bayes, BLAST, CARMA. TreePhyler, MetaDomain, Markov model, MEGAN, TOCOA и др. Источниками нуклеотидных последовательностей служат различные базы данных: GenBank (www.ncbi.nlm.nih.
gov/genbank/), DNA Data Bank of Japan (www.ddbj.nig. ac.jp), GreenGens (www.greengenes.secondgenome.com), Genomes Online Database (www.genomesonline.org), Ribosomal Database Project (www.rdp.cme.msu.edu), SILVA (www.arb-silva.de) и др. [13, 15, 16, 61, 63, 64].
Для обработки результатов секвенирования гена 16SрРНК в метагеномных исследованиях J.G. Caporaso et al. (2010) создано программное обеспечение QIIME (quantitative insights into microbial ecology). Данная программа является комплексной и позволяет проводить полный анализ результатов секвенирования, включая фильтрацию неправильных чтений, коррекцию ошибок, интерпретацию полученных последовательностей в соответствии с базами данных [17, 47].
При анализе результатов секвенирования проводятся множественные сравнения полученных нуклеотидных последовательностей с целью выявления внутривидовой изменчивости микроорганизма в виде однонуклеотидных полиморфизмов (single nucleotide polymorphism – SNP), инсерций, делеций и т. д. Подобный анализ актуален при идентификации возбудителя инфекционного заболевания, установления его филогенетических связей, а также для определения ареала распространения. При сравнении очень близких (схожих) нуклеотидных последовательностей, например, при определении междуили внутриштаммовых различий, наиболее достоверные результаты дают программы, основанные на подходах maximum likelihood (наибольшего подобия) или maximum parsimony (максимальной экономии) [65]. Данные подходы были успешно применены в исследованиях C.U. Koser et al. (2012), D.W. Eyre et al. (2012) для дифференциации штаммов MRSA [31, 22]. Подход maximum likelihood послужил основой для обновления программного обеспечения MEGA (Molecular Evolutionary Genetics Analysis) – MEGA5, являющегося наиболее популярным в исследованиях, посвященных изучению филогенетических связей и построению филогенетического дерева исследуемых микроорганизмов [48].
ГЛАВА 2. ПРИМЕНЕНИЕ ТЕХНОЛОГИЙ NGS
В ДИАГНОСТИКЕ И ЭПИДЕМИОЛОГИЧЕСКОМ НАДЗОРЕ ЗА ИНФЕКЦИОННЫМИ ЗАБОЛЕВАНИЯМИ
Молекулярно-генетические методы занимают одно из важнейших мест в диагностике и эпидемиологическом надзоре за инфекционными заболеваниями, поскольку отличаются высокой чувствительностью, специфичностью, экспрессностью. Использование молекулярно-гене- тических подходов, основанных, в частности, на амплификации нуклеиновых кислот, особенно актуально и дает прекрасные результаты в случаях, когда возбудитель относится к группе труднокультивируемых или некультивируемых, а также присутствует в небольшом количестве.
14 |
№ 2 (12) май 2014 |
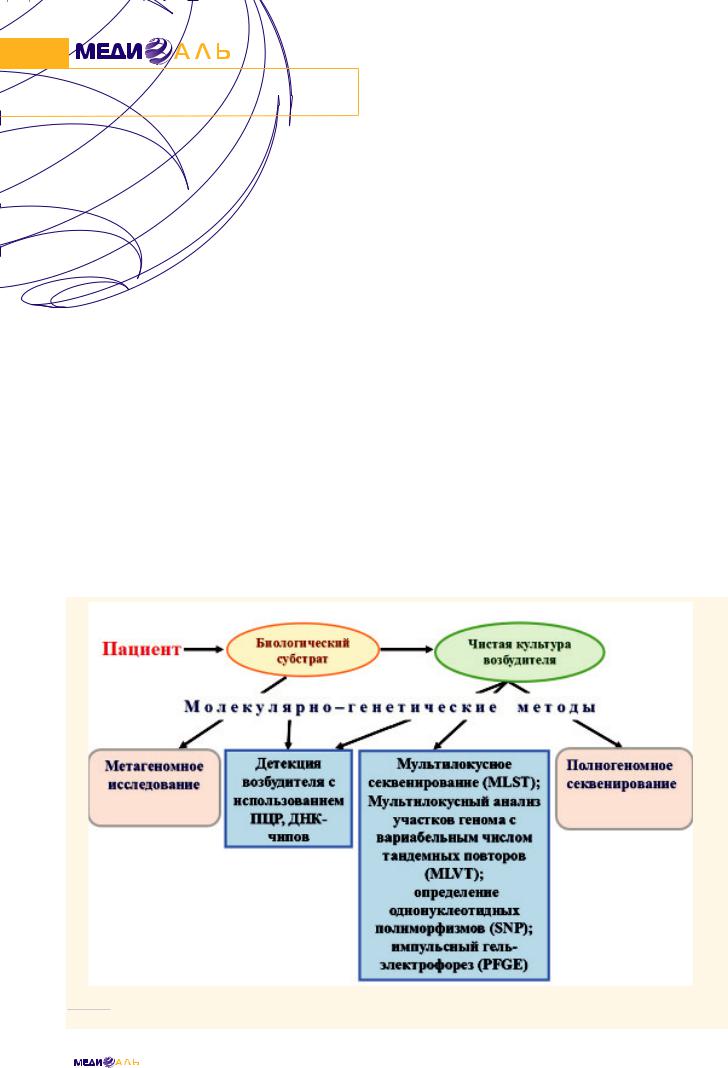
На рисунке 7 представлены основные молекулярно-гене- тические методы, используемые в диагностике и эпидемиологическом надзоре за инфекционными заболеваниями.
2.1. Метагеномные исследования
Метагеномика – это один из разделов геномики, посвященный изучению всего генетического материала (метагенома) сообществ микроорганизмов, присутствующих в исследуемом образце [3, 18, 25, 61, 66, 67, 68]. Объектами изучения метагеномики могут являться любые популяции микроорганизмов, обитающих в воде, почве, организме животного, человека или любой другой среде. Главной целью метагеномики является получение и анализ всех геномов для установления видового состава и метаболических взаимосвязей в сообществе [68]. Важной особенностью метагеномных исследований является отсутствие необходимости в выделении и культивировании микроорганизмов, что является принципиальным моментом, поскольку не все из них растут на питательных средах. Метагеномное исследование позволяет выявить в любом объекте не только широкий спектр бактерий, но также присутствие вирусов и простейших [18, 68].
При проведении метагеномных исследований используют следующие методы:
• секвенирование фрагментов ДНК, кодирующих эволюционно консервативные гены;
•секвенирование метагеномной ДНК сообщества путем
ееслучайного фрагментирования (whole-metagenome shotgun sequencing).
Одним из важнейших объектов изучения метагеномики является симбиотический микробиом человека. Изучение микробиома различных биотопов тела человека позволяет дать характеристику микробиоты здоровых лиц взрослого населения, способствует пониманию взаимоотношений между микроорганизмами, поскольку микробиом человека представляет собой не просто совокупность микроорганизмов, но сложную и многокомпонентную систему с внутренней структурой, динамикой, активно взаимодействующую с организмом хозяина. Известно, что микробиота участвует в формировании иммунной системы, развитии тканей, влияет на защитные механизмы, препятствующие проникновению патогенов. Патогенез множества заболеваний прямо или косвенно связан с ферментативной и биохимической активностью микробиоты и ее влиянием на организм человека [18, 33, 69].
Для проведения широкомасштабных дорогостоящих исследований микробиома человека ученые различных стран мира объединились в консорциумы. Крупнейшими объединениями по изучению микробиома являются европейский консорциум Metagenomics of the Human Intestinal Tract (MetaHit) и американский «Микробиом человека» (Human Microbiome Project – HMP). MetaHit основан в
РИС. 7.
Применение молекулярно-генетических методов в диагностике и эпидемиологическом надзоре за инфекционными заболеваниями [67].
15 |
№ 2 (12) май 2014 |
