
- •РЕПАРАЦИЯ ДНК
- •Видимо, уже на ранних стадиях эволюции ДНК заменила РНК в качестве носителя генетической
- •Репарация (repair) ДНК - один из общих биологических процессов, направленный на исправление ошибок
- •Часто системы репарации работают во время или сразу после репликации.
- •Причины появления повреждений в ДНК
- •Основные типы повреждения ДНК
- •Гидролиз
- •Дезаминирование
- •Чаще всего дезаминируется цитозин (в каждой клетке человека за день происходит около 100
- •Алкилирование
- •Иногда может происходить размыкание пуринового кольца.
- •Главным нарушением, возникающим под действием ультрафиолета, является насыщение двойных связей оснований и образование
- •Типы репарации:
- •Cтратегии коррекции повреждений
- •Очевидным случаем прямой репарации является сшивание
- •О6-meG образует пару с Т
- •Прямое удаление повреждений
- •Происходит необратимое ковалентное присоединение метильной или этильной группы к остатку Cys в активном
- •Лиа́зы — отдельный класс ферментов, катализирующих реакции негидролитического и неокислительного разрыва различных химических
- •Более радикальным и эффективным путем исправления нарушений нуклеотидов является
- •Последовательность событий:
- •Base excision repair (BER)
- •Как работают ДНК-гликозилазы?
- •2. После действия ДНК-N-гликозилаз в ДНК остается
- •(фосфодиэстераза отщепляет
- •3.Затем фосфодиэстераза отщепляет фосфодезоксирибозу, и в ДНК остается пробел длиной в один нуклеотид.
- •Механизм BER
- •Эксцизия нуклеотидов NER (nucleotide excision repair)
- •Последовательность событий:
- •1.UvrA и UvrB сканируют ДНК и выявляют поврежденные места
- •Узнавание и связывание
- •Вывод: эксцизионная репарация всегда использует один принцип: нарушенный участок ДНК удаляется, а затем
- •Последовательность событий:
- •Исправление ошибок репликации ( Мismatch repair )
- •Неспаренные основания ДНК могут возникать в результате трех событий:
- •Система репарации должна каким-то образом отличать друг от друга две цепи одной молекулы
- •В случае нарушений спаривания, возникающих в результате репликации или репарации, новосинтезированная цепь ДНК
- •По бактериальному геному распределены (на среднем расстоянии 256 пар оснований) короткие палиндромные последовательности
- •В бактериальной клетке за репарацию ошибочно спаренных нуклеотидов (мисметчей) отвечает система
- •Dam -зависимая система репарации mutHLSU
- •Как работает dam -зависимая система репарации?
- •У E. coli MutS сканирует ДНК. Ошибки (мисметчи) опознаются в силу того, что
- •-Белки МutН связываются с тетрануклеотидными
- •По логике событий ошибочно включенный нуклеотид должен находиться в новосинтезированной цепи ДНК. Эта
- •В эукариотических клетках также существует система коррекции ошибок репликации
- •Уэукариот механизм распознавания новосинтезированной цепи не известен.
- •2. Mut Y-зависимая система репарации
- •А если не успели все починить а ДНК уже реплицируется?
- •Репликативная машина обычно
- •Обход препятствия посредством смены матричных цепей - продолжение репликации с сохранением ошибки в
- •Репарация без репарации - SOS-репарация –
- •Таким образом,
- •Репарация двунитевых разрывов
- •С рекомбинацией
- •Процесс репарации двунитевых разрывов разделяют на три этапа.
- •Репарация двунитевого разрыва посредством гомологичной рекомбинации
- •Homologous Recombination Repairs ds Breaks
- •Non-Homologous End Joining (Double Strand Breaks)
- •Non-Homologous End Joining (Double Strand Breaks)
- •Таким образом, в процессе соединения происходит потеря нескольких пар оснований.

У E. coli MutS сканирует ДНК. Ошибки (мисметчи) опознаются в силу того, что они индуцируют нарушения в правильной структуре двойной спирали.
Найдя ошибку, MutS изменяет конформацию, что закрепляет его на цепи ДНК,
-привлекает MutL
-образуется комплекс MutS -MutL, стабилизирующий связывание, белка MutS с
неспаренным основанием
- MutL взаимодействует с двумя белками MutH

-Белки МutН связываются с тетрануклеотидными
палиндромными сайтами GATC по обе стороны от мисметча
-MutH вносит однонитевой разрыв
в неметилированной цепи ДНК в пределах одного из сайтов (один из двух сайтов избирается случайно)
-хеликаза mutU (UvrD) расплетает двойную спираль
-одна из экзонуклеаз удаляет фрагмент ДНК от разрыва и до неспаренного нуклеотида (включая последний)
-Брешь (до нескольких тысяч оснований) застраивается ДНК полимеразой III в присутствии белков SSB.
-Одноцепочечный разрыв сшивается ДНК-лигазой.
NB!
У белка MutL не обнаружено ферментативной активности, хотя он взаимодействует с MutS и необходим для активации
МutН - эндонуклеазы, осуществляющей одноцепочечный разрыв ДНК.
Таким образом, комплекс MutS-MutL, собранный на участке ДНК с ошибочно спаренным нуклеотидом, стимулирует эндонуклеазную активность MutH.
хеликаза MutU = хеликаза II = тот же белок, что и UvrD
продукт гена mutU = uvrD = uvrE = recL
В зависимости от направления деградации в сторону неспаренного основания (в зависимости от расположения GATC-сайта по отношению к корректируемому нуклеотиду) в реакции принимает участие одна из однонитевых ДНК-экзонуклеаз:
Exo I (3'-экзо), Exo Х (3'-экзо), Exo VIII (3'- или 5'-экзо) или Rec J (5'-экзо).

По логике событий ошибочно включенный нуклеотид должен находиться в новосинтезированной цепи ДНК. Эта цепь опознается благодаря отсутствию Dam метилирования. MutH связывается только с неметилированной цепью ДНК и вносит разрыв именно в эту цепь 
GATC Метилирование CTAG аденина
В эукариотических клетках также существует система коррекции ошибок репликации
Обнаружены гомологи MutS и MutL; гомолога MutH не обнаружено
гомологи MutS (MSH — MutS homolog) образуют два гетеродимерных комплекса
--MSH2-MSH6 (MutSα) узнает неспаренные нуклеотиды и короткие «инделы»
--MSH2-MSH3 (MutSβ) узнает длинные «инделы»
Гомологи бактериального белка MutS - белок GTBP человека
белок Msh6 дрожжей
Гомологи MutL - у человека MSH2-GTB
у S. cerevisiae –
гетеродимерные комплексы белков MLH
Уэукариот механизм распознавания новосинтезированной цепи не известен.
Уэукариот метилирование аденина не используется для маркировки цепей: белки эукариотической системы репарации мисметчей связанны с реплисомой и синтезирующимися цепями ДНК, т.е. срабатывают непосредственно во время репликации.
Возможно наличие разрывов в новосинтезированных цепях является опознавательным сигналом ?
2. Mut Y-зависимая система репарации
специфически ликвидирует последствия окислительных повреждений гуанина ( dGTP -> окисление -> 8-оксо-dGTP)
Если dGTP окисляется с образованием 8-оксо-dGTP, белок
MutT расщепляет последний, предотвращая его включение в ДНК.
Если же он все-таки включается напротив остатка С, то Fpg-гликозилаза (MutM) удаляет это модифицированное основание.
В том случае, когда 8-оксо-G остается в составе ДНК, в следующем раунде репликации он спаривается с А, и в итоге может произойти
трансверсия G-C -> T-A.
белок Mut Y действует - как ДНК-гликозилаза -> удаляет остаток A из некорректной пары
- как AP-лиаза вносит одноцепочечный разрыв по соседству с AP-сайтом.
Далее следуют процессы, вызываемые функционированием системы репарации BER.
Последовательность реакций с участием MutY также репарирует некомплементарные пары A-G и A-C с образованием соответственно пар
C-G и G-C.
А если не успели все починить а ДНК уже реплицируется?
Пострепликативная (рекомбинационная)
репарация
Путь осуществления репликации через тиминовый димер (или другое
препятствие) в составе матрицы получил название
пострепликативной (рекомбинационной) репарации.

Репликативная машина обычно
останавливается, встречая повреждения в составе матрицы. Возможно после остановки
брешь вилки репликация начнется снова после препятствия (с нового праймера)
Тогда напротив препятствия будет
брешь.
На место пробела путем гомологичной рекомбинации вставляется участок
сестринской молекулы ДНК. Пробел, оставшийся при этом в сестринской молекуле, легко заполняется ДНК- полимеразой.
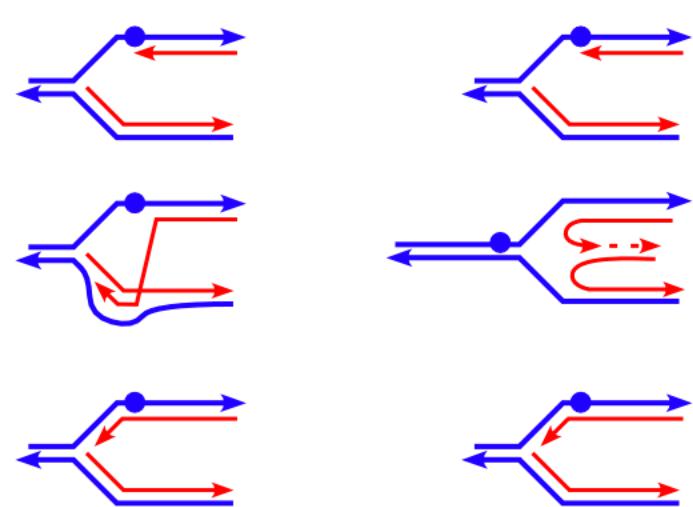
Обход препятствия посредством смены матричных цепей - продолжение репликации с сохранением ошибки в одной из родительских цепей
Вариант 1 |
Вариант 2 |
