
Лабораторная работа №3
.docxМИНИСТЕРСТВО ОБРАЗОВАНИЯ И НАУКИ КЫРГЫЗСКОЙ РЕСПУБЛИКИ
КЫРГЫЗСКИЙ ГОСУДАРСТВЕННЫЙ ТЕХНИЧЕСКИЙ
УНИВЕРСИТЕТ ИМ. И. РАЗЗАКОВА
Лабораторная работа
Бишкек 2021
Лабораторная работа №3
Оптимизация процессов с использованием генетических алгоритмов
Разработать программу, реализующую генетический алгоритм поиска максимального и минимального значений целевой функции f(x) = a + bx + cx 2 + dx 3 в интервале x = [-10, 53]. Мой вариант №7 и входные данные a = 23, b = - 80, c = - 64, d = 5.
Фото результат:
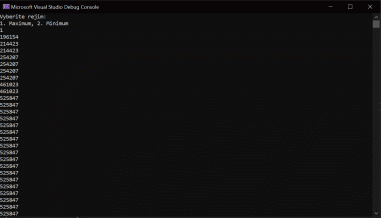
Рис 1. Нахождение максимального значения функции
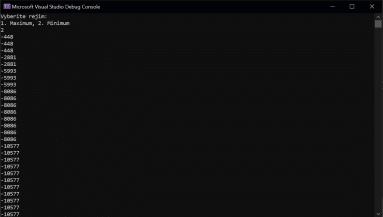
Рис 2. Нахождение миниального значения функции
Листинг кода:
#include <iostream>
#include <cstdlib>
#include <algorithm>
#include <random>
#include <cmath>
#include <bitset>
#include <clocale>
#include <Windows.h>
using namespace std;
struct Indval;
void mutate(int& fenotype_item, int negative); //Ссылка на элемент массива
void reduce(int* genotype_with_parents, size_t size_gen_par, int* genotype, size_t size_gen); //ссылка на уменьшение
void crossing(int& left, int& right, int* genotype_with_parents, size_t from, int negative, size_t range); //ссылка на пересечение
int* populate_genotype(size_t size, int from, int to); //популяция генотипа
size_t* get_random_indexes(size_t n, size_t m2); //получение случайного индекса
bool compare(const Indval& left, const Indval& right); //сравнение
double get_y(int x); //результат функции f(x)
static size_t mode = -1;
static const int staticlog = 6;
struct Indval { //индекс значения
Indval(int val, size_t index) : val_(val), index_(index) {}
int val_;
size_t index_;
};
// максимальное значение функции 525847
// минимальное значение функции -10577
int main() {
setlocale(LC_ALL, "Rus");
// 1 - max
// 2 - min
cout << "Vyberite rejim: \n" << "1. Maximum, 2. Minimum" << endl;
cin >> mode;
const size_t n = 8, m = n / 2, mut = m == 1 ? 1 : m;
const int first_elem = -10; //значение первого интервала -10 [-10, 53]
const int last_elem = 53; // значение второго интервала 53 [-10, 53]
const size_t range = std::abs(first_elem) + std::abs(last_elem) + 1;
int* genotype = populate_genotype(n, first_elem, last_elem);
std::sort(genotype, genotype + n);
size_t counter = 0;
while (counter != 1000) {
int* genotype_with_parents = new int[(n + m * 2)];
std::copy(genotype, genotype + n, genotype_with_parents);
counter++;
// Выберем случайные пары для скрещивания
size_t* indexes = new size_t[m * 2];
indexes = get_random_indexes(n, m * 2);
for (size_t i = 0; i != m * 2; i += 2) {
crossing(genotype[indexes[i]], genotype[indexes[i + 1]], genotype_with_parents, n + i, -first_elem, range);
}
size_t* indexes_to_mutate = new size_t[mut];
indexes_to_mutate = get_random_indexes(m, mut);
for (size_t i = 0; i != mut; ++i) {
mutate(genotype_with_parents[n + indexes_to_mutate[i]], -first_elem);
}
reduce(genotype_with_parents, n + m, genotype, n);
double max_or_min = genotype[0];
for (size_t i = 1; i != n; ++i) {
double val = get_y(genotype[i]);
switch (mode) {
case 1:
if (val > max_or_min)
max_or_min = val;
break;
case 2:
if (val < max_or_min)
max_or_min = val;
break;
}
}
std::cout << max_or_min << std::endl;
}
return 0;
}
double get_y(int x) {
//Варианты выполнения, мой вариант №7 a = 23, b = -80, c = -64, d = 5.
int a = 23, b = -80, c = -64, d = 5;
return a + (b * x) + (c * std::pow(x, 2)) + (d * std::pow(x, 3));
}
void reduce(int* genotype_with_parents, size_t size_gen_par, int* genotype, size_t size_gen) {
std::vector<Indval> indval_arr;
for (size_t i = 0; i != size_gen_par; ++i) {
int x = genotype_with_parents[i];
indval_arr.push_back(Indval(get_y(x), i));
}
std::sort(indval_arr.begin(), indval_arr.end(), compare);
for (size_t i = 0; i != size_gen; ++i) {
genotype[i] = genotype_with_parents[indval_arr[i].index_];
}
}
void mutate(int& fenotype_item, int negative) {
const int log = 6;
int val = fenotype_item + negative;
std::bitset<log> bitval(val);
// srand (time(NULL));
size_t pos = rand() % staticlog;
bitval.flip(pos);
fenotype_item = (int)bitval.to_ulong() - negative;
}
void crossing(int& left, int& right, int* genotype_with_parents, size_t from, int negative, size_t range) {
int l_val = left + negative;
int r_val = right + negative;
// Вместо log используем статическую, потому что шаблон принимает на вход constexpr - т.е. размер долен быть известен на момент компиляции
std::bitset<staticlog> l_bitset(l_val);
std::bitset<staticlog> r_bitset(r_val);
size_t shift = rand() % (staticlog - 1) + 1;
bitset<staticlog> new_l_bitset = (l_bitset & r_bitset) << shift;
bitset<staticlog> new_r_bitset = (l_bitset | r_bitset) << shift;
genotype_with_parents[from] = (int)new_l_bitset.to_ulong() - negative;
genotype_with_parents[from + 1] = (int)new_r_bitset.to_ulong() - negative;
}
// n - число особей всего, m2 - необходимое число особей
size_t* get_random_indexes(size_t n, size_t m2) {
size_t* indexes = new size_t[n];
size_t* values = new size_t[n];
for (size_t i = 0; i != n; ++i) {
values[i] = i;
}
std::random_device rd;
std::mt19937 gen(rd());
std::shuffle(values, values + n, gen);
for (size_t i = 0; i != m2; ++i) {
indexes[i] = values[i];
}
return indexes;
}
int* populate_genotype(size_t size, int from, int to) {
int* genotype = new int[(size)];
for (size_t i = 0; i != size; ++i) {
std::random_device rd;
std::mt19937 gen(rd());
std::uniform_int_distribution<> dis(from, to);
genotype[i] = dis(gen);
}
return genotype;
}
bool compare(const Indval& left, const Indval& right) {
bool comp = false;
switch (mode) {
case 1:
comp = left.val_ > right.val_;
break;
case 2:
comp = left.val_ < right.val_;
break;
}
return comp;
}
