
Лекция_6_БИ_М_2014
.pdf
BLAST – Basic Local Alignment
and Search Tool
üЛокальное выравнивание

Элементы теории
локального выравнивания
•Задача: по заданной последовательности найти другие в базе данных последовательностей, которые “показывают схожесть” на статистически значимом уровне.
•Цели:
–мы предполагаем, что похожие
последовательности кодируют белки со схожими функциями => предсказывание
функций
–мы предполагаем, что похожие
последовательности произошли от одного
общего предка => проследить эволюционную
историю
Алгоритм BLAST
•Программы BLAST (Basic Local Alignment Search Tools) представляют собой набор алгоритмов для сравнения последовательностей. Были впервые опубликованы в 1990 году для поиска оптимального локального выравнивания данной последовательности с другими в базе данных последовательностей.
–Altschul SF, Gish W, Miller W, Myers EW, Lipman DJ (1990) “Basic local alignment search tool.” J. Mol. Biol. 215:403-410.
–Altschul SF, Madden TL, Schaeffer AA, Zhang J, Zhang Z, Miller W, Lipman DJ (1997) “Gapped BLAST and PSI-BLAST: a new generation of protein database search programs.” NAR 25:33893402.
23
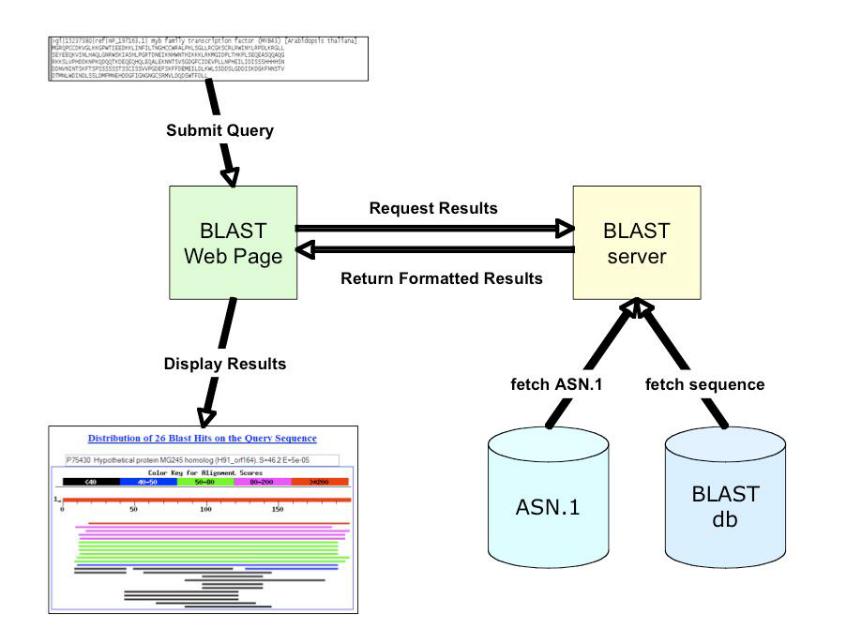
24

Программы BLAST
Программа Описание
blastp |
базы данных белков |
|
|
Сравнивает исходную аминокислотную последовательность с последовательностями из |
|
|
|
|
blastn |
Сравнивает исходную нуклеотидную последовательность с последовательностями из |
|
базы данных нуклеотидных последовательностей |
||
|
|
|
blastx |
Сравнивает исходную нуклеотидную последовательность, оттранслированную в |
|
аминокислотную по всем шести рамкам считывания, с последовательностями из базы |
||
данных белков. Используется для нахождения потенциальных продуктов трансляции |
||
|
неизвестной нуклеотидной последовательности. |
|
|
|
|
tblastn |
Сравнивает исходную аминокислотную последовательность с базой данных |
|
нуклеотидных последовательностей, динамически транслируемых по всем шести |
||
рамкам считывания |
||
|
||
|
|
|
tblastx |
Сравнивает все шесть трансляций исходной нуклеотидной последовательности со |
|
всеми шестью трансляциями из базы данных нуклеотидных последовательностей. |
25

Дополнительные программы
BLAST
Программы |
Характеристики |
||
|
|
|
|
Megablast |
Непрерывный |
Для близких последовательностей |
|
|
|
||
Разрывный |
Для межвидового сравнения |
||
|
|||
|
|
|
|
|
PSI-BLAST |
Автоматически генерирует матрицу счета |
|
|
специфичную к позициями (Position Specific |
||
Специфичен к |
|
Score Matrix, PSSM) |
|
|
|
||
позициям |
|
|
|
RPS-BLAST |
Совершает поиск в базе данных матриц |
||
|
|||
|
PSSMs, сгенерируемых программой PSI- |
||
|
|
BLAST. |
|
|
|
|
|
26

Алгоритм BLAST
•Вес совпадений считается по матрицам счета
Параметр, • Последовательности разбиты на можно менять слова (words) (по умолчанию длина
n=3)
–Обеспечивает скорость и вычислительную эффективность
•Алгоритм BLAST расширяет исзначальный “зародыш” (“seed”) до сегмента с большим весом (High Scoring Pairs, HSP)
27

Алгоритм BLAST
BLAST – алгоритм эффективного локального выравнивания
Три стадии работы алгоритма:
1)LIST
2)SCAN
3)EXTEND

Три стадии работы алгоритма BLAST
1) LIST
Создается список слов (w=3) со скором выше порога T
Пример: …FSGTWYA… |
|
|
A list of words (w=3) is: |
|
|
… FSG SGT GTW TWY WYA … GTW |
6,5,11 |
22 |
GSW |
6,1,11 |
18 |
ATW |
0,5,11 |
16 |
NTW |
0,5,11 |
16 |
T=11 GTY |
6,5,2 |
13 |
GNW |
|
10 |
GAW |
|
9 |
|
|
|
|
|
|
|
|
|
|
|
|
|
|
|
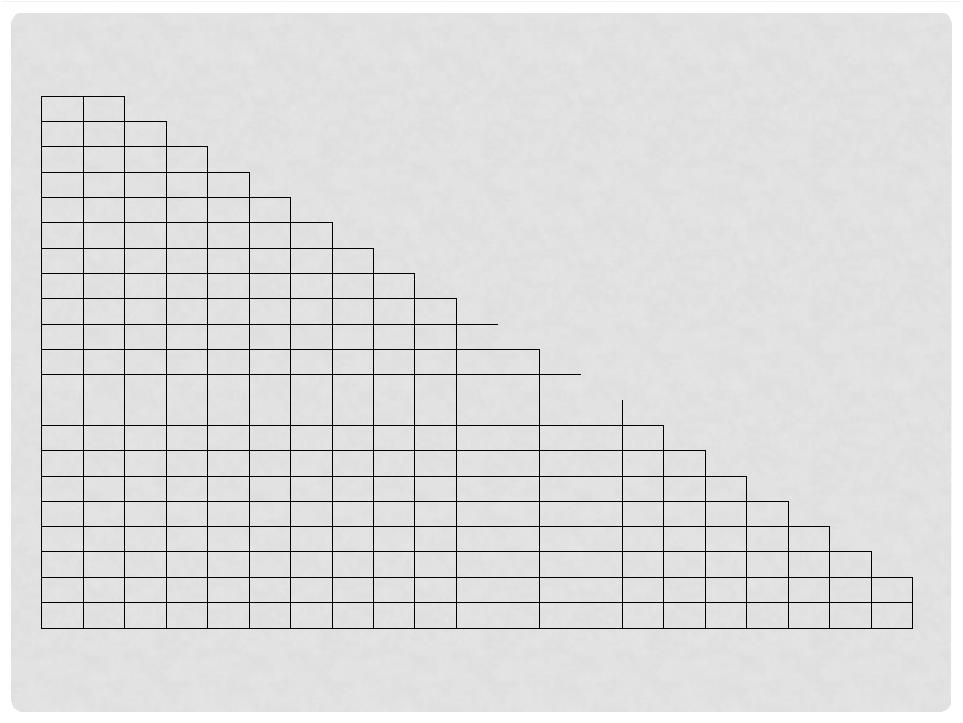
Для оценки |
|
A |
4 |
0 |
6 |
|
|
|
|
|
|
|
|
|
|
выравнивания |
||
N |
-2 |
|
|
|
|
|
|
|
используется Blosum62 |
|||||||
R |
-1 |
5 |
|
|
|
|
|
|
|
|
|
|
|
|
|
|
D |
-2 |
-2 |
1 |
6 |
|
|
|
|
|
|
|
|
|
|
|
|
C |
0 |
-3 |
-3 |
-3 |
9 |
|
|
|
|
|
|
|
|
|
|
|
Q |
-1 |
1 |
0 |
0 |
-3 |
5 |
|
|
|
|
|
|
|
|
|
|
E |
-1 |
0 |
0 |
2 |
-4 |
2 |
5 |
|
|
|
|
|
|
|
|
|
G |
0 |
-2 |
0 |
-1 -3 -2 -2 |
6 |
|
|
|
|
|
|
|
|
|||
H |
-2 |
0 |
1 |
-1 -3 |
0 |
0 |
-2 |
8 |
|
|
|
|
|
|
|
|
I |
-1 -3 -3 -3 -1 -3 -3 -4 -3 |
4 |
|
|
|
|
|
|
||||||||
L |
-1 -2 -3 -4 -1 -2 -3 -4 -3 |
2 |
4 |
|
|
|
|
|
||||||||
K |
-1 |
2 |
0 |
-1 -1 |
1 |
1 |
-2 -1 -3 -2 |
5 |
|
|
|
|
||||
M |
-1 -2 -2 -3 -1 |
0 |
-2 -3 -2 |
1 |
2 |
-1 |
5 |
|
|
|
||||||
F |
-2 -3 -3 -3 -2 -3 -3 -3 -1 |
0 |
0 |
-3 |
0 |
6 |
|
|
||||||||
P |
-1 -2 -2 -1 -3 -1 -1 -2 -2 -3 -3 -1 -2 -4 7 |
|
|
|||||||||||||
S |
1 |
-1 |
1 |
0 |
-1 |
0 |
0 |
0 -1 -2 -2 |
0 |
-1 -2 -1 |
4 |
|
||||
T |
0 |
-1 |
0 |
-1 -1 -1 -1 -2 -2 -1 -1 -1 -1 -2 -1 |
1 |
5 |
||||||||||
W |
-3 -3 -4 -4 -2 -2 -3 -2 -2 -3 -2 -3 -1 |
1 -4 -3 |
-2 11 |
|||||||||||||
Y |
-2 -2 -2 -3 -2 -1 -2 -3 |
2 -1 -1 -2 -1 |
3 -3 -2 |
-2 2 7 |
||||||||||||
V |
0 |
-3 -3 -3 -1 -2 -2 -3 -3 |
3 |
1 |
-2 |
1 |
-1 -2 -2 |
0 -3 -1 4 |
||||||||
|
A R N D C Q E G H I L K M F P S T W Y V |
|||||||||||||||
