
4 / laba4
.docxУФИМСКИЙ ГОСУДАРСТВЕННЫЙ АВИАЦИОННЫЙ ТЕХНИЧЕСКИЙ УНИВЕРСИТЕТ
ФАКУЛЬТЕТ ИНФОРМАТИКИ И РОБОТОТЕХНИКИ
КАФЕДРА ВЫЧИСЛИТЕЛЬНОЙ МАТЕМАТИКИ И КИБЕРНЕТИКИ
|
|
|
||
|
УТВЕРЖДАЮ Проректор университета по научной работе ФИО |
|||
|
|
|
||
|
"___" ______________ _______г. |
|||
|
|
|
||
ОТЧЕТ О ПРОВЕДЕНИИ ЛАБОРАТОРНОЙ РАБОТЫ № 4 «Модели множественного выбора ROC-анализ» |
||||
|
||||
по предмету: Статистическое моделирование |
||||
Преподаватель |
|
Е.Ю. Сазонова |
||
|
|
|
||
|
|
|
||
|
|
|
||
Исполнители |
|
К.Б. Ибрагимова А.Р. Шакиров |
||
Уфа 2021
Laba4
Ибрагимова, Шакиров
24 05 2021
library(ggplot2) library(memisc)
## Loading required package: lattice
## Loading required package: MASS
## ## Attaching package: 'memisc'
## The following object is masked from 'package:ggplot2': ## ## syms
## The following objects are masked from 'package:stats': ## ## contr.sum, contr.treatment, contrasts
## The following object is masked from 'package:base': ## ## as.array
library(DescTools)
## ## Attaching package: 'DescTools'
## The following objects are masked from 'package:memisc': ## ## %nin%, Cor, Cov, Mean, Median, Range, Var
library(lmtest)
## Loading required package: zoo
## ## Attaching package: 'zoo'
## The following objects are masked from 'package:base': ## ## as.Date, as.Date.numeric
library(caTools) library(dplyr)
## ## Attaching package: 'dplyr'
## The following objects are masked from 'package:memisc': ## ## collect, recode, rename, syms
## The following object is masked from 'package:MASS': ## ## select
## The following objects are masked from 'package:stats': ## ## filter, lag
## The following objects are masked from 'package:base': ## ## intersect, setdiff, setequal, union
library(readxl) library(knitr) library(kernlab)
## ## Attaching package: 'kernlab'
## The following object is masked from 'package:ggplot2': ## ## alpha
library(caret)
## ## Attaching package: 'caret'
## The following objects are masked from 'package:DescTools': ## ## MAE, RMSE
library(mfx)
## Loading required package: sandwich
## Loading required package: betareg
library(pROC)
## Type 'citation("pROC")' for a citation.
## ## Attaching package: 'pROC'
## The following objects are masked from 'package:stats': ## ## cov, smooth, var
library(ResourceSelection)
## ResourceSelection 0.3-5 2019-07-22
library(ROCR) library(nortest) #library(MKmisc) #R version - 64-bit 3.6.3
Исходные данные были собраны и предоставлены “Национальным институтом диабета, болезней органов пищеварения и почек” в рамках Базы данных по диабету индейцев Пима.
Источник: https://www.kaggle.com/kandij/diabetes-dataset
Выбранные поля для анализа: 1. Беременность 2. Глюкоза 3. Кровяное давление 4. Индекс массы тела 5. Наследственость 6. Возраст 7. Исход
Цель исследования – выяснить,какие из перечисленных выше факторов влияют на исход заболевания диабетом у пациентов. Если есть связь – установить ее характер и силу.
Загрузим набор данных
setwd("C:/Users/Karina/Desktop/BigData") d<- read.csv("diabetes.csv", sep=";", dec = ",", header=TRUE, fileEncoding="UTF-8-BOM")
Преобразуем зависимую качественную переменную.
d$Outcome <- as.factor(d$Outcome)
Разделим выборку на тестовую и обучающую
set.seed(1) split <- sample.split(d$Outcome, SplitRatio = 0.7) train <- subset(d, split == TRUE) test <- subset(d, split == FALSE)
Построим регрессионные модели (пробит (Probit), логит (Logit) или гомпит (Extreme Value)) с бинарной зависимой переменной. В качестве зависимой переменной выступает – Outcome, все остальные являются независимыми.
Логит-модель
model_1 <- glm(Outcome ~ ., train, family = binomial(link = "logit")) summary(model_1)
## ## Call: ## glm(formula = Outcome ~ ., family = binomial(link = "logit"), ## data = train) ## ## Deviance Residuals: ## Min 1Q Median 3Q Max ## -2.5749 -0.7130 -0.3885 0.6521 3.0943 ## ## Coefficients: ## Estimate Std. Error z value Pr(>|z|) ## (Intercept) -8.779090 0.893384 -9.827 < 2e-16 *** ## Pregnancies 0.099954 0.038935 2.567 0.01025 * ## Glucose 0.037456 0.004274 8.764 < 2e-16 *** ## BloodPressure -0.013451 0.006024 -2.233 0.02554 * ## BMI 0.090985 0.017452 5.213 1.85e-07 *** ## DiabetesPedigreeFunction 1.082489 0.366335 2.955 0.00313 ** ## Age 0.012748 0.011439 1.114 0.26509 ## --- ## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1 ## ## (Dispersion parameter for binomial family taken to be 1) ## ## Null deviance: 696.28 on 537 degrees of freedom ## Residual deviance: 489.64 on 531 degrees of freedom ## AIC: 503.64 ## ## Number of Fisher Scoring iterations: 5
Пробит – модель
model_2 <- glm(Outcome ~ ., train, family =binomial(link = "probit")) summary(model_2)
## ## Call: ## glm(formula = Outcome ~ ., family = binomial(link = "probit"), ## data = train) ## ## Deviance Residuals: ## Min 1Q Median 3Q Max ## -2.5970 -0.7264 -0.3781 0.6932 3.3732 ## ## Coefficients: ## Estimate Std. Error z value Pr(>|z|) ## (Intercept) -5.048193 0.480644 -10.503 < 2e-16 *** ## Pregnancies 0.060290 0.022373 2.695 0.00704 ** ## Glucose 0.021250 0.002358 9.013 < 2e-16 *** ## BloodPressure -0.008238 0.003489 -2.361 0.01821 * ## BMI 0.053666 0.009830 5.460 4.77e-08 *** ## DiabetesPedigreeFunction 0.575509 0.207173 2.778 0.00547 ** ## Age 0.008058 0.006647 1.212 0.22545 ## --- ## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1 ## ## (Dispersion parameter for binomial family taken to be 1) ## ## Null deviance: 696.28 on 537 degrees of freedom ## Residual deviance: 491.06 on 531 degrees of freedom ## AIC: 505.06 ## ## Number of Fisher Scoring iterations: 5
Гомпит – модель
model_3 <- glm(Outcome ~ ., train, family =binomial(link = "cloglog")) summary(model_3)
## ## Call: ## glm(formula = Outcome ~ ., family = binomial(link = "cloglog"), ## data = train) ## ## Deviance Residuals: ## Min 1Q Median 3Q Max ## -2.8332 -0.7109 -0.4679 0.7004 2.7759 ## ## Coefficients: ## Estimate Std. Error z value Pr(>|z|) ## (Intercept) -6.244960 0.593335 -10.525 < 2e-16 *** ## Pregnancies 0.069187 0.025825 2.679 0.00738 ** ## Glucose 0.026219 0.002767 9.475 < 2e-16 *** ## BloodPressure -0.010942 0.003993 -2.740 0.00614 ** ## BMI 0.062446 0.011794 5.295 1.19e-07 *** ## DiabetesPedigreeFunction 0.432414 0.237267 1.822 0.06838 . ## Age 0.006009 0.008059 0.746 0.45591 ## --- ## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1 ## ## (Dispersion parameter for binomial family taken to be 1) ## ## Null deviance: 696.28 on 537 degrees of freedom ## Residual deviance: 498.31 on 531 degrees of freedom ## AIC: 512.31 ## ## Number of Fisher Scoring iterations: 10
В моделях должны остаться только статистически значимые переменные, оценим параметры методом максимального правдоподобия, применяя метод пошагового исключения. В моделях все переменные должны быть статистически значимы.
Исключим незначимый параметр Age.
d1 <- data.frame(select(d, -Age))
Преобразуем зависимую качественную переменную.
d1$Outcome <- as.factor(d1$Outcome)
Разделим выборку на тестовую и обучающую
set.seed(2) split1 <- sample.split(d1$Outcome, SplitRatio = 0.7) train1 <- subset(d1, split == TRUE) test1 <- subset(d1, split == FALSE)
Логит-модель
model_1 <- glm(Outcome ~ ., train1, family = binomial(link = "logit")) summary(model_1)
## ## Call: ## glm(formula = Outcome ~ ., family = binomial(link = "logit"), ## data = train1) ## ## Deviance Residuals: ## Min 1Q Median 3Q Max ## -2.5644 -0.7036 -0.3910 0.6664 3.1355 ## ## Coefficients: ## Estimate Std. Error z value Pr(>|z|) ## (Intercept) -8.524517 0.855772 -9.961 < 2e-16 *** ## Pregnancies 0.122825 0.033290 3.690 0.000225 *** ## Glucose 0.038319 0.004217 9.088 < 2e-16 *** ## BloodPressure -0.012711 0.005980 -2.126 0.033541 * ## BMI 0.088796 0.017290 5.136 2.81e-07 *** ## DiabetesPedigreeFunction 1.093122 0.365595 2.990 0.002790 ** ## --- ## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1 ## ## (Dispersion parameter for binomial family taken to be 1) ## ## Null deviance: 696.28 on 537 degrees of freedom ## Residual deviance: 490.87 on 532 degrees of freedom ## AIC: 502.87 ## ## Number of Fisher Scoring iterations: 5
Пробит – модель
model_2 <- glm(Outcome ~ ., train1, family = binomial(link = "probit")) summary(model_2)
## ## Call: ## glm(formula = Outcome ~ ., family = binomial(link = "probit"), ## data = train1) ## ## Deviance Residuals: ## Min 1Q Median 3Q Max ## -2.5848 -0.7243 -0.3790 0.6975 3.4415 ## ## Coefficients: ## Estimate Std. Error z value Pr(>|z|) ## (Intercept) -4.898971 0.460302 -10.643 < 2e-16 *** ## Pregnancies 0.073572 0.019067 3.859 0.000114 *** ## Glucose 0.021772 0.002318 9.391 < 2e-16 *** ## BloodPressure -0.007601 0.003455 -2.200 0.027813 * ## BMI 0.052508 0.009748 5.387 7.18e-08 *** ## DiabetesPedigreeFunction 0.583169 0.206858 2.819 0.004815 ** ## --- ## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1 ## ## (Dispersion parameter for binomial family taken to be 1) ## ## Null deviance: 696.28 on 537 degrees of freedom ## Residual deviance: 492.55 on 532 degrees of freedom ## AIC: 504.55 ## ## Number of Fisher Scoring iterations: 5
Гомпит – модель
model_3 <- glm(Outcome ~ ., train1, family = binomial(link = "cloglog")) summary(model_3)
## ## Call: ## glm(formula = Outcome ~ ., family = binomial(link = "cloglog"), ## data = train1) ## ## Deviance Residuals: ## Min 1Q Median 3Q Max ## -2.8294 -0.7108 -0.4662 0.6960 2.8078 ## ## Coefficients: ## Estimate Std. Error z value Pr(>|z|) ## (Intercept) -6.122352 0.562341 -10.887 < 2e-16 *** ## Pregnancies 0.077535 0.022482 3.449 0.000563 *** ## Glucose 0.026791 0.002721 9.846 < 2e-16 *** ## BloodPressure -0.010661 0.003969 -2.686 0.007222 ** ## BMI 0.060981 0.011644 5.237 1.63e-07 *** ## DiabetesPedigreeFunction 0.448611 0.238826 1.878 0.060326 . ## --- ## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1 ## ## (Dispersion parameter for binomial family taken to be 1) ## ## Null deviance: 696.28 on 537 degrees of freedom ## Residual deviance: 498.95 on 532 degrees of freedom ## AIC: 510.95 ## ## Number of Fisher Scoring iterations: 9
Выведем информацию по трем моделям.
mtable(model_1, model_2, model_3,summary.stats =c('AIC', 'BIC', 'McFadden R-sq.', 'Likelihood-ratio', 'Log-likelihood', 'p'))
## ## Calls: ## model_1: glm(formula = Outcome ~ ., family = binomial(link = "logit"), ## data = train1) ## model_2: glm(formula = Outcome ~ ., family = binomial(link = "probit"), ## data = train1) ## model_3: glm(formula = Outcome ~ ., family = binomial(link = "cloglog"), ## data = train1) ## ## =================================================================== ## model_1 model_2 model_3 ## ------------------------------------------------------------------- ## (Intercept) -8.525*** -4.899*** -6.122*** ## (0.856) (0.460) (0.562) ## Pregnancies 0.123*** 0.074*** 0.078*** ## (0.033) (0.019) (0.022) ## Glucose 0.038*** 0.022*** 0.027*** ## (0.004) (0.002) (0.003) ## BloodPressure -0.013* -0.008* -0.011** ## (0.006) (0.003) (0.004) ## BMI 0.089*** 0.053*** 0.061*** ## (0.017) (0.010) (0.012) ## DiabetesPedigreeFunction 1.093** 0.583** 0.449 ## (0.366) (0.207) (0.239) ## ------------------------------------------------------------------- ## AIC 502.870 504.554 510.948 ## BIC 528.597 530.281 536.676 ## McFadden R-sq. 0.295 0.293 0.283 ## Likelihood-ratio 205.410 203.726 197.332 ## Log-likelihood -245.435 -246.277 -249.474 ## p 0.000 0.000 0.000 ## =================================================================== ## Significance: *** = p < 0.001; ** = p < 0.01; * = p < 0.05
Наименьшие информационные критерии у логит-модели AIC - 502.870 и BIC - 528.597.
Коэффициент детерминации R2 Макфаддена - 0.295 Коэффициент показывает, насколько изменения зависимой переменной (в процентах) объясняются изменениями совокупности независимых переменных Модель считается адекватной.
Критическая статистика для теста отношения правдоподобия p(Likelihoodratio) - 205.410 Если p(Likelihood-ratio) мала и меньше уровня значимости α, модель является значимой.
Критерий правдоподобия (Log-likelihood) - -245.435 Логарифмическое правдоподобие показывает, насколько хорошо модель соответствует исходным данным. Снижение его величины означает улучшение качества модели (чем меньше значение, тем выше качество модели).
У модели высокое качество.
Критерий согласия Хосмера–Лемешоу y = числовой вектор наблюдений, двоичный (0/1) yhat = ожидаемые значения (вероятности )
y <- model_1$y yhat <- predict(model_1,train1, type="response") hoslem.test(y, yhat)
## ## Hosmer and Lemeshow goodness of fit (GOF) test ## ## data: y, yhat ## X-squared = 4.381, df = 8, p-value = 0.8212
Уровень значимости является большим, модель хорошо откалибрована и достаточно точно описывает реальные данные.
Графический тест:
qqnorm(model_1$residuals)

Рассмотрим параметрические тесты:
lillie.test(model_1$residuals)
## ## Lilliefors (Kolmogorov-Smirnov) normality test ## ## data: model_1$residuals ## D = 0.30414, p-value < 2.2e-16
Поскольку значение p-value меньше 0.05 нулевая гипотеза о согласие распределения остатков с нормальным законом распределения отвергается.
Спрогнозируем значения по выбранной модели на тестовой выборке.
test1$pred_1 <- predict(model_1, test1, type = 'response')
Расчетаем маржинальный эффект для логит-модели
logitmfx(Outcome ~ Pregnancies + Glucose + BloodPressure + BMI + DiabetesPedigreeFunction, data = test1)
## Call: ## logitmfx(formula = Outcome ~ Pregnancies + Glucose + BloodPressure + ## BMI + DiabetesPedigreeFunction, data = test1) ## ## Marginal Effects: ## dF/dx Std. Err. z P>|z| ## Pregnancies 0.0476313 0.0109780 4.3388 1.433e-05 *** ## Glucose 0.0059406 0.0012286 4.8352 1.330e-06 *** ## BloodPressure -0.0020503 0.0020566 -0.9970 0.318783 ## BMI 0.0160701 0.0052483 3.0620 0.002199 ** ## DiabetesPedigreeFunction 0.1255835 0.1078624 1.1643 0.244305 ## --- ## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
Маржинальные эффекты умножаются на 100% и интерпретируются как предсказательный эффект влияния независимого фактора на вероятность положительной альтернативы.
Оценим качество классификации
test1$pred1 <- ifelse(test1$pred_1 < 0.8, 0, 1) test1$pred1 <- as.factor(test1$pred1) confusionMatrix(data = test1$pred1,reference = test1$Outcome, positive = "1")
## Confusion Matrix and Statistics ## ## Reference ## Prediction 0 1 ## 0 147 66 ## 1 3 14 ## ## Accuracy : 0.7 ## 95% CI : (0.6363, 0.7585) ## No Information Rate : 0.6522 ## P-Value [Acc > NIR] : 0.07181 ## ## Kappa : 0.1899 ## ## Mcnemar's Test P-Value : 8.398e-14 ## ## Sensitivity : 0.17500 ## Specificity : 0.98000 ## Pos Pred Value : 0.82353 ## Neg Pred Value : 0.69014 ## Prevalence : 0.34783 ## Detection Rate : 0.06087 ## Detection Prevalence : 0.07391 ## Balanced Accuracy : 0.57750 ## ## 'Positive' Class : 1 ##
Для оценки качества построенной модели бинарного выбора используется ROC кривая. ROC-кривая показывает зависимость количества верно классифицированных положительных примеров от количества неверно классифицированных отрицательных примеров.
Чем ближе кривая к верхнему левому углу, тем выше предсказательная способность модели. Наоборот, чем меньше изгиб кривой и чем ближе она расположена к диагональной прямой, тем менее эффективна модель.
Построим ROC-кривую для Логит-модели
pr <- prediction(test1$pred_1, test1$Outcome) prf <- performance(pr, measure = "tpr", x.measure = "fpr") plot(prf) lines(c(0,1),c(0,1)) title("ROC-кривая")

Численный показатель площади под кривой AUC (Area Under Curve)
auc.tmp <- performance(pr,"auc"); auc <- as.numeric(auc.tmp@y.values) show(auc)
## [1] 0.8054167
Качество модели очень хорошее.
Коэффициент Джини статистический показатель степени неоднородности (расслоения) по отношению к какому-либо изучаемому признаку.
Коэффициент Джини изменяется от 0 до 1. Чем больше его значение отклоняется от нуля и приближается к единице, тем в большей степени признак сконцентрирован в отдельных группах.
Gini <- 2 * auc * (auc - 0.5) show(Gini)
## [1] 0.4919753
Построим ROC-кривую для Пробит-модели
test1$pred_2 <- predict(model_2, test1, type = 'response') pr <- prediction(test1$pred_2, test1$Outcome) prf <- performance(pr, measure = "tpr", x.measure = "fpr") plot(prf) lines(c(0,1),c(0,1)) title("ROC-кривая")
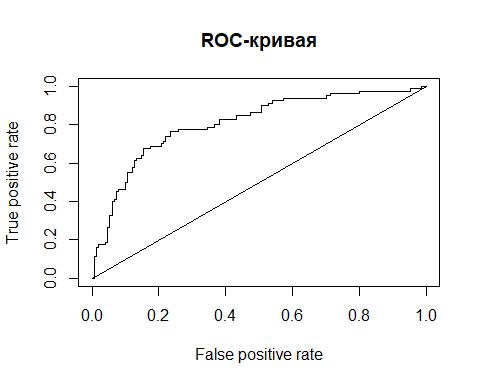
Численный показатель площади под кривой AUC (Area Under Curve)
auc.tmp <- performance(pr,"auc"); auc <- as.numeric(auc.tmp@y.values) show(auc)
## [1] 0.80725
Качество модели очень хорошее.
Коэффициент Джини статистический показатель степени неоднородности (расслоения) по отношению к какому-либо изучаемому признаку.
Коэффициент Джини изменяется от 0 до 1. Чем больше его значение отклоняется от нуля и приближается к единице, тем в большей степени признак сконцентрирован в отдельных группах.
Gini <- 2 * auc * (auc - 0.5) show(Gini)
## [1] 0.4960551
Построим ROC-кривую для Гомплит-модели
test1$pred_3 <- predict(model_3, test1, type = 'response') pr <- prediction(test1$pred_3, test1$Outcome) prf <- performance(pr, measure = "tpr", x.measure = "fpr") plot(prf) lines(c(0,1),c(0,1)) title("ROC-кривая")

Численный показатель площади под кривой AUC (Area Under Curve)
auc.tmp <- performance(pr,"auc"); auc <- as.numeric(auc.tmp@y.values) show(auc)
## [1] 0.80325
Качество модели очень хорошее.
Коэффициент Джини статистический показатель степени неоднородности (расслоения) по отношению к какому-либо изучаемому признаку.
Коэффициент Джини изменяется от 0 до 1. Чем больше его значение отклоняется от нуля и приближается к единице, тем в большей степени признак сконцентрирован в отдельных группах.
Gini <- 2 * auc * (auc - 0.5) show(Gini)
## [1] 0.4871711
Выбранная Логит-модель лучше откалибрована и достаточно точно описывает реальные данные.
Сэмплирование метод выбора подмножества наблюдаемых величин из множества, с целью выделения неких свойств исходного множества.
Неслучайный Undersampling
set.seed(1) train_up <- upSample(x = train1[, 1:5], y = train1$Outcome, yname = "Outcome")
Логит-модель
model1 <- glm(Outcome ~ ., train_up, family = binomial(link = "logit")) summary(model1)
## ## Call: ## glm(formula = Outcome ~ ., family = binomial(link = "logit"), ## data = train_up) ## ## Deviance Residuals: ## Min 1Q Median 3Q Max ## -2.80926 -0.78772 0.00292 0.73350 2.97982 ## ## Coefficients: ## Estimate Std. Error z value Pr(>|z|) ## (Intercept) -7.983759 0.712262 -11.209 < 2e-16 *** ## Pregnancies 0.109125 0.028715 3.800 0.000145 *** ## Glucose 0.039598 0.003740 10.588 < 2e-16 *** ## BloodPressure -0.011415 0.004782 -2.387 0.016986 * ## BMI 0.086796 0.014452 6.006 1.9e-09 *** ## DiabetesPedigreeFunction 1.054531 0.306448 3.441 0.000579 *** ## --- ## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1 ## ## (Dispersion parameter for binomial family taken to be 1) ## ## Null deviance: 970.41 on 699 degrees of freedom ## Residual deviance: 689.55 on 694 degrees of freedom ## AIC: 701.55 ## ## Number of Fisher Scoring iterations: 5
Пробит-модель
model2 <- glm(Outcome ~ ., train_up, family = binomial(link = "probit")) summary(model2)
## ## Call: ## glm(formula = Outcome ~ ., family = binomial(link = "probit"), ## data = train_up) ## ## Deviance Residuals: ## Min 1Q Median 3Q Max ## -2.9256 -0.8061 0.0040 0.7736 3.2202 ## ## Coefficients: ## Estimate Std. Error z value Pr(>|z|) ## (Intercept) -4.614641 0.386328 -11.945 < 2e-16 *** ## Pregnancies 0.064556 0.016581 3.893 9.88e-05 *** ## Glucose 0.022753 0.002048 11.112 < 2e-16 *** ## BloodPressure -0.006929 0.002777 -2.495 0.01260 * ## BMI 0.051667 0.008138 6.349 2.17e-10 *** ## DiabetesPedigreeFunction 0.553270 0.177343 3.120 0.00181 ** ## --- ## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1 ## ## (Dispersion parameter for binomial family taken to be 1) ## ## Null deviance: 970.41 on 699 degrees of freedom ## Residual deviance: 691.89 on 694 degrees of freedom ## AIC: 703.89 ## ## Number of Fisher Scoring iterations: 5
Гомпит-модель
model3 <- glm(Outcome ~ ., train_up, family = binomial(link = "cloglog")) summary(model3)
## ## Call: ## glm(formula = Outcome ~ ., family = binomial(link = "cloglog"), ## data = train_up) ## ## Deviance Residuals: ## Min 1Q Median 3Q Max ## -3.1897 -0.8266 -0.0910 0.8413 2.5184 ## ## Coefficients: ## Estimate Std. Error z value Pr(>|z|) ## (Intercept) -5.135718 0.427524 -12.013 < 2e-16 *** ## Pregnancies 0.063046 0.017693 3.563 0.000366 *** ## Glucose 0.024067 0.002146 11.215 < 2e-16 *** ## BloodPressure -0.008599 0.002891 -2.975 0.002933 ** ## BMI 0.054194 0.008610 6.295 3.08e-10 *** ## DiabetesPedigreeFunction 0.394970 0.190950 2.068 0.038598 * ## --- ## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1 ## ## (Dispersion parameter for binomial family taken to be 1) ## ## Null deviance: 970.41 on 699 degrees of freedom ## Residual deviance: 704.09 on 694 degrees of freedom ## AIC: 716.09 ## ## Number of Fisher Scoring iterations: 8
Выведем информацию по трем моделям.
mtable(model1, model2, model3,summary.stats =c('AIC', 'BIC', 'McFadden R-sq.', 'Likelihood-ratio', 'Log-likelihood', 'p'))
## ## Calls: ## model1: glm(formula = Outcome ~ ., family = binomial(link = "logit"), ## data = train_up) ## model2: glm(formula = Outcome ~ ., family = binomial(link = "probit"), ## data = train_up) ## model3: glm(formula = Outcome ~ ., family = binomial(link = "cloglog"), ## data = train_up) ## ## =================================================================== ## model1 model2 model3 ## ------------------------------------------------------------------- ## (Intercept) -7.984*** -4.615*** -5.136*** ## (0.712) (0.386) (0.428) ## Pregnancies 0.109*** 0.065*** 0.063*** ## (0.029) (0.017) (0.018) ## Glucose 0.040*** 0.023*** 0.024*** ## (0.004) (0.002) (0.002) ## BloodPressure -0.011* -0.007* -0.009** ## (0.005) (0.003) (0.003) ## BMI 0.087*** 0.052*** 0.054*** ## (0.014) (0.008) (0.009) ## DiabetesPedigreeFunction 1.055*** 0.553** 0.395* ## (0.306) (0.177) (0.191) ## ------------------------------------------------------------------- ## AIC 701.550 703.893 716.087 ## BIC 728.856 731.199 743.394 ## McFadden R-sq. 0.289 0.287 0.274 ## Likelihood-ratio 280.856 278.513 266.319 ## Log-likelihood -344.775 -345.946 -352.044 ## p 0.000 0.000 0.000 ## =================================================================== ## Significance: *** = p < 0.001; ** = p < 0.01; * = p < 0.05
Наименьшие информационные критерии у логит-модели AIC - 701.550 и BIC - 728.856.
Коэффициент детерминации R2 Макфаддена - 0.289 Коэффициент показывает, насколько изменения зависимой переменной (в процентах) объясняются изменениями совокупности независимых переменных Модель считается адекватной.
Критическая статистика для теста отношения правдоподобия p(Likelihoodratio) - 280.856 Если p(Likelihood-ratio) мала и меньше уровня значимости α, модель является значимой.
Критерий правдоподобия (Log-likelihood) - -344.775 Логарифмическое правдоподобие показывает, насколько хорошо модель соответствует исходным данным. Снижение его величины означает улучшение качества модели (чем меньше значение, тем выше качество модели).
У модели ухудшилось качество.
Критерий согласия Хосмера–Лемешоу y = числовой вектор наблюдений, двоичный (0/1) yhat = ожидаемые значения (вероятности )
y <- model1$y yhat <- predict(model1,train_up, type="response") hoslem.test(y, yhat)
## ## Hosmer and Lemeshow goodness of fit (GOF) test ## ## data: y, yhat ## X-squared = 10.97, df = 8, p-value = 0.2034
Уровень значимости является небольшим, модель не точно описывает реальные данные.
Графический тест:
qqnorm(model1$residuals)

Рассмотрим параметрические тесты:
lillie.test(model1$residuals)
## ## Lilliefors (Kolmogorov-Smirnov) normality test ## ## data: model1$residuals ## D = 0.23456, p-value < 2.2e-16
Поскольку значение p-value меньше 0.05 нулевая гипотеза о согласие распределения остатков с нормальным законом распределения отвергается.
Спрогнозируем значения по выбранной модели на тестовой выборке.
test1$pred_1 <- predict(model1, test1, type = 'response')
Расчетаем маржинальный эффект для логит-модели
logitmfx(Outcome ~ Pregnancies + Glucose + BloodPressure + BMI + DiabetesPedigreeFunction, data = test1)
## Call: ## logitmfx(formula = Outcome ~ Pregnancies + Glucose + BloodPressure + ## BMI + DiabetesPedigreeFunction, data = test1) ## ## Marginal Effects: ## dF/dx Std. Err. z P>|z| ## Pregnancies 0.0476313 0.0109780 4.3388 1.433e-05 *** ## Glucose 0.0059406 0.0012286 4.8352 1.330e-06 *** ## BloodPressure -0.0020503 0.0020566 -0.9970 0.318783 ## BMI 0.0160701 0.0052483 3.0620 0.002199 ** ## DiabetesPedigreeFunction 0.1255835 0.1078624 1.1643 0.244305 ## --- ## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
Маржинальные эффекты умножаются на 100% и интерпретируются как предсказательный эффект влияния независимого фактора на вероятность положительной альтернативы.
Оценим качество классификации
test1$pred1 <- ifelse(test1$pred_1 < 0.8, 0, 1) test1$pred1 <- as.factor(test1$pred1) confusionMatrix(data = test1$pred1,reference = test1$Outcome, positive = "1")
## Confusion Matrix and Statistics ## ## Reference ## Prediction 0 1 ## 0 141 54 ## 1 9 26 ## ## Accuracy : 0.7261 ## 95% CI : (0.6636, 0.7826) ## No Information Rate : 0.6522 ## P-Value [Acc > NIR] : 0.01019 ## ## Kappa : 0.305 ## ## Mcnemar's Test P-Value : 2.965e-08 ## ## Sensitivity : 0.3250 ## Specificity : 0.9400 ## Pos Pred Value : 0.7429 ## Neg Pred Value : 0.7231 ## Prevalence : 0.3478 ## Detection Rate : 0.1130 ## Detection Prevalence : 0.1522 ## Balanced Accuracy : 0.6325 ## ## 'Positive' Class : 1 ##
Для оценки качества построенной модели бинарного выбора используется ROC кривая. ROC-кривая показывает зависимость количества верно классифицированных положительных примеров от количества неверно классифицированных отрицательных примеров.
Чем ближе кривая к верхнему левому углу, тем выше предсказательная способность модели. Наоборот, чем меньше изгиб кривой и чем ближе она расположена к диагональной прямой, тем менее эффективна модель.
pr <- prediction(test1$pred_1, test1$Outcome) prf <- performance(pr, measure = "tpr", x.measure = "fpr") plot(prf) lines(c(0,1),c(0,1)) title("ROC-кривая")

Численный показатель площади под кривой AUC (Area Under Curve)
auc.tmp <- performance(pr,"auc"); auc <- as.numeric(auc.tmp@y.values) show(auc)
## [1] 0.804
Качество модели очень хорошее.
Коэффициент Джини статистический показатель степени неоднородности (расслоения) по отношению к какому-либо изучаемому признаку.
Коэффициент Джини изменяется от 0 до 1. Чем больше его значение отклоняется от нуля и приближается к единице, тем в большей степени признак сконцентрирован в отдельных группах.
Gini <- 2 * auc * (auc - 0.5) show(Gini)
## [1] 0.488832
Выбранная недостаточно точно описывает реальные данные.
Random Undersampling
set.seed(951) train_down <- downSample(x = train1[, 1:5], y = train1$Outcome, yname = "Outcome")
Логит-модель
model1 <- glm(Outcome ~ ., train_down, family = binomial(link = "logit")) summary(model1)
## ## Call: ## glm(formula = Outcome ~ ., family = binomial(link = "logit"), ## data = train_down) ## ## Deviance Residuals: ## Min 1Q Median 3Q Max ## -2.71100 -0.82442 -0.00089 0.77339 2.92184 ## ## Coefficients: ## Estimate Std. Error z value Pr(>|z|) ## (Intercept) -7.753189 0.982718 -7.890 3.03e-15 *** ## Pregnancies 0.083270 0.036723 2.268 0.0234 * ## Glucose 0.037544 0.004882 7.690 1.47e-14 *** ## BloodPressure -0.012795 0.007211 -1.774 0.0760 . ## BMI 0.091462 0.020179 4.533 5.83e-06 *** ## DiabetesPedigreeFunction 1.028889 0.406608 2.530 0.0114 * ## --- ## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1 ## ## (Dispersion parameter for binomial family taken to be 1) ## ## Null deviance: 521.25 on 375 degrees of freedom ## Residual deviance: 379.13 on 370 degrees of freedom ## AIC: 391.13 ## ## Number of Fisher Scoring iterations: 5
model2 <- glm(Outcome ~ ., train_down, family = binomial(link = "probit")) summary(model2)
## ## Call: ## glm(formula = Outcome ~ ., family = binomial(link = "probit"), ## data = train_down) ## ## Deviance Residuals: ## Min 1Q Median 3Q Max ## -2.77219 -0.84967 0.00099 0.81086 3.10234 ## ## Coefficients: ## Estimate Std. Error z value Pr(>|z|) ## (Intercept) -4.458259 0.533916 -8.350 < 2e-16 *** ## Pregnancies 0.049983 0.021293 2.347 0.0189 * ## Glucose 0.021401 0.002679 7.989 1.36e-15 *** ## BloodPressure -0.007979 0.004146 -1.925 0.0543 . ## BMI 0.055178 0.011473 4.809 1.51e-06 *** ## DiabetesPedigreeFunction 0.524105 0.232973 2.250 0.0245 * ## --- ## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1 ## ## (Dispersion parameter for binomial family taken to be 1) ## ## Null deviance: 521.25 on 375 degrees of freedom ## Residual deviance: 380.91 on 370 degrees of freedom ## AIC: 392.91 ## ## Number of Fisher Scoring iterations: 5
model3 <- glm(Outcome ~ ., train_down, family = binomial(link = "cloglog")) summary(model3)
## ## Call: ## glm(formula = Outcome ~ ., family = binomial(link = "cloglog"), ## data = train_down) ## ## Deviance Residuals: ## Min 1Q Median 3Q Max ## -2.9943 -0.8387 -0.0918 0.8518 2.5217 ## ## Coefficients: ## Estimate Std. Error z value Pr(>|z|) ## (Intercept) -5.195991 0.607546 -8.552 < 2e-16 *** ## Pregnancies 0.044454 0.023128 1.922 0.0546 . ## Glucose 0.024062 0.002875 8.370 < 2e-16 *** ## BloodPressure -0.010192 0.004350 -2.343 0.0191 * ## BMI 0.061400 0.012551 4.892 9.98e-07 *** ## DiabetesPedigreeFunction 0.327995 0.248515 1.320 0.1869 ## --- ## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1 ## ## (Dispersion parameter for binomial family taken to be 1) ## ## Null deviance: 521.25 on 375 degrees of freedom ## Residual deviance: 383.13 on 370 degrees of freedom ## AIC: 395.13 ## ## Number of Fisher Scoring iterations: 8
Выведем информацию по трем моделям.
mtable(model1, model2, model3,summary.stats =c('AIC', 'BIC', 'McFadden R-sq.', 'Likelihood-ratio', 'Log-likelihood', 'p'))
## ## Calls: ## model1: glm(formula = Outcome ~ ., family = binomial(link = "logit"), ## data = train_down) ## model2: glm(formula = Outcome ~ ., family = binomial(link = "probit"), ## data = train_down) ## model3: glm(formula = Outcome ~ ., family = binomial(link = "cloglog"), ## data = train_down) ## ## =================================================================== ## model1 model2 model3 ## ------------------------------------------------------------------- ## (Intercept) -7.753*** -4.458*** -5.196*** ## (0.983) (0.534) (0.608) ## Pregnancies 0.083* 0.050* 0.044 ## (0.037) (0.021) (0.023) ## Glucose 0.038*** 0.021*** 0.024*** ## (0.005) (0.003) (0.003) ## BloodPressure -0.013 -0.008 -0.010* ## (0.007) (0.004) (0.004) ## BMI 0.091*** 0.055*** 0.061*** ## (0.020) (0.011) (0.013) ## DiabetesPedigreeFunction 1.029* 0.524* 0.328 ## (0.407) (0.233) (0.249) ## ------------------------------------------------------------------- ## AIC 391.129 392.909 395.133 ## BIC 414.707 416.487 418.711 ## McFadden R-sq. 0.273 0.269 0.265 ## Likelihood-ratio 142.118 140.338 138.113 ## Log-likelihood -189.564 -190.455 -191.567 ## p 0.000 0.000 0.000 ## =================================================================== ## Significance: *** = p < 0.001; ** = p < 0.01; * = p < 0.05
Наименьшие информационные критерии у логит-модели AIC - 391.129 и BIC - 414.707.
Коэффициент детерминации R2 Макфаддена - 0.273 Коэффициент показывает, насколько изменения зависимой переменной (в процентах) объясняются изменениями совокупности независимых переменных Модель считается адекватной.
Критическая статистика для теста отношения правдоподобия p(Likelihoodratio) - 142.118 Если p(Likelihood-ratio) мала и меньше уровня значимости α, модель является значимой.
