
Результат
Были подобраны следующие праймеры удовлетворяющие требованиям:
For: cccgcgtcctgaattacaatcacca
Rev: ggcagcgccggcttattattgttga
Длинна обоих праймеров равна 25 нуклеотида, содержание (g-c) равно 52%, температуры отжига 77С.
Расчет праймеров и параметров пцр с помощью специальных программ
Имея некоторый опыт в постановке ПЦР в целях экономии времени для подбора праймеров можно использовать коммерческие программные продукты, а также Интернет-ресурсы. Аналогично можно произвести другие расчеты, связанные с ПЦР. В частности сайт MOLBIOL.RU – это профессиональная интернет-территория для русскоязычных молекулярных биологов, генетиков, биохимиков, биофизиков, биоинженеров (http://www.molbiol.ru). Выложенная на сайте специальная форма поможет рассчитать параметры ПЦР, программы для подбора праймеров на сайте нет.
Рассчитать (подобрать) праймеры для ПЦР-амплификации конкретной ДНК можно с помощью других Интернет-ресурсов. В частности, можно использовать программу Primer3 (http://frodo.wi.mit.edu) или другие.
В приведенной ниже работе предлагается ознакомиться с интернет-порталами, где можно произвести расчѐты для ПЦР (англ. PCR – PolymeraseChainReaction), т.е. получить практические навыки в работе c программами [3].
Ход работы
1. Откройте сайт программы Primer3 (http://frodo.wi.mit.edu). Введите в специальное окно нуклеотидную последовательность гена НАДН-дегидрогеназы гадюки обыкновенной (источник – http://www.pubmed.сом).

2. Нажмите «PickPrimers» и программа подберѐт вам несколько пар праймеров. Длина ПЦР-продукта, или ампликона, при этом составит около 200 п.н. Это удобно для выявления какой-либо ДНК, но не подходит для работ, когда нужен полноразмерный ген или конкретный участок гена [5].
ADDITIONAL OLIGOS
startlen tmgc% any 3'seq
1 LEFT PRIMER 497 20 60.20 50.00 8.00 1.00 ggaggcctcaatcaaactca
RIGHT PRIMER 674 20 59.84 45.00 2.00 0.00 ttgttgctgacgatgaaagg
PRODUCT SIZE: 178, PAIR ANY COMPL: 4.00, PAIR 3' COMPL: 1.00
2 LEFT PRIMER 497 20 60.20 50.00 8.00 1.00 ggaggcctcaatcaaactca
RIGHT PRIMER 671 20 59.84 45.00 2.00 0.00 ttgctgacgatgaaaggttg
PRODUCT SIZE: 175, PAIR ANY COMPL: 4.00, PAIR 3' COMPL: 2.00
3 LEFT PRIMER 496 20 60.20 50.00 8.00 0.00 tggaggcctcaatcaaactc
RIGHT PRIMER 671 20 59.84 45.00 2.00 0.00 ttgctgacgatgaaaggttg
PRODUCT SIZE: 176, PAIR ANY COMPL: 4.00, PAIR 3' COMPL: 3.00
4 LEFT PRIMER 310 20 60.24 50.00 8.00 2.00 aatagctgcagcaccattcc
RIGHT PRIMER 495 20 59.87 55.00 4.00 1.00 aggccaccgactaagacaga
PRODUCT SIZE: 186, PAIR ANY COMPL: 3.00, PAIR 3' COMPL: 1.00
3. Проанализируйте подобранные пары праймеров и выберите на ваш взгляд наиболее подходящую пару, предложенную по умолчанию.
Наиболее целесообразно выбрать четвертую пару, т. к. с помощью этой пары прамеров можно получит наиболее длинный ампликон.
4. Задайте длину амплифицируемого участка ДНК, к примеру, 851–1000 п.н. и подберите праймеры. Узнайте, сколько нуклеотидов потеряет ампликон.
ADDITIONAL OLIGOS
startlen tmgc% any 3'seq
1 LEFT PRIMER 31 21 59.70 42.86 6.00 0.00 cattttcatgaccaccacctt
RIGHT PRIMER 917 20 58.98 55.00 4.00 2.00 ggtggggtgggatagttatg
PRODUCT SIZE: 887, PAIR ANY COMPL: 6.00, PAIR 3' COMPL: 3.00
2 LEFT PRIMER 29 20 58.42 45.00 6.00 0.00 agcattttcatgaccaccac
RIGHT PRIMER 916 20 58.98 55.00 4.00 0.00 gtggggtgggatagttatgg
PRODUCT SIZE: 888, PAIR ANY COMPL: 5.00, PAIR 3' COMPL: 3.00
3 LEFT PRIMER 31 20 58.26 45.00 6.00 0.00 cattttcatgaccaccacct
RIGHT PRIMER 916 20 58.98 55.00 4.00 0.00 gtggggtgggatagttatgg
PRODUCT SIZE: 886, PAIR ANY COMPL: 5.00, PAIR 3' COMPL: 3.00
4 LEFT PRIMER 31 21 59.70 42.86 6.00 0.00 cattttcatgaccaccacctt
RIGHT PRIMER 918 19 59.47 57.89 2.00 2.00 gggtggggtgggatagtta
PRODUCT SIZE: 888, PAIR ANY COMPL: 6.00, PAIR 3' COMPL: 1.00
При использовании второй или четвертой пары праймеров (соответствуют самому большому размеру ампликона) ампликон потеряет 130 пар нуклеотидов.
5. Задайте один из праймеров, к примеру, праймерND2 For из работы 6.2 и рассчитайте второй. Для этого вставьте последовательность праймера в специальное окно и нажмите «Pick Left Primer».
OLIGO startlen tmgc% any 3'seq
LEFT_PRIMER 30 20 61.18 50.00 6.00 0.00 gcattttcatgaccaccacc
SEQUENCE SIZE: 1007
INCLUDED REGION SIZE: 1007
6. Проделайте те же операции с праймером ND2 Rev и после этого со своими праймерами, подобранными «вручную» ранее.
OLIGO startlen tmgc% any 3'seq
RIGHT_PRIMER 980 20 51.23 50.00 2.00 0.00 gagtgaggggtaagatagtg
SEQUENCE SIZE: 1007
INCLUDED REGION SIZE: 1007
7. Задайте дополнительные требования к праймерам и проведите расчѐт.
8. Откройте главную страницу сайта MOLBIOL.RU (http://www.molbiol.ru). На главной странице найдите раздел «Расчѐты», выберите пункт «Расчѐт параметров ПЦР».
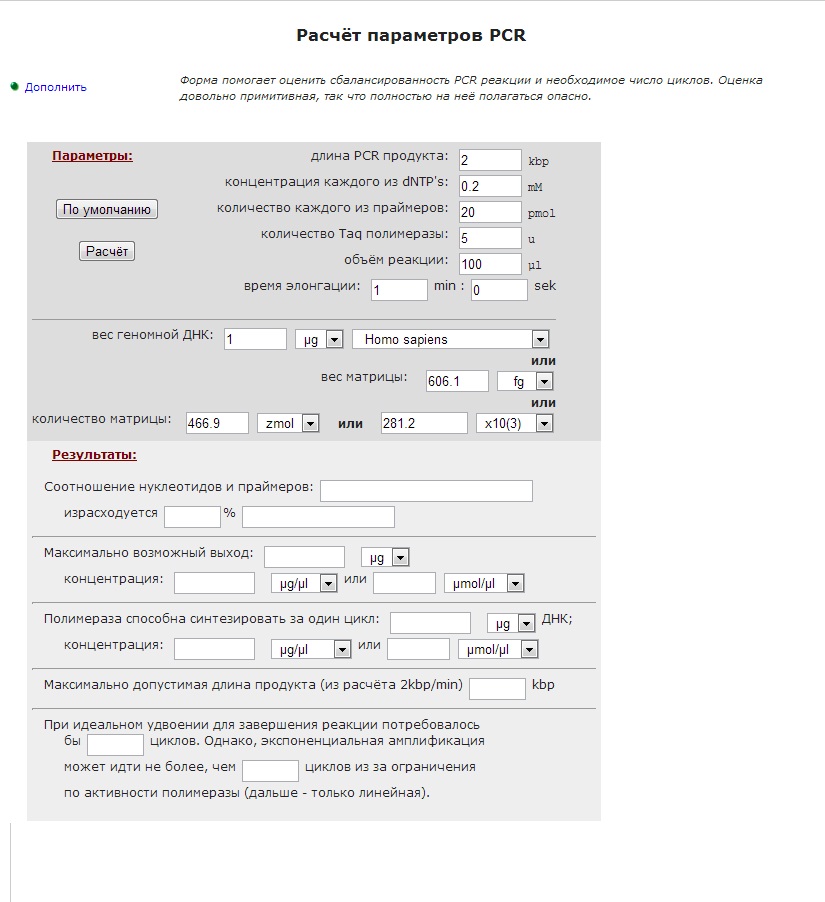
9. Вводя в специальные окна варьирующие параметры, такие как длина ПЦР-продукта, молярное количество праймеров, Taq-полимеразы и другие параметры рассчитайте, сколько нуклеотидов израсходуется в реакции и каков возможный максимальный выход ПЦР-продукта. Определите число циклов достаточное для завершения реакции при «идеальном» удвоении [6].
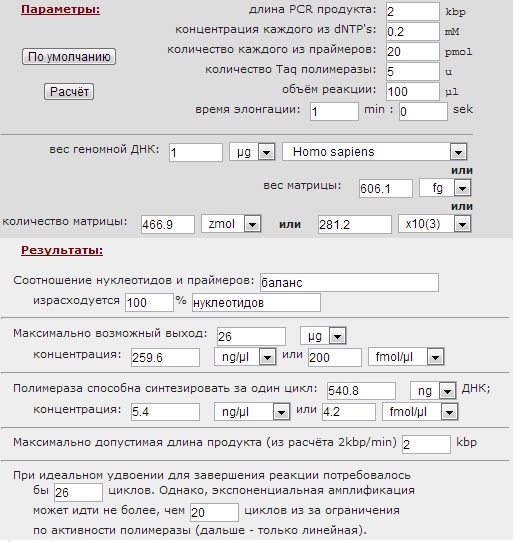
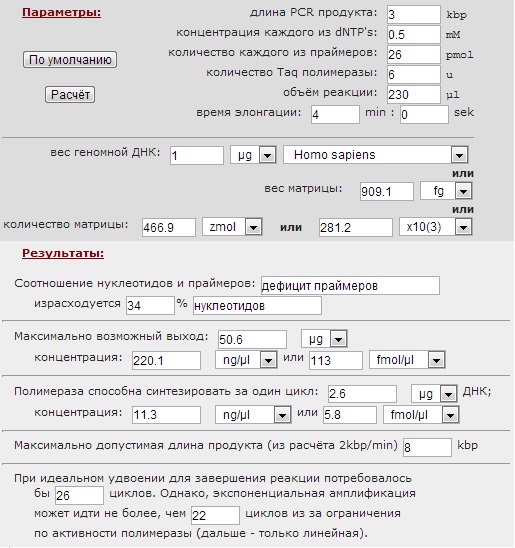

10. Ознакомьтесь с комментариями к расчѐтам. Обратите внимание на то, что расчѐтведѐтся для идеальных условий, когда в качестве матрицы берѐтся 0,5 мкг геномной ДНК человека. Эти данные носят оценочный характер.
Параметры по умолчанию соответствуют амплификации 2kbp фрагмента из 0.5µg человеческой DNA. Предполагается, что:
условия реакции близки к нормальным, так что не приходится беспокоиться о самой возможности проведения PCR (слишком высокая концентрация полимеразы чревата амплификацией грязи, слишком много праймеров - образованием праймер-димеров);
A, T и G, C входят в PCR продукт поровну;
праймеры не образуют праймер-димеров;
Taq полимераза не теряет активность в ходе реакции.
Оценка проводится следующим образом. Обозначим:
длина PCR продукта = L [kbp];
концентрация каждого из dNTP's = с [mM];
количество каждого из праймеров = q [pmol];
количество Taq полимеразы = a [u];
объём реакции = V [µl];
время элонгации = t [min];
количество матрицы = mo;
Тогда:
1.количество синтезированного PCR продукта можно оценить, как минимум из двух: количество при полном расходе нуклеотидов: mn = 4[нуклеотида] x с[mmol/l] 324.5[g/mol] x V[µl] = 1300сV [ng] количество при полном расходе праймеров: mp = q[pmol] x 2[цепи] 324.5[g/mol] x L[kbp] = 650qL [ng]
2.максимальное количество PCR продукта, которое может быть синтезировано за один цикл, зависит от двух факторов:
скорости движения Taq полимеразы по матрице: 2-4[kbp/min];
активности полимеразы (1 u активности - количество фермента, которое включает 10nmol всех четырёх dNTP’s за 30 min при 72oC).
mcycle = 10[nmol] x 324.5[g/mol] x a [u] t[min] / 30[min] = 108at [ng]
3.количество циклов, которое требуется для синтеза "mmax" PCR-продукта: mmax = 2n x mo => n = ln(mmax/mo)/ln2
4.связь молярных и весовых количеств: m[µg] = 649[g/mol] x q[µmol] x L[kbp] x 1000 [6].
Литература
http://ru.wikipedia.org/
http://usca.ulx.ru
Великов В.А. Молекулярная биология. Практическое руководство: Учеб. пособие для студ. биол. фак. и фак. Нано - и биомед. технол., обуч-ся по напр. «Биология (020400)», «Биология-пед (050100)», «Биотехнические системы и технологии (200100)», «Медицинская физика (011200)» и по спец. «Биоинженерия и биоинформатика (020501)». – Саратов: Издательство «Саратовский источник», 2013. – 84 с.: ил.
http://www.pubmed.com
http://frodo.wi.mit.edu
http://www.molbiol.ru/
