
- •58 Лекция 14. Масс-спектрометрия лекция 14. Масс-спектрометрия Что такое масс-спектрометрия?
- •Ионы в электрическом и магнитном полях Масса и заряд
- •Ионы в электромагнитном поле. Закон Лоренца
- •Разрешающая способность масс-спектрометрии
- •Точность измерения массы
- •Методы ионизации
- •От ионов в растворе к ионам в газовой фазе
- •Электронная ионизация
- •Ионизация полем
- •Бомбардировка быстрыми атомами
- •Плазменная десорбция
- •Ионизация лазерной десорбцией при помощи матрицы
- •Химическая ионизация при атмосферном давлении
- •Фотонная ионизация при атмосферном давлении
- •Ионизация лазерной десорбцией с поверхности кремния
- •Ионизация электрораспылением
- •Наноэлектрораспыление
- •Сравнение возможностей различных методов образования ионов
- •Масс-спектрометры Общая архитектура масс-спектрометра
- •Чувствительность
- •Динамический диапазон
- •Вакуумирование
- •Различные типы масс-анализаторов
- •Масс-спектрометры с одиночной и двойной фокусировкой
- •Квадрупольный масс-фильтр
- •Kвадрупольная ионная ловушка
- •Масс-спектрометрия с фурье-преобразованием
- •Времяпролетная масс-спектрометрия
- •Детекторы
- •Тандемная масс-спектрометрия
- •Орбитальная ловушка ионов
- •Масс-спектры биологических макромолекул Представление масс-спектра вещества и его интерпретация
- •Масс-спектры простых соединений
- •Масс-спектры биологических макромолекул
- •Нуклеиновые кислоты
- •Карбогидраты
Нуклеиновые кислоты
Идентификация оснований
Эффективность исследования нуклеиновых кислот с помощью масс-спектрометрии отстает от изучения белков. Основная причина заключается в высоком сродстве нуклеиновых кислот к ионам натрия, которые резко снижают эффективность ионизации. Эти трудности проявляются уже на первых этапах структурных исследований самих оснований ДНК. На рисунке 9.10 показаны масс спектры трех оснований: аденина (C5H5N5, 135 Дa), цитозина (C4H5N3O, 111 Да) и тимина (C5H6N2O2, 126 Да). Масс-спектр, полученный методом прямой ионизация – с удалением электрона из основания ДНК (без нейтрализации налетающей частицы) представлен на рисунке в виде черных профилей, а методом захвата электрона (перенос электрона из основания на налетающую частицу) – в виде серых. Как видно из приведенных рисунков, спектры очень сложны. На них кроме основного иона проявляются еще несколько фрагментарных групп ионов, число и относительный вклад в спектры которых меняется от основания к основанию. Строгая интерпретация спектров еще предстоит.
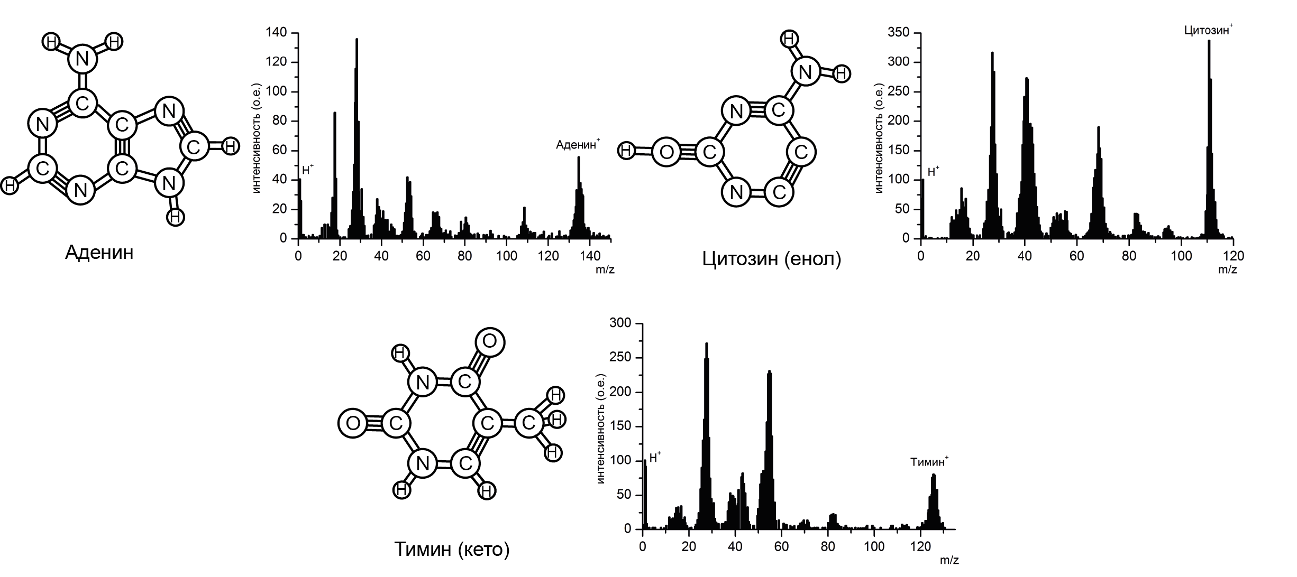
Рис. 9.10. Масс-спектры трех оснований ДНК, полученные методом ТОF-спектрометрии. Цитозин в этих экспериментах существует в виде двух таутомерных форм: енольной (показана) и кето-формы (не показана). Энергия ионизации составляла 80 кэВ. Оба способа ионизации оснований дают сходные масс-спектры. Спектры не нормированы на основной пик. Изображение спектров взято из статьи Tabet et al., 2009
Спектр четвертого основания (гуанин) пока не получен в силу трудностей его перевода в газовую фазу без изомеризации и термической деструкции.
Идентификация олигонуклеотидов
Получение интактных молекулярных ионов олигомеров, содержащих более двух нуклеотидов, становится затруднительным, если применять классические техники ионизации, такие как электронная и химическая ионизация, ввиду высокой полярности нуклеиновых кислот и склонности их ионов к фрагментации. Точность определения массы олигонуклеотидов сильно уменьшается с ростом молекулярной массы из-за фрагментации вследствии потери пуриновых оснований и в несколько меньшей степени из-за потери цитозина.
Еще в 1982 году была продемонстрирована способность FAB техники получать интактные ионы нуклеотидов, на основе которой вскоре было предложено ультраскоростное секвенирование олигонуклеотидов. Внедрение ESI и MALDI значительно расширило диапазон масс при анализе нуклеиновых кислот. Затем последовал заметный прогресс в секвенировании олигонуклеотидов, анализе смесей, исследовании нековалентных комплексов и манипуляции с микрообразцами. Попытки анализа больших нуклеиновых кислот с помощью MALDI-МS с лазерным излучением в ультрафиолетовой области имели весьма умеренный успех. Верхний предел измеряемых масс находился в районе 90 кДа для ДНК и 150 кДа для РНК и был ограничен в основном фрагментацией ионов. Оказалось, что использование инфракрасного лазера и глицериновой матрицы представляет собой наиболее мягкую комбинацию для интактной десорбции и ионизации нуклеиновых кислот в большом диапазоне от небольших олигонуклеотидов до макромолекул длиной в 2000 нуклеотидов. Так, для синтетической ДНК длиной в 21 нуклеотид (молекулярная масса 6398 Да) разрешение составило 1200, что сходно с разрешением для белков, имеющих сравнимую массу.
Упомянем также, что основным требованием для анализа ДНК независимо от способа ионизации является высокая чистота исследуемых молекул. Такие вещества как SDS (английская абревиатура, DTT, EDTA, хлориды металлов, с которыми мы привыкли работать в растворе, должны быть обязательно удалены.
На первый взгляд кажется, что комбинация жидкостной хроматографии и техники ионизации электрораспылением в масс-спектрометрии должна быть очень информативной в анализе нуклеотидных смесей. Однако дальнейшее развитие показало ограниченность такого подхода ввиду несовместимых требований двух методов. Анионно-обменная и обратно-фазная хроматография используют солесодержащую подвижную фазу для эффективного разделения, которая ухудшает качество масс-спектра. Решение последней проблемы было найдено добавлением специальной добавки (гексафлуро-2-пропанол) в подвижную фазу. Потенциал нового метода был продемонстрирован на синтетическом гомополимере тимидина длиной в 75 оснований, для фрагментов плазмидной ДНК pBR322 и фосфотиоэфирах антисмысловых олигонуклеотидов. Оказалось, что такая методика наиболее подходит для характеристики ДНК зондов, ПЦР праймеров, причем для тестирования достаточно менее 10 пМ материала.
Идентификация больших молекул ДНК
Может показаться, что для интактной десорбции больших нуклеиновых кислот ESI более подходит, чем MALDI. Но точность отнесения отдельных пиков к определенным значениям массы в этом случае очень невелика и неопределенность достигает около 10%. Самые большие нуклеиновые кислоты, детектированные с помощью ESI-МS, имели массу около 40 кДа.
Для детектирования очень больших молекул ДНК наиболее подходящим является метод ICR-FT, когда за один проход можно провести более 450 повторных измерений одного и того же иона. Это позволило значительно повысить отношение сигнал – шум и точность измерения массы и заряда очень больших ионов. На рисунке 9.11 a приведен масс-спектр ионов ДНК с молекулярной массой 2.88 МДа, несущих более 250 зарядов, полученный с низким разрешением – около 25. Комбинирование ESI и ICR-FT в масс-спектрометрии дает возможность анализа отдельных макроионов, несущих множественный заряд. Рисунок 9.11 б показывает ICR-FT-спектр, полученный для ДНК бактериофага T4 (ожидаемая молекулярная масса примерно 111.5 МДа). Отметим, что сигнал индивидуальных ионов этой ДНК регистрировался под давлением около 1.010-9 Торр через 1.5 часа после первоначального возбуждения.
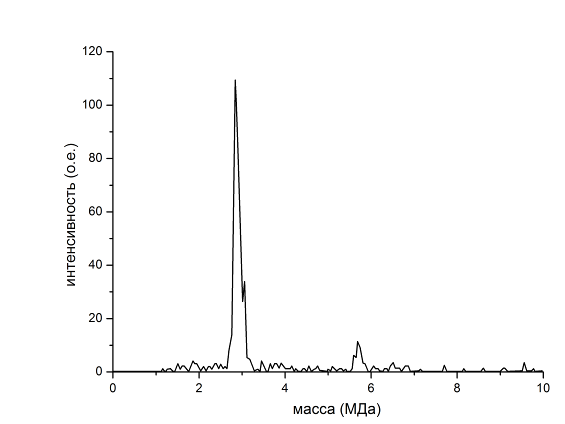
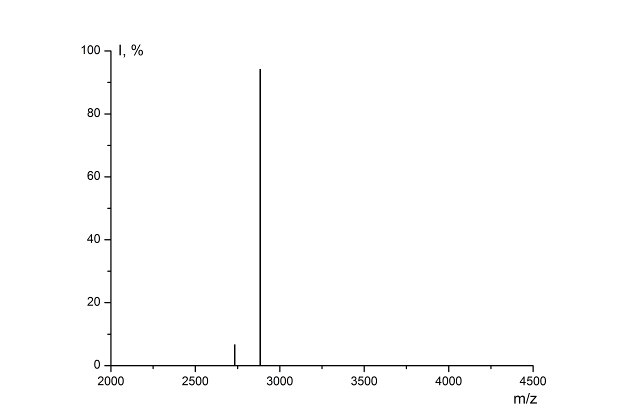
Рис. 9.11 a) Гистограмма средней массы, полученная для образца ДНК. Основной пик при 2.88 МДа соответствует ионам натриевой соли бактериальной плазмиды pBR322, а гораздо меньший пик при 5.85 МДа – ионам димера pBR322 б) ICR-FT спектр иона с m/z =2883, полученный для ДНК T4 бактериофага. Расчетная молекулярная масса равна (90.9 ± 9.1)×106 Да
Расшифровка нуклеотидной последовательности
Как и в случае с белками, существуют правила расшифровки нуклеотидной последовательности. Напомним, что такая расшифровка возможна, только в случае если разрешающая способность использованного масс-анализатора достаточна, чтобы уверенно определять различие в массе нуклеотидов. В таблице 9.2 приведены мононуклеотидные массы остатков.
Таблица 9.2. Мононуклеотидные массы остатков
Обозначение |
Элементный состав |
Масса (Дa) |
Adenosine (A) |
C10H12N5O6P |
329 |
Guanosine (G) |
C10H12N5O7P |
324 |
Cytosine (C) |
C9H12N3O7P |
305 |
Uracil (U) |
C9H11N2O3P |
306 |
Deoxyadenosine (dA) |
C10H13N2O7P |
313 |
Deoxyguanosine (dG) |
C10H12N3O5P |
329 |
Deoxycytidine (dC) |
C9H12N3O6P |
289 |
Thymidine (T) |
C10H13N2O7P |
304 |
На рисунке 9.12 схематически представлена структура олигодезоксирибонуклеотида d(A–C–T–C–G–A–T–G), на которой отмечены связи, расщепление которых приводит к образованию основных ионных фрагментов.
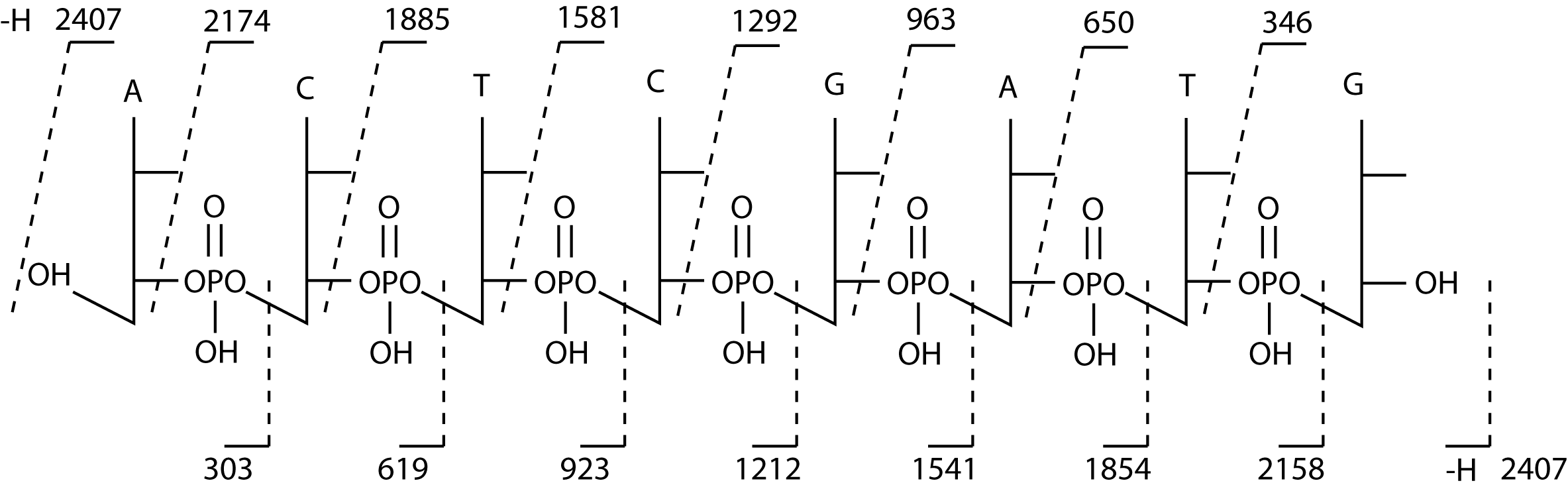
Рис 9.12 Схематическая структура октамерного олигодезоксирибонуклеотида d(A–C–T–C–G–A–T–G), с отмеченными точками разрыва связей, в результате которых образуются основные ионные фрагменты. Пунктиром отмечены основные фрагменты с указанием массы: сверху – фрагменты, соответствующие ионам с 5’-фосфатными концами (5’ – P), снизу – ионы с 3’-фосфатными концами (3’ – P)
Приведем пример определения последовательности олигодезоксирибонуклеотида из следующей серии данных:
m/z (5' – P): 2407, 2174, 1885, 1581, 1292, 963, 650, 346
m/z (3' – P): 330, 619, 923, 1212, 1541, 1854, 2158, 2407
Расчет последовательности идет следующим образом.
Разность масс (Δ) в значениях m/z между последующими ионами представляет собой массу нуклеотидного остатка. Следовательно, набор m/z данных может быть преобразован в олигонуклеотидную последовательность следующим образом:
(5' – P) Δ 233* 289 304 289 329 313 304
m/z 2407 2174 1885 1581 1292 963 650 346*
нуклеотиды A C T C G A T G
Молекулярная масса первого нуклеотида М = 233 + 96(PO4H) – 17(OH) + 1(H) = 313 Да
Следовательно, это А.
Молекулярная масса последнего нуклеотида М = 346 – 17(OH) = 329 Да
Следовательно, это G.
Получаем последовательность
d(A–C–T–C–G–A–T–G)
Можно проверить последовательность нуклеотидов, применяя те же правила для ряда значений m/z для (3' – P) конца.
Наибольший пик в FAB-спектре m/z = 2407, который представляет (M – H)–. Отсюда M = 2408 + 96(PO4H) – 34(2OH) = 2470 Да.
Сумма нуклеотидов дает 2470 Да, что соответствует масс-спектрометрическому значению.
РНК
Точность определения массы методом MALDI-MS-TOF вполне достаточна для определения положения аденозина (329.2 Дa), гуанозина (345.2 Дa) и пиримидиновых нуклеотидов в спектрах РНК длиной 55 нуклеотидных пар, полученных расщеплением экзонуклеазой. Однако, массовые различия в уридине (306.2 Дa) и цитидине (305.2 Дa) невелики и не доступны TOF-методу при молекулярных массах 55 н.п. и более. Однако для молекулярных масс меньших 30 н.п. вполне можно отличить цитидин от уридина. С использованием ПЦР-реакции это открывает путь для секвенирования ДНК с помощью MS-TOF. Верхний предел применения MALDI-MS-TOF для РНК составляет около 461 н.п., что позволяет применить этот подход и для анализа ДНК.
