
- •Лабораторная работа № 1. Построение математических моделей Методические указания
- •1. Построение математической модели технологического аппарата экспериментальным методом
- •Метод наименьших квадратов
- •Проверка адекватности модели
- •Исходные данные
- •Составление дифференциальных уравнений
- •Математическое моделирование процессов периодического культивирования микроорганизмов
- •Математическое моделирование процессов непрерывного культивирования микроорганизмов
- •Основные принципы работы в RasMol
- •Меню графического окна
- •Команды
- •Операции с цветом
- •Другие операторы и команды
- •Лабораторная работа №2
- •Лабораторная работа №3
- •Лабораторная работа №4
- •Лабораторная работа №5
- •Лабораторная работа №6
- •Вопросы для контроля знаний
Основные принципы работы в RasMol
Работа идет в двух окнах – графическом и командном. Графическое окно визуализирует молекулу, отображая все изменения, происходящие в результате выполнения команд текстового меню или прописанных в командном окне.
В каждый момент работы имеется некоторое выделенное множество атомов. Все действия производятся с этим множеством. Для того, чтобы выполнить действие над всей молекулой, нужно сначала выделить все атомы.
Каждому действию соответствует команда, набираемая в командном окне. Так же в нём прописывается действие, произведённое над изображением. Например, при нажатии на атом в графическом окне, в командном мы видим:
- название атома,
- номер его положения,
- название аминокислоты, в которую он входит, либо молекула воды,
- цепь, в которую он входит.
После запуска программы открываются два окна. Их внешний вид показан на рисунках 4.1 и 4.2.
Рисунок 4.1 – Графическое окно
Рисунок 4.2 – Окно командной строки
Меню графического окна
Основное окно содержит главное (текстовое) меню, представляющее собой линейку раскрывающихся меню. Меню содержит следующие основные команды:
File (Файл),
Edit (Правка),
Display (Отображение),
Colours (Окрашивание),
Options (Операции),
Export (Экспорт в формат),
Help (Справка).
Команда меню Fail включает в себя подменю:
Open – открывает выбранный файл. Исходными данными для визуализации в RasMol служат координаты атомов молекулы или комплекса молекул, содержащиеся в файле формата Protein Data Bank (PDB). Файлы могут быть скопированы с одного из сайтов PDB.
Information – содержит информацию о структуре открытого файла: название молекулы, классификация, число цепей, групп, атомов, связей.
Print – для печати изображения.
Exit – выход из программы.
Команда меню Edit включает в себя подменю:
Select all – позволяет выбрать все атомы и проводить операции с ними.
Cut – вырезать.
Copy – копировать.
Paste – вставить.
Delete – удалить.
Команда меню Display показывает различные способы визуализации молекул. Включает в себя подменю:
Wireframe (проволочная модель) – представляет каждую связь в виде линии, цилиндра или вектора.
Backbone (остовная модель) – позволяет представить полипептидную основу в виде серии связей смежных α-углеродов каждой аминокислоты в цепочке.
Spacefill (шариковая модель) - используется для обозначения всех выбранных атомов в виде твёрдых сфер.
Ball & Stick (наложение проволочной и шариковой модели) – позволяет представить атомы и связи в виде шариков и цилиндров.
Ribbons (ленточная модель) – позволяет представить загруженный белок или нуклеиновую кислоту, как гладкую твёрдую "ленту", проходящую вдоль аминокислотного скелета.
Strands – отображает загруженные белки или нуклеиновые кислоты как гладкие ленты, состоящие из кривых, параллельных друг другу и проходящих вдоль плоскости пептидных связей.
Cartoons – отображает молекулы как ленты с учётом вторичной структуры, при этом визуализируются альфа-спирали в виде спиралей и бета-складки в виде стрелок (С-концевой участок выводится в виде стрелки).
Команда меню Colours показывает различные способы окрашивания молекул. Включает в себя подменю:
Monochrome – окрашивает молекулу в белый цвет.
CPK – цветовая схема, разработанная Кореем и Полингом, затем улучшенная Колтуном. Каждый элемент окрашивается своим цветом. Схема представлена на рисунке 4.3.
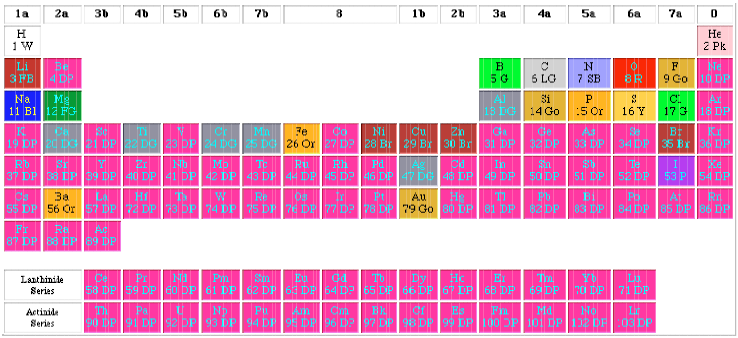
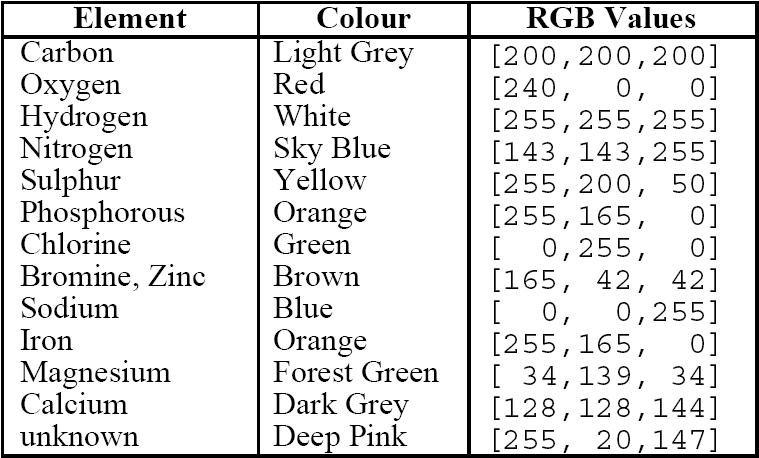
Рисунок 4.3 – Схема окрашивания элементов
Shapely – схема окрашивания аминокислот в соответствии со свойствами каждой аминокислоты с целью идентифицировать аминокислоту. Причём гидрофильные участки белка имеют цвет интенсивнее, чем гидрофобные. Большинство цветов традиционны, схема представлена на рисунке 4.4.

Рисунок 4.4 – Схема окрашивания аминокислот
Group – при окрашивании по этой схеме каждая полипептидная цепь представляет собой спектр от синего на 3’ конце до красного на 5’ конце.
Chains – эта цветовая схема каждой цепи макромолекулы присваивает свой цвет. Особенно полезна для выделения части структуры нити в цепи ДНК.
Temperature – окрашивает каждый атом в соответствии с анизотропной температурой, которая хранится в pdb-файле. Синий цвет соответствует самой низкой температуре, красный – самой высокой.
Structure – окрашивает загруженные белки по вторичной структуре, при этом альфа-спирали окрашиваются всегда в розовый, а бета-складки - в жёлтый.
User – данная команда позволяет использовать цветовую схему, хранящуюся в pdb-файле.
Команда меню Options включает в себя подменю:
Slab Mode – загруженный белок разрезается по z-плоскости, причём визуализируется только часть молекулы, находящаяся за плоскостью разреза (то есть дальше от наблюдателя).
Hydrogen – параметр используется для изменения поведения RasMol «по умолчанию». Когда параметр выключен, по умолчанию выбирается регион, не включающий атомы водорода, дейтерия и трития. При включенном значении регион может содержать атомы водорода.
Hetero Atoms – параметр используется для изменения поведения RasMol «по умолчанию». Когда параметр выключен, по умолчанию выбирается регион, не включающий неоднородные атомы. При включенном значении регион может содержать неоднородные атомы. Команда похожа на предыдущую.
Specular – команда включает или отключает отображение бликов на твердых предметах загруженного изображения.
Shadows – позволяет отображать тени на объектах.
Stereo – визуализируются две молекулы рядом.
Labels – команда позволяет вывести в графическом окне строку текста, связанную с выделенным атомом. Здесь указывается аминокислота, в составе которой находится атом, а так же номер, обозначающий его положение и цепь.
Команда меню Export позволяет сохранить изображение в нужном формате. Поддерживаемые форматы для сохранения: bmp, gif, ppm, epsf, rast.
